您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章跟大家分析一下“怎樣進行motif分析”。內容詳細易懂,對“怎樣進行motif分析”感興趣的朋友可以跟著小編的思路慢慢深入來閱讀一下,希望閱讀后能夠對大家有所幫助。下面跟著小編一起深入學習“怎樣進行motif分析”的知識吧。
在chip_seq數據分析中,motif分析是一項重要的分析內容。通過motif分析,我們可以對轉錄因子結合位點的序列模式有進一步的了解,那么什么是motif呢?
蛋白質中功能的基本單元是domain,是一種特殊的三維結構,不同結構的domain與其他分子特異性結合從而發揮功能。與此類似,轉錄因子在于DNA序列結合時,其結合位點的序列也由于一定的特異性,不同轉錄因子結合的DNA序列的模式是不同的。為了更好的描述結合位點序列的模式,科學家們提出了motif的概念。
motif表示特定堿基序列的模式,這種教科書式的名詞解釋不夠直觀難以理解,下面通過一個示例來看下
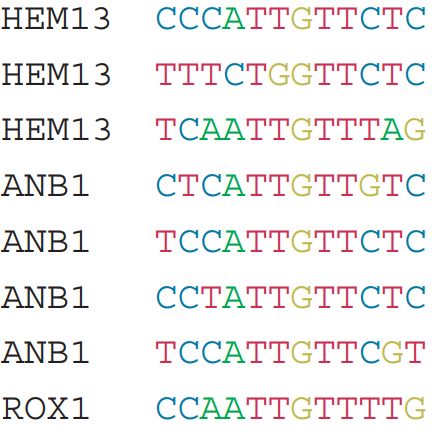
上圖表示的是一個轉錄因子在多個基因上的結合位點的序列,在采用motif描述上面這段信息時,具體用到以下幾個元素
上述示例中結合位點的序列長度為12bp, 每個位置4種堿基的頻數統計如下

用一段序列來描述所有序列的堿基組成,稱之為一致性序列,采用IUPAC標準的堿基表示法,不同字母對應的堿基如下所示

上述例子中的一致性序列如下

為了更加直觀的描述motif, 結合所有序列中的堿基分布情況和一致性序列的特征,提出了sequence logo的表示方法,
上述例子中的堿基分布頻數繪圖如下

類似每個位置上堿基分布頻數的堆積柱狀圖,而sequence logo則采用以下公式來計算位置堿基的高度
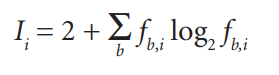
上述公式中的最大值為2,對應所有序列中該位置都是同一個堿基,比如示例中的第5個位置全部都是T堿基。通過這種表示方法,可以突出堿基的分布情況,示意如下

可以非常明顯的看到,在第5,8,9三個位置上都只出現了T堿基。其他位置都是幾種堿基混合出現,總的高度都比這里這些位置低很多。值得一提的是,之前我們說單一堿基最大值為2,而上圖中單一堿基的位置比2小了一點,這是因為在原始公式的基礎上進行了微調,詳細情況如下
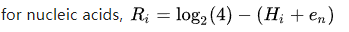
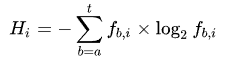
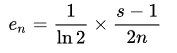
在原始公式的基礎上減去了一個e值,在e值的計算公式中,對于DNA序列,s的值為4,n代表的是motif的長度,上述示例中就是12。減去e值后,最大值相比2自然會小一點。
有很多的軟件可以進行motif分析,比如meme-chip, homer等。
關于怎樣進行motif分析就分享到這里啦,希望上述內容能夠讓大家有所提升。如果想要學習更多知識,請大家多多留意小編的更新。謝謝大家關注一下億速云網站!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。