您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用seqLogo可視化motif”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用seqLogo可視化motif”吧!
seqLogo是一個bioconductor上的R包,專門用于DNA序列的motif可視化,網址如下
https://www.bioconductor.org/packages/release/bioc/html/seqLogo.html
因為功能的單一性,所以其用法也特別的簡單,只需要輸入motif對應的PPM矩陣就可以了,下面通過一個實際例子來看下

上圖為一個motif的PFM矩陣,只需要通過以下幾個步驟就可以得到對應的sequence logo。
將PFM矩陣保存在一個文件pfm.txt中,內容如下
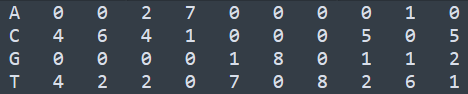
注意PFM矩陣中規定堿基順序為ACGT, 不能任意調換堿基的順序。通過R語言來讀取,代碼如下
data <- read.table("pfm.txt", header = F, sep = "\t", row.names = 1)PPM矩陣就是將PFM矩陣中的頻數轉化成頻率,除以每列的總和就可以了, 代碼如下
ppm <- sapply(1:ncol(data), function(t){ data[[t]] / sum(data[[t]]) })ppm的內容如下
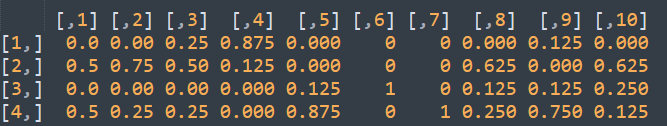
因為行的順序統一為ACGT, 所以我們不需要設置行名稱。
基本用法如下
p <- makePWM(ppm)
seqLogo(p)輸出結果示意如下
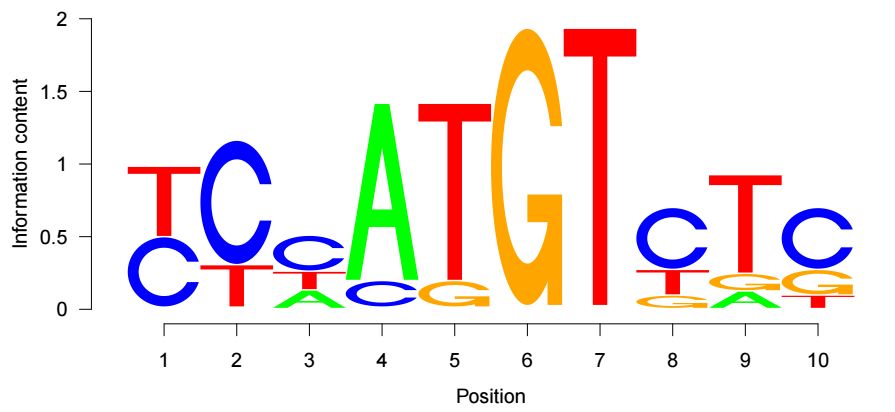
相比其他工具,這個R包的功能較為單一,存在無法調整配色方案等問題,但是作為一個可視化工具,其足夠簡單,已經能滿足基本要求。
感謝各位的閱讀,以上就是“如何使用seqLogo可視化motif”的內容了,經過本文的學習后,相信大家對如何使用seqLogo可視化motif這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。