您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用ggseqlogo可視化motif”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用ggseqlogo可視化motif”吧!
ggseqlogo是一個motif可視化的R包,可以看做是seqLogo的加強版。除了基本的創建sequence logo的功能,新增了許多自定義的選項,靈活性更強,項目網址如下
https://omarwagih.github.io/ggseqlogo/
接受兩種格式的motif信息,第一種為序列數據,以下圖為例
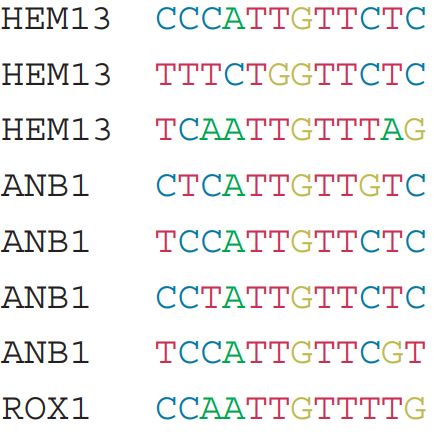
在R中的表示方式如下
motif_seq <- c(
"CCCATTGTTCTC",
"TTTCTGGTTCTC",
"TCAATTGTTTAG",
"CTCATTGTTGTC",
"TCCATTGTTCTC",
"CCTATTGTTCTC",
"TCCATTGTTCGT",
"CCAATTGTTTTG"
)第二種為motif的PFM矩陣,以下圖為例

在R中的表示方式如下
motif_pfm <- rbind(
c(0, 0, 2, 7, 0, 0, 0, 0, 0, 0, 1, 0),
c(4, 6, 4, 1, 0, 0, 0, 0, 0, 5, 0, 5),
c(0, 0, 0, 0, 0, 1, 8, 0, 0, 1, 1, 2),
c(4, 2, 2, 0, 8, 7, 0, 8, 8, 2, 6, 1)
)
rownames(motif_pfm) <- c("A", "C", "G", "T")準備好任意一種符合格式的輸入文件之后,就可以進行可視化了。對于序列格式的信息,繪圖方式如下
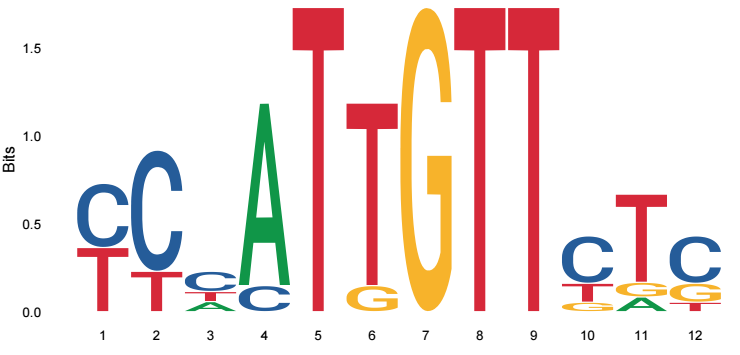
ggseqlogo(motif_seq)
輸出結果如下
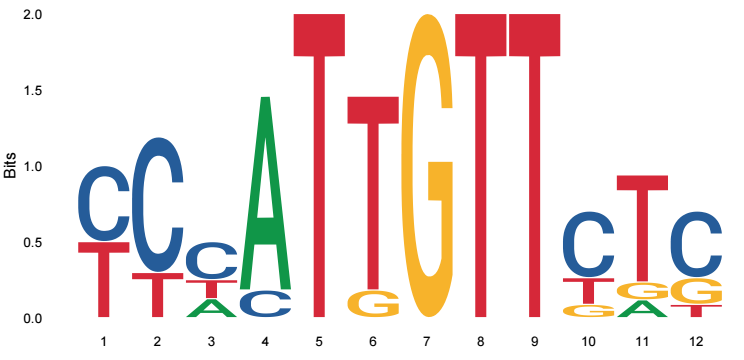
對于PFM矩陣,繪圖方式如下
ggseqlogo(motif_pfm)
輸出結果如下
以上只是ggseqlogo的基本用法,除此之外,還有以下幾種額外功能
和webLogo類似,針對核酸和蛋白序列,內置了多種配色方案。對于DNA和RNA序列,支持以下兩種配色防范
nucleotide
base_pairing
對于蛋白質序列,支持以下幾種配色方案
chemistry
hydrophobicity
clustalx
taylor
默認情況下會自動根據輸入的序列類型自動匹配配色方案,可以通過col_scheme強制指定,用法如下
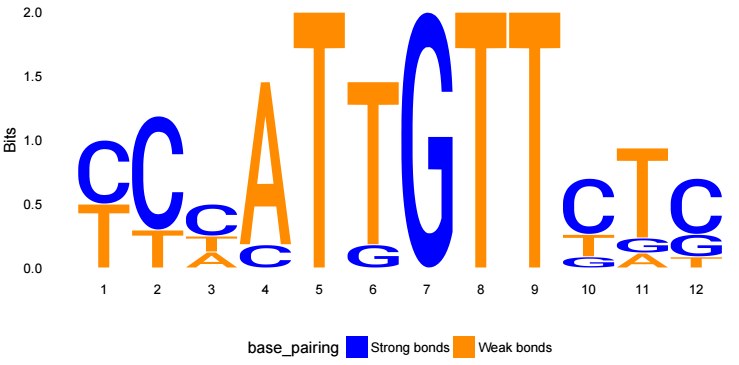
ggseqlogo(motif_pfm, col_scheme='base_pairing')
輸出結果示意如下
還支持自定義配色方案,具體細節請參考官方文檔。
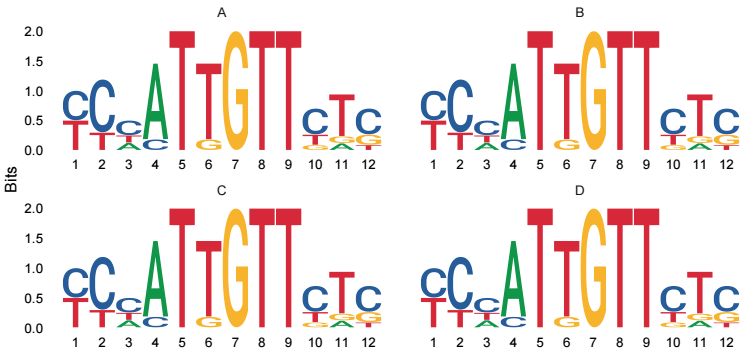
當有多個motif信息時,支持一鍵繪制多個motif的sequence logo, 用法如下
motif_list <- list(
"A"=motif_pfm,
"B"=motif_pfm,
"C"=motif_pfm,
"D"=motif_pfm)
ggseqlogo(motif_list,ncol = 2)將多個motif信息存儲在list中,直接操作這個list對象即可,輸出結果如下
除了這些功能外,還提供了自定義字符集,字符高度,字體等各種功能,更多用法與細節請參閱幫助文檔。
感謝各位的閱讀,以上就是“如何使用ggseqlogo可視化motif”的內容了,經過本文的學習后,相信大家對如何使用ggseqlogo可視化motif這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。