您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關如何使用clusterProfiler包利用eggnog-mapper軟件注釋結果做GO和KEGG富集分析,小編覺得挺實用的,因此分享給大家做個參考,希望大家閱讀完這篇文章后可以有所收獲。
conda activate emapper
python emapper.py -i orgdb_example/GCF_000002945.1_ASM294v2_protein.faa --output orgdb_example/out -m diamond --cpu 8
將注釋結果下載到本地,手動刪除前三行帶井號的行,第四行開頭的井號去掉,文件末尾帶井號的行去掉。
library(stringr)
library(dplyr)
egg<-read.table("out.emapper.annotations",sep="\t",header=T)
egg[egg==""]<-NA
gterms <- egg %>%
select(query_name, GOs) %>%
na.omit()
gene2go <- data.frame(term = character(),
gene = character())
for (row in 1:nrow(gterms)) {
gene_terms <- str_split(gterms[row,"GOs"], ",", simplify = FALSE)[[1]]
gene_id <- gterms[row, "query_name"][[1]]
tmp <- data_frame(gene = rep(gene_id, length(gene_terms)),
term = gene_terms)
gene2go <- rbind(gene2go, tmp)
}
head(gene2go)
> head(gene2go)
# A tibble: 6 x 2
gene term
<chr> <chr>
1 NP_001018179.1 GO:0003674
2 NP_001018179.1 GO:0003824
3 NP_001018179.1 GO:0004418
4 NP_001018179.1 GO:0005575
5 NP_001018179.1 GO:0005622
6 NP_001018179.1 GO:0005623
獲得一個兩列的數據框,有了這個數據框就可以做GO富集分析了
在 https://www.jianshu.com/p/9c9e97167377 這篇文章里的評論區有人提到上面用到的for循環代碼效率比較低,他提供的代碼是
gene_ids <- egg$query_name
eggnog_lines_with_go <- egg$GOs!= ""
eggnog_lines_with_go
eggnog_annoations_go <- str_split(egg[eggnog_lines_with_go,]$GOs, ",")
gene_to_go <- data.frame(gene = rep(gene_ids[eggnog_lines_with_go],
times = sapply(eggnog_annoations_go, length)),
term = unlist(eggnog_annoations_go))
head(gene_to_go)
> head(gene_to_go)
gene term
1 NP_001018179.1 GO:0003674
2 NP_001018179.1 GO:0003824
3 NP_001018179.1 GO:0004418
4 NP_001018179.1 GO:0005575
5 NP_001018179.1 GO:0005622
6 NP_001018179.1 GO:0005623
用這個代碼替換for循環,確實快好多。
首先準備一個基因列表,我這里選取gene2go中的前40個基因作為測試 還需要為TERM2GENE=參數準備一個數據框,第一列是term,第二列是基因ID,只需要把gene2go的列調換順序就可以了。
library(clusterProfiler)
gene_list<-gene2go$gene[1:40]
term2gene<-gene2go[,c(2,1)]
df<-enricher(gene=gene_list,
pvalueCutoff = 0.05,
pAdjustMethod = "BH",
TERM2GENE = term2gene)
head(df)
barplot(df)
dotplot(df)
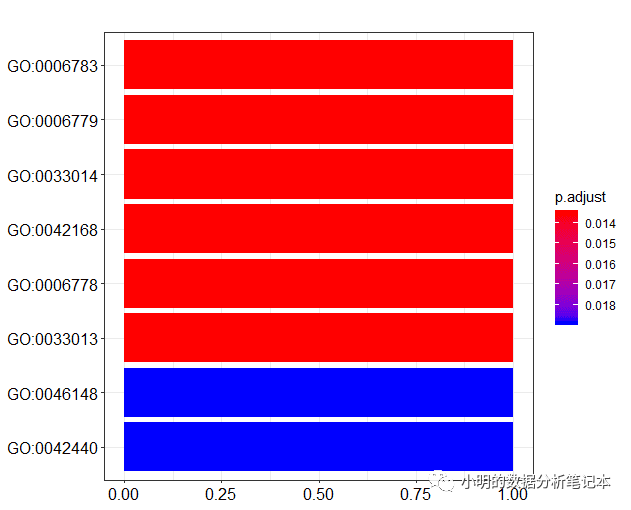
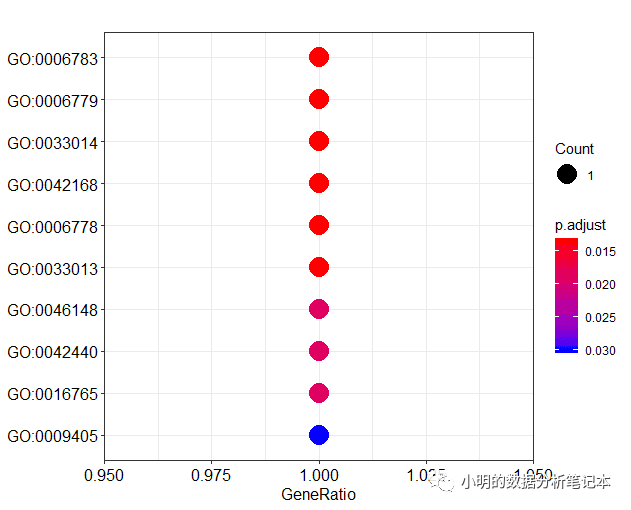
y軸的標簽通常是GO term (就是文字的那個)而不是GO id。clusterProfiler包同樣提供了函數對ID和term互相轉換。go2term()go2ont()
df<-as.data.frame(df)
head(df)
dim(df)
df1<-go2term(df$ID)
dim(df1)
head(df1)
df$term<-df1$Term
df2<-go2ont(df$ID)
dim(df2)
head(df2)
df$Ont<-df2$Ontology
head(df)
df3<-df%>%
select(c("term","Ont","pvalue"))
head(df3)
library(ggplot2)
ggplot(df3,aes(x=term,y=-log10(pvalue)))+
geom_col(aes(fill=Ont))+
coord_flip()+labs(x="")+
theme_bw()
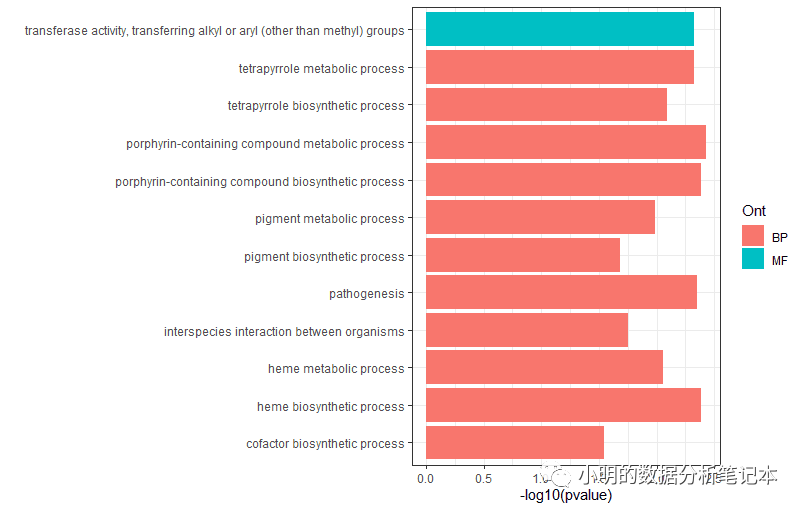
這里遇到一個問題:數據框如何分組排序?目前想到一個比較麻煩的辦法是將每組數據弄成一個單獨的數據框,排好序后再合并。
library(stringr)
library(dplyr)
library(clusterProfiler)
egg<-read.table("out.emapper.annotations",sep="\t",header=T)
egg[egg==""]<-NA
gene2ko <- egg %>%
dplyr::select(GID = query_name, Ko = KEGG_ko) %>%
na.omit()
head(gene2ko)
head(gene2go)
gene2ko[,2]<-gsub("ko:","",gene2ko[,2])
head(gene2ko)
#kegg_info.RData這個文件里有pathway2name這個對象
load(file = "kegg_info.RData")
pathway2gene <- gene2ko %>% left_join(ko2pathway, by = "Ko") %>%
dplyr::select(pathway=Pathway,gene=GID) %>%
na.omit()
head(pathway2gene)
pathway2name
df<-enricher(gene=gene_list,
pvalueCutoff = 0.05,
pAdjustMethod = "BH",
TERM2GENE = pathway2gene,
TERM2NAME = pathway2name)
dotplot(df)
barplot(df)
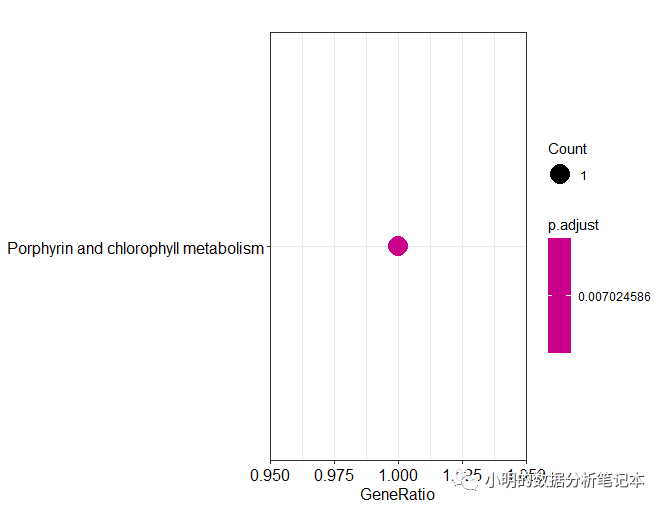
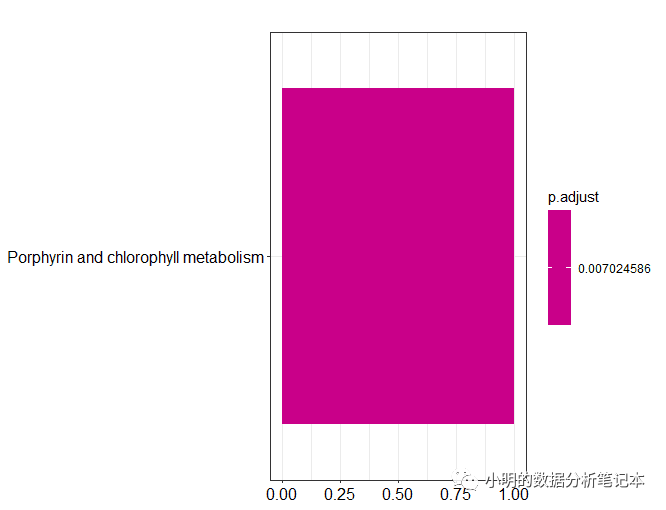
以上最開始的輸入文件是eggnog-mapper軟件本地版注釋結果,如果用在線版獲得的注釋結果,下載的結果好像沒有表頭,需要自己對應好要選擇的列。
關于“如何使用clusterProfiler包利用eggnog-mapper軟件注釋結果做GO和KEGG富集分析”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,使各位可以學到更多知識,如果覺得文章不錯,請把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。