您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“如何使用CNVkit進行CNV分析”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“如何使用CNVkit進行CNV分析”吧!
CNVkit是一款CNV預測軟件,適用于全外顯子,目的區域靶向測序等數據的CNV檢測,官網如下
https://cnvkit.readthedocs.io/en/stable/
被很多高分文章引用, 示意如下

文章發表在PLos上,鏈接如下
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1004873
和Excavator類似,CNVKit也將基因組劃分為in-target和off-target兩部分,流程圖示意如下
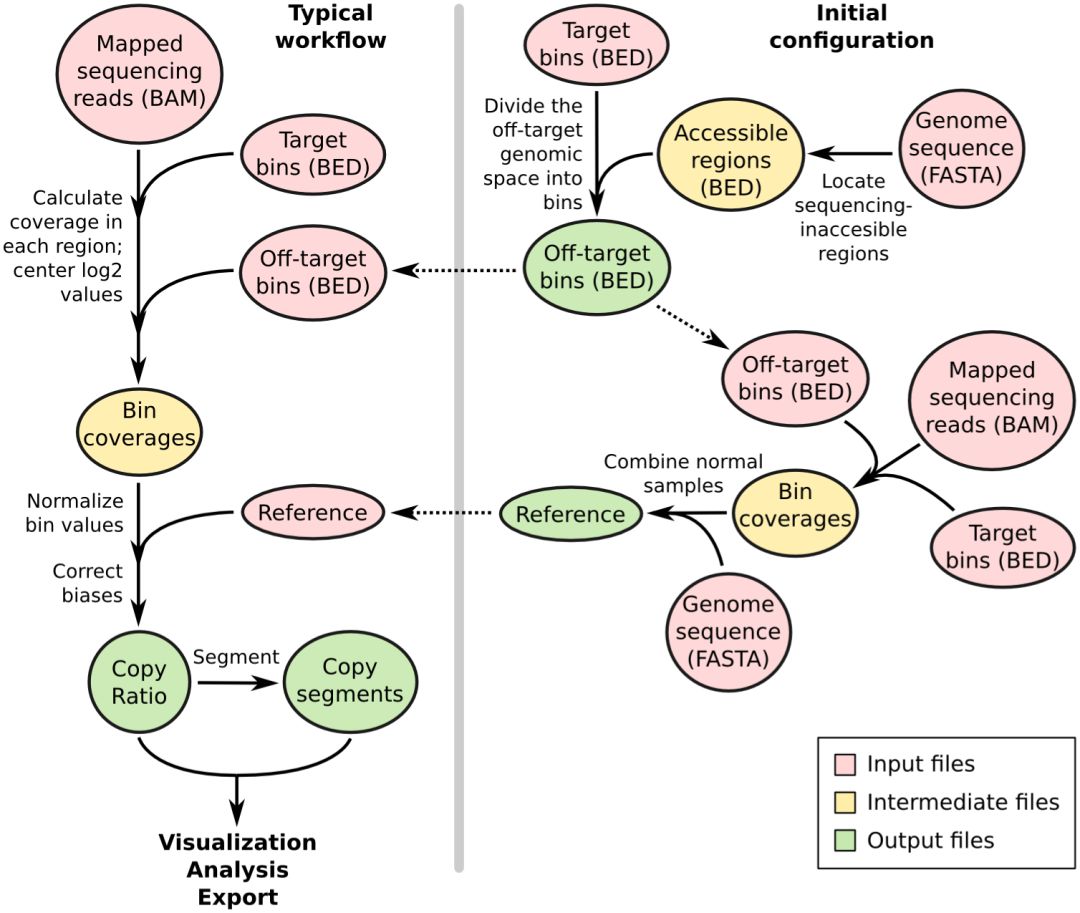
將in-target和off-target區域劃分成小的bin區間,統計bin區間內的測序深度,綜合考慮GC含量,目的區域的大小和分布密度, 重復元件等因素,對原始測序深度進行校正,然后計算相對對照樣本的log2 ratio, 通過segmentation算法來劃分segment, 支持cbs, haar, flasso多種segmentation算法。
CNVkit采用python進行開發,使用方便,集成了可視化功能,可以直觀的展示分析結果,支持導出多種格式的結果文件,可以很好的與下游軟件相結合。采用了模塊化的開發思想,按照功能拆分成了獨立的模塊,示意如下
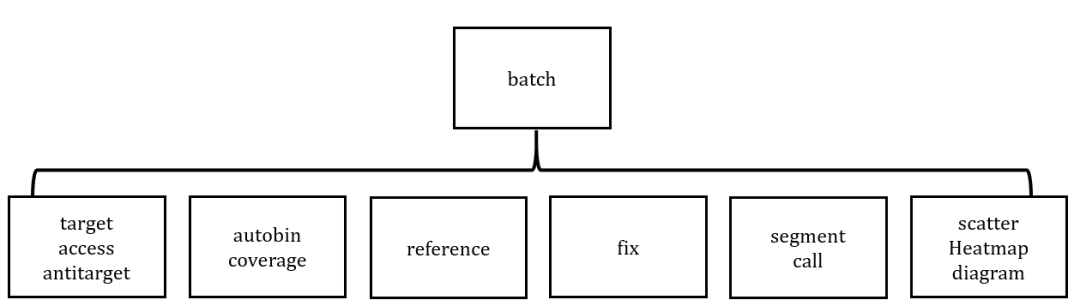
每個功能模塊對應一個子命令,為了方便調用,將pipeline的完整功能封裝在batch子命令中,只通過這一個命令來運行整個pipeline。整個數據分析的過程可以分為以下幾個部分
對于靶向測序而言,肯定需要輸入目的區域的文件,格式為bed, target子命令用于處理目的區域的bed文件,可以添加對應的基因注釋等信息,用法如下
cnvkit.py target \
my_baits.bed \
--annotate refFlat.txt \
-o my_targets.bed除了目的區域in-target,還需要計算off-set區域,也稱之為antitarget, in-target和off-target區域加起來就是基因組上所有可覆蓋的區域。二測測序并不能達到100%的覆蓋度,基因組上的高重復區域,端粒,著絲粒等區域就無法覆蓋,所以cnvkit通過access子命令來計算基因組上可以覆蓋到的區域,命令如下
cnvkit.py access \
hg19.fa \
-x excludes.bed \
-o access.hg19.bed計算出可覆蓋的區域之后,減去in-target區域, 就可以得到off-target區域,通過antitarget子命令來實現,代碼如下
cnvkit.py antitarget \
my_targets.bed \
-g access.hg19.bed \
-o my_antitargets.bedcoverage和autobin兩個子命令都可以用來計算測序深度,以coverage為例,用法如下
cnvkit.py coverage \
Sample.bam \
my_targets.bed \
-o Sample.targetcoverage.cnn
cnvkit.py coverage \
Sample.bam \
my_antitargets.bed \
-o Sample.antitargetcoverage.cnn分別統計target和antitarget區域的測序深度信息,輸出結果后綴為cnn,是cnvkit中定義的一種格式,專門用來存儲測序深度信息。
通過reference子命令來構建正常基因組的測序分布模型,采用對照樣本的測序深度,校正GC含量等系統誤差。有多個對照樣本時,可以將所有的對照樣本合并來創建,用法如下
cnvkit.py reference \
*coverage.cnn \
-f hg19.fa \
-o Reference.cnn當沒有對照樣本時,軟件可以模擬出一個正常的測序深度分布模型,用法如下
cnvkit.py reference \
-o FlatReference.cnn \
-f hg19.fa \
-t my_targets.bed \
-a my_antitargets.bed通過fix子命令計算log2 ratio,用法如下
cnvkit.py fix \
Sample.targetcoverage.cnn \
Sample.antitargetcoverage.cnn \
Reference.cnn \
-o Sample.cnr輸出結果后綴為cnr, 是cnvkit中定義的一種格式,專門用來存儲log2ratio的信息。
通過segment子命令進行segment的劃分,用法如下
cnvkit.py segment \
Sample.cnr \
-o Sample.cns輸出結果后綴為cns, 是cnvkit中定義的一種格式,和SEG格式類似,用來存儲CNV分析的結果。接下來還可以通過call子命令,計算每個segment區域的絕對拷貝數,用法如下
cnvkit.py call \
Sample.cns \
-o Sample.call.cns關于各種文件格式的具體解釋可以參考以下鏈接
https://cnvkit.readthedocs.io/en/stable/fileformats.html
提供了以下3種可視化的子命令
diagram
scatter
heatmap
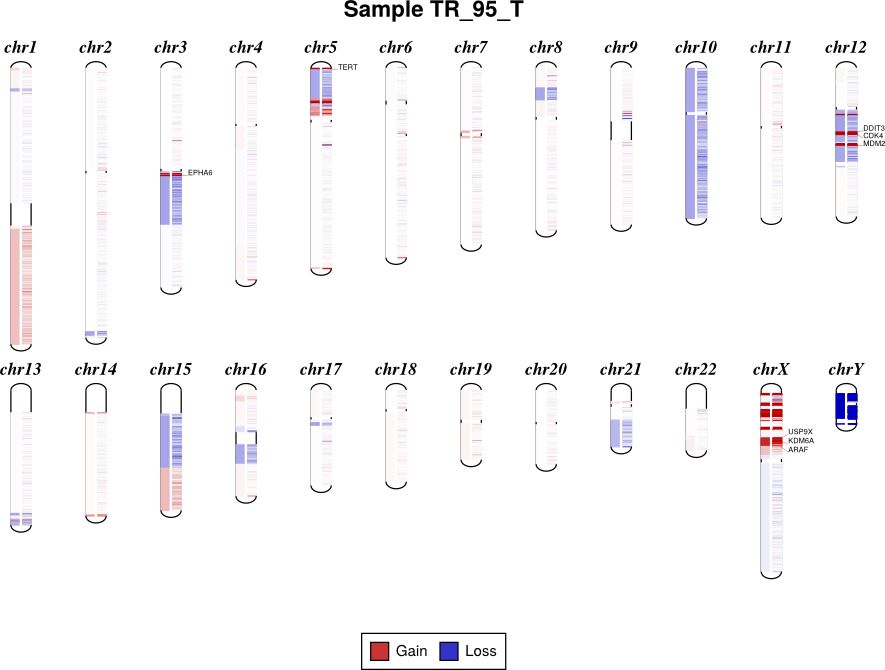
diagram用于展示單個樣本的CNV在染色體上的分布,用法如下
cnvkit.py diagram \
-s Sample.cns \
Sample.cnr可視化結果如下
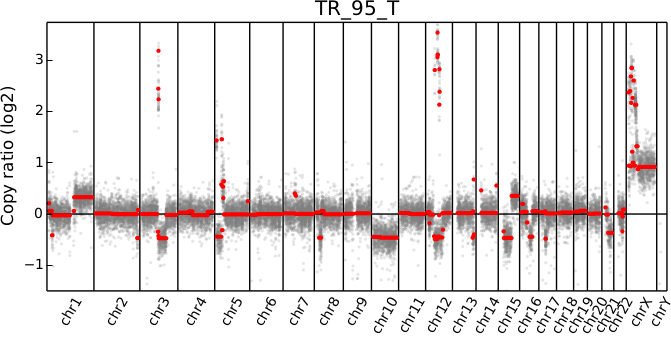
scatter子命令展示單個樣本染色體區域上log2 ratio值的分布,用法如下
cnvkit.py scatter \
-s Sample.cns \
Sample.cnr可視化結果示意如下
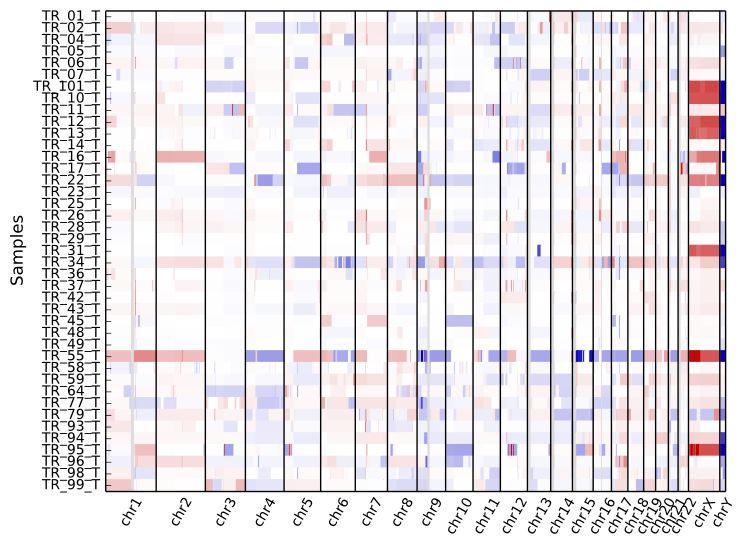
heatmap子命令展示多個樣本的CNV分布情況,用法如下
cnvkit.py heatmap *.cns
可視化結果示意如下
分布運行步驟繁瑣,上面的代碼可以通過batch一個子命令搞定,用法如下
cnvkit.py batch \
*Tumor.bam \
--normal *Normal.bam \
--targets my_baits.bed \
--annotate refFlat.txt \
--fasta hg19.fasta \
--access access.hg19.bed \
--output-reference my_reference.cnn \
--output-dir results/ \
--diagram --scatter到此,相信大家對“如何使用CNVkit進行CNV分析”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。