您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎么使用EXCAVATOR2檢測WES的CNV,針對這個問題,這篇文章詳細介紹了相對應的分析和解答,希望可以幫助更多想解決這個問題的小伙伴找到更簡單易行的方法。
excavator2是一款利用WES數據進行CNV分析的軟件,其他同類軟件通常只關注捕獲的exon區域,而該軟件則進行了延伸,將捕獲區域劃分為exon和非exon區域兩部分,在校正測序深度的分布時對這兩部分區域分別分別進行處理,對應的文章發表在Nucleic Acids Research上,鏈接如下
https://academic.oup.com/nar/article/44/20/e154/2607979
該軟件的源代碼保存在sourceforge上,鏈接如下
https://sourceforge.net/projects/excavator2tool/
excavator2在計算測序深度時將reads分為了以下兩個部分
in-target reads
off-target reads
in-target表示的是位于exon上的序列,off-target表示的是位于基因間區或者內含子區的序列,同樣采用了滑動窗口的方式來統計每個區域的測序深度,只不過稍作變化,全稱如下
mean windows read count
簡稱WMRC, 計算公式如下

單個外顯子直接作為一個窗口,而非外顯子區域則采用了一個固定長度的窗口,分開統計不同區域的測序深度,并進行校正,校正的時候考慮了GC含量,不同區域的mappability, 外顯子的大小等因素。
利用歸一化之后的測序深度,計算對照樣本和實驗樣本的log2 ratio值,然后采用HSLM segmentation算法劃分segment, 最后通過FastCall算法預測每個segment的拷貝數情況,細分為以下5種
two-copy deletion
one-copy deletion
normal
one-copy duplication
multiple-copy duplicaiton
該軟件支持hg19和hg38兩個版本,內置了對應的數據庫,示意如下
該軟件分成了3個模塊,對應3個腳本,具體操作步驟如下
提供一個捕獲區域的bed文件,計算in-target和off-target區域的GC含量,mappability值,用于后續的歸一化操作,用法如下
perl TargetPerla.pl \
SourceTarget.txt \
myTarget.bed \
MyTarget_w50000 \
50000 \
hg19第一個參數為source target文件,該文件記錄了基因組對應的bw文件和fasta文件的路徑,內容示意如下
/data/ucsc.hg19.bw /data/hg19.fasta
空格分隔的兩列,第一列為bw文件的路徑,該文件是軟件自帶的,位于軟件的安裝目錄,用于計算基因組不同區域的mappability,第二列為fasta文件的路徑,用于計算不同區域的GC含量。
第二個參數為捕獲區域的bed文件,第三個參數為輸出結果的前綴,第四個參數為窗口的固定長度,第五個參數指定基因組的版本。
這一步類似于比對時建立參考基因組的索引,一個芯片建立一次即可,運行成功后,會生成一個文件夾,前綴為MyTarget_w50000。
計算測序深度,進行歸一化處理,用法如下
perl EXCAVATORDataPrepare.pl \
ExperimentalFile.txt \
--processors 6 \
--target MyTarget_w50000 \
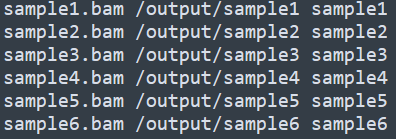
--assembly hg19第一個參數是一個空格分隔的txt文件,指定了樣本對應的bam文件,輸出結果的路徑,樣本名稱信息,內容示意如下
--processors指定并行的線程數,--target參數指定第一步生成的target的名稱,--assembly指定參考基因組的版本。
執行HSLM segmentation算法和FastCall算法,進行CNV分析,用法如下
perl EXCAVATORDataAnalysis.pl \
ExperimentalFileAnalysis.txt \
--processors 6 \
--target MyTarget_w50000 \
--assembly hg19 \
--output Results_MyProject_w50K \
--mode pooling--mode參數表示樣本如何進行比較,支持pooling和paired兩種模式,第一種模式將所有的實驗樣本混合與對照樣本進行比較,第二種模式則是配對樣本模式,比如癌和癌旁,兩兩之間進行比較,計算log2 ration值。
第一個參數ExperimentalFileAnalysis.txt是一個空格分隔的txt文件,指定了樣本的比較操作,對于pooling模式,其內容示意如下
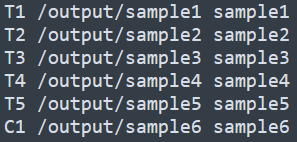
對于paired模式,其內容示意如下

T表示Treat, C表示Control, 后面的數字用于區分不同樣本。
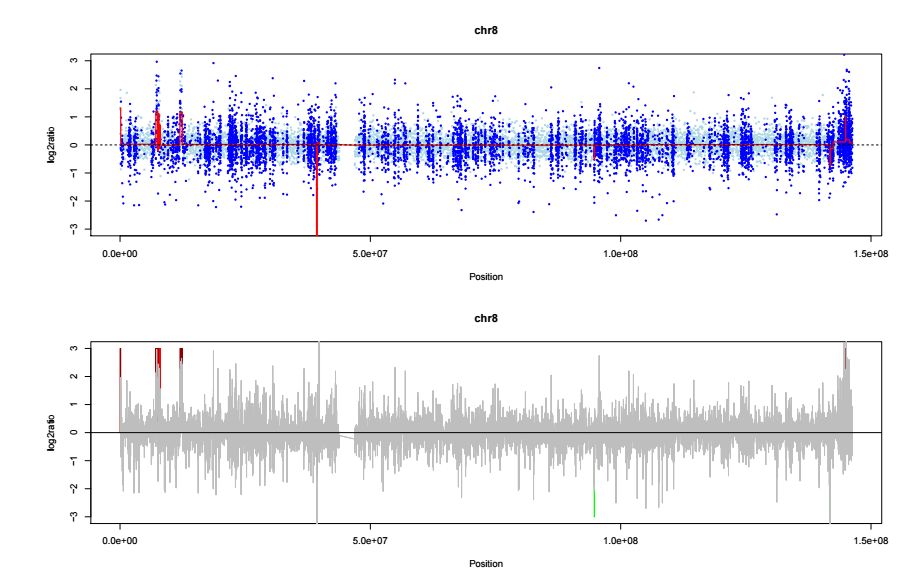
--output指定了輸出結果的目錄,輸出結果中提供了CNV區域對應的txt, VCF等文件,同時還提供了可視化的結果,示意如下
關于怎么使用EXCAVATOR2檢測WES的CNV問題的解答就分享到這里了,希望以上內容可以對大家有一定的幫助,如果你還有很多疑惑沒有解開,可以關注億速云行業資訊頻道了解更多相關知識。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。