您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“如何使用WashU Epigenome Browser可視化hi-c數據”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“如何使用WashU Epigenome Browser可視化hi-c數據”吧!
Washu Epigenome Browser是一款基因組瀏覽器,相比UCSC等基因組瀏覽器,其不僅能夠展示RNA_seq, chip_seq等二維信息,還可以展示Hi-C等三維基因組學數據分析結果,網址如下
http://epigenomegateway.wustl.edu/browser/
該瀏覽器的功能特別強大,本文只是介紹下如何通過這個瀏覽器來展示Hi-C數據,步驟如下
內置了人,小鼠,大鼠等常用物種,在展示信息之前,首先需要選擇對應的參考物種和基因組版本,如下圖所示

選擇好參考物種之后,下一步就是添加需要展示的信息,每個信息稱之為track。 通過導航欄的Tracks進行添加,既可以從公共數據庫選擇,也支持自己上傳數據。這里我們從ENCODE數據庫選擇數據進行展示,通過Public Data Hubs選擇公共數據集, 如下圖所示,點擊右邊的加號選擇感興趣的數據集
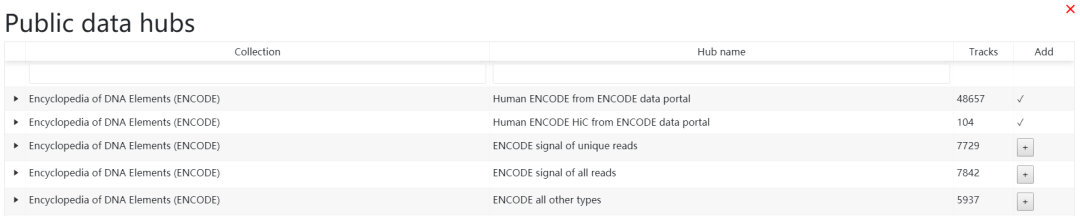
在下方提供了數據集的視圖,通過加號展開對應的數據集,然后點擊右側的Assay區域可以進一步選擇數據集
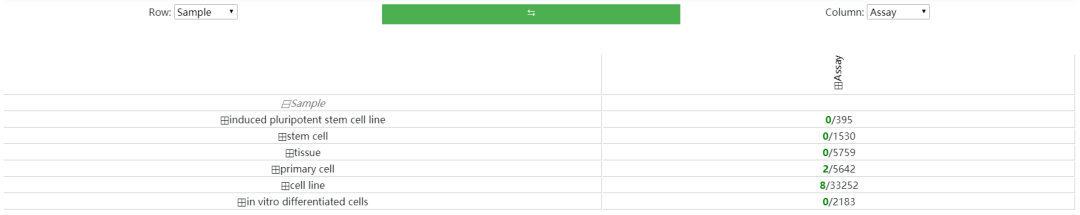
這里我們選擇IM90細胞系相關數據,示意如下
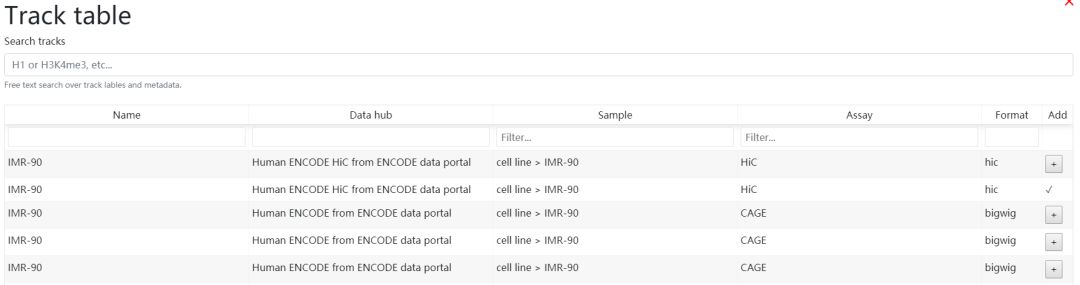
可以多選,這里我選擇了hic和多種組蛋白修飾和CTCF的chip_seq以及RNA_seq的數據即興展示。
默認情況下track的順序是按照導入的依次順序進行排列,通過下圖紅色方框標記的工具集可以調整tracks的排列順序

其次鼠標右鍵點擊對應的track, 可以對其顏色,y軸范圍等進行詳細調整,如下圖所示
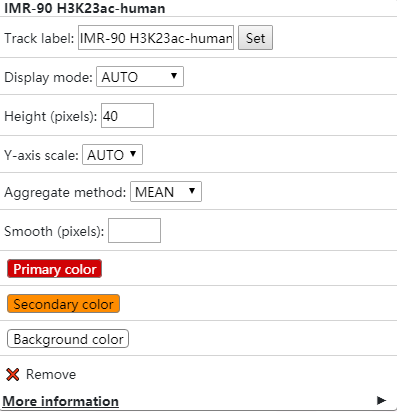
簡單調整了每種track的顏色,效果圖如下所示
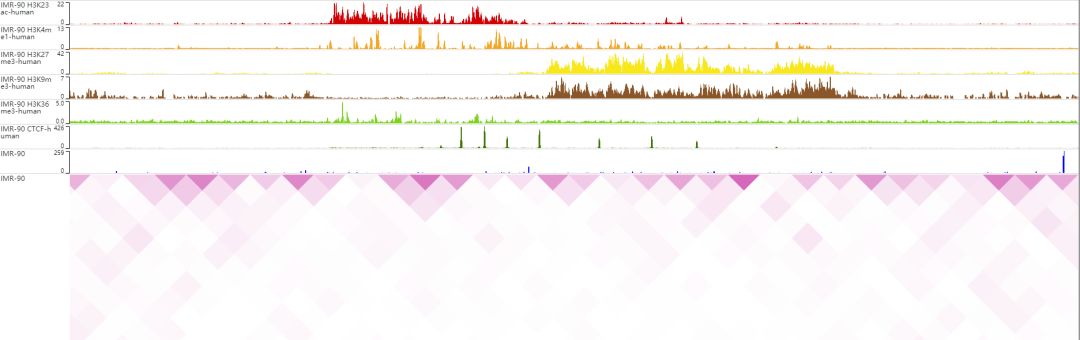
到此,相信大家對“如何使用WashU Epigenome Browser可視化hi-c數據”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。