您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用TADbit識別拓撲關聯結構域”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用TADbit識別拓撲關聯結構域”吧!
TADbit是一個hi-c數據分析的軟件,提供了從原始數據處理到染色質三維模型構建的完整功能,對應的文章鏈接如下
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5540598/
該軟件的pipeline如下圖所示
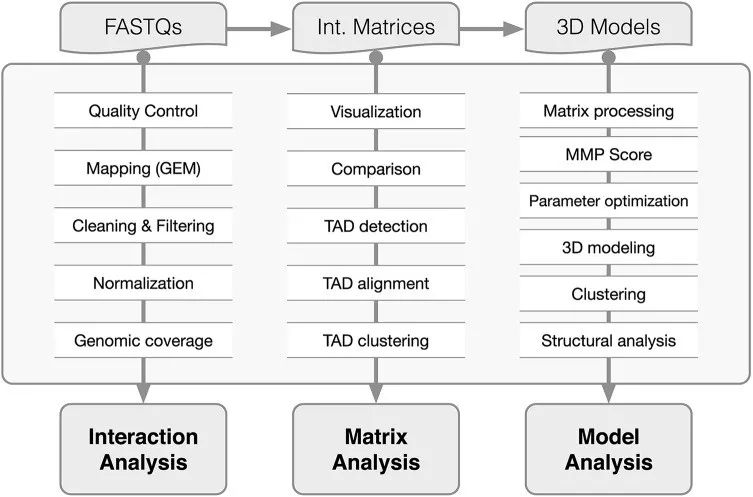
總體分成以下3個功能模塊
FASTQ
Interacton Matrix
3D Models
第一個模塊從原始的fastq文件開始,對序列進行質量過濾,采用GEM軟件將clean reads比對參考基因組,然后進行篩選,構建原始的交互矩陣,并進行歸一化處理,得到歸一化之后的交互矩陣。
第二個模塊用于可視化hi-c交互矩陣,并且可以在交互矩陣的基礎上,識別TAD拓撲關聯結構域,對TAD進行可視化,聚類等分析。
第三個模塊用于構建染色質三維構象的模型,并進行結構分析。
本文簡單整理下第二個模塊的具體用法,詳細步驟如下
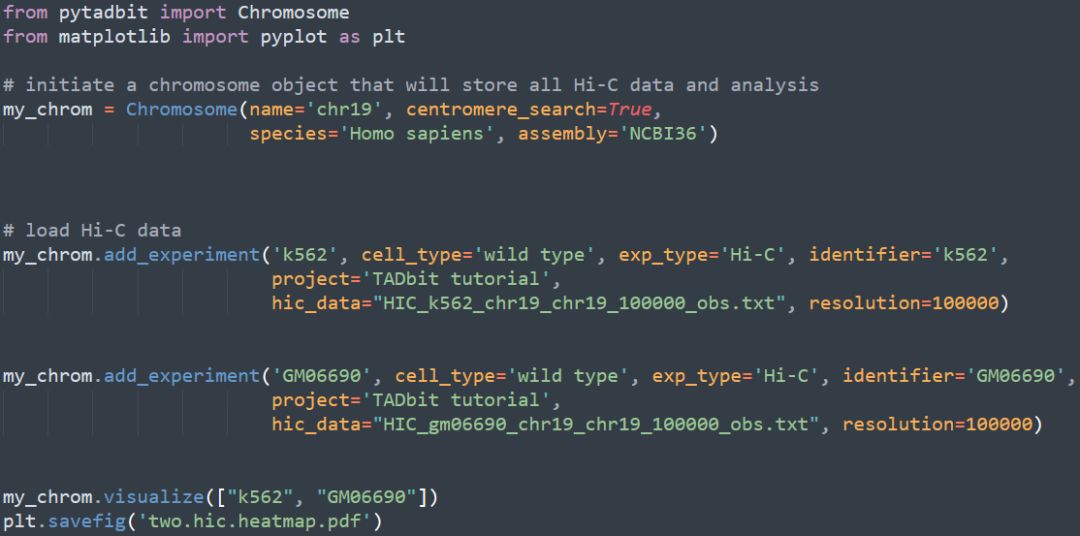
該軟件采用python進行開發,采用了面向對象的編程思想,首先要做的就是構建一個object, 構建的過程中需要對應的hi-c交互矩陣, 軟件自帶的測試數據集包含了以下兩個hi-c矩陣
HIC_gm06690_chr19_chr19_100000_obs.txt
HIC_k562_chr19_chr19_100000_obs.txt
對應GM06690和K562兩種細胞系19號染色體100kb分辨率下的交互矩陣。基于這兩個交互矩陣構建對象并可視化的代碼如下
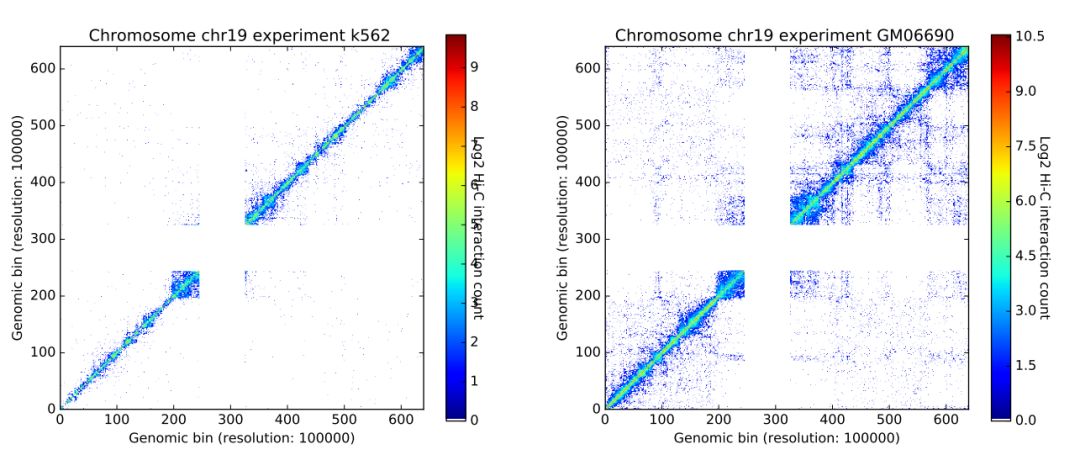
可視化的效果圖如下
有兩種可視化的策略,第一種是在hi-c的熱圖上用矩形標記TAD區域,第二種稱之為density plot, 用法如下
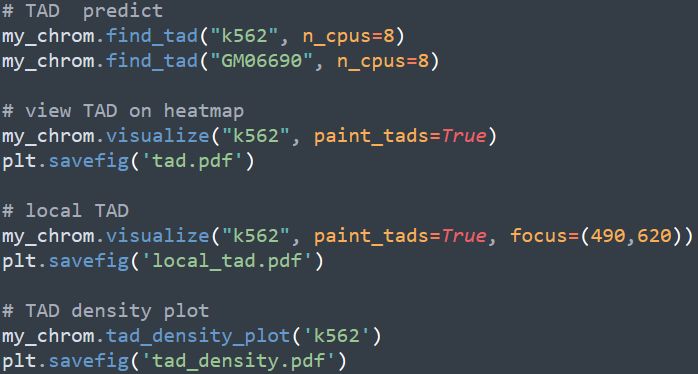
熱圖標記TAD之后的效果圖如下
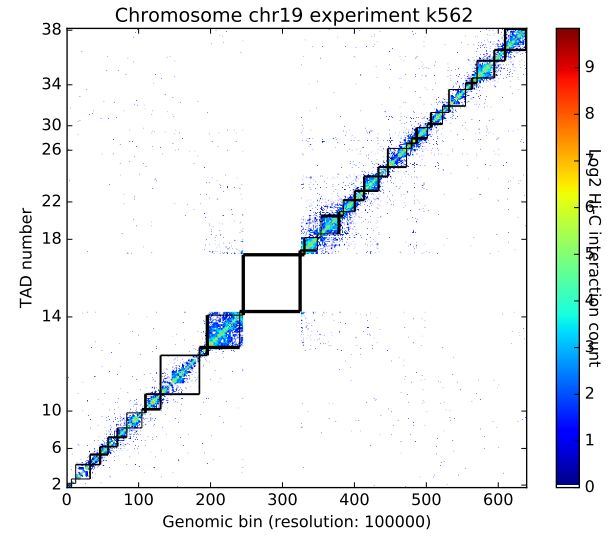
density plot的效果圖如下
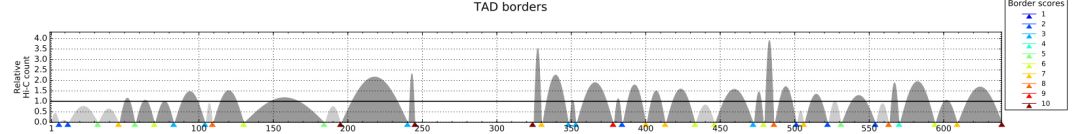
將多個細胞或組織的TAD進行比較,可以分析其位置是否具有保守性。用法如下
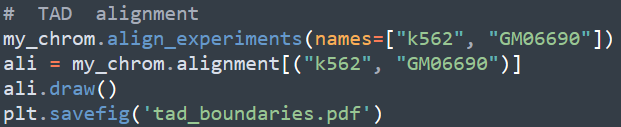
效果圖如下所示
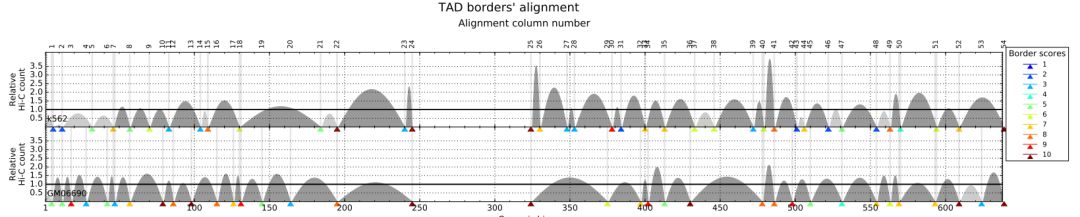
TADbit的用法簡單,可視化效果也很棒,唯一的缺點就是安裝特別費勁。
感謝各位的閱讀,以上就是“如何使用TADbit識別拓撲關聯結構域”的內容了,經過本文的學習后,相信大家對如何使用TADbit識別拓撲關聯結構域這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。