您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“如何使用ChIPpeakAnno進行peak注釋”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“如何使用ChIPpeakAnno進行peak注釋”吧!
ChIPpeakAnno是一個bioconductor上的R包,針對peak calling之后的下游分析,提供了以下多種功能
查找與peak區域最相鄰的基因, 也支持自定義查找的特征,可以是exon,miRNA等
peak相鄰基因的GO富集分析
提取peak及其周圍區域的序列
在ChIPpeakAnno中,無論是peak區間信息還是基因組的注釋信息,都通過toGRanges方法轉化為R語言中的GRanges對象,以peak為例,bed格式的內容如下
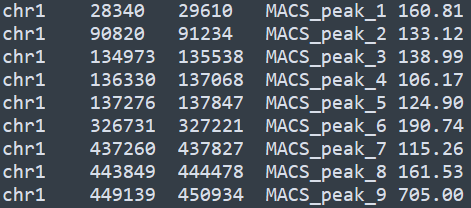
通過如下代碼可以導入該信息
library(ChIPpeakAnno)
bed <- "peaks.bed"
gr <- toGRanges(bed, format="BED", header=FALSE)除了BED格式外,該方法也支持導入GTF格式的信息,只需要修改format參數即可。導入peak信息和基因組注釋信息后就可以進行后續分析了。
當導入了多個樣本的peak信息時,可以進行venn分析,用法如下
# 導入A樣本的peak
bedA <- "sampleA_peaks.bed"
sampleA <- toGRanges(bedA, format="BED", header=FALSE)
# 導入B樣本的peak
bedB <- "sampleB_peaks.bed"
sampleB <- toGRanges(bedB, format="BED", header=FALSE)
# 求交集
ol <- findOverlapsOfPeaks(sampleA, sampleB)
# 繪制venn圖
makeVennDiagram(ol)結果示意如下
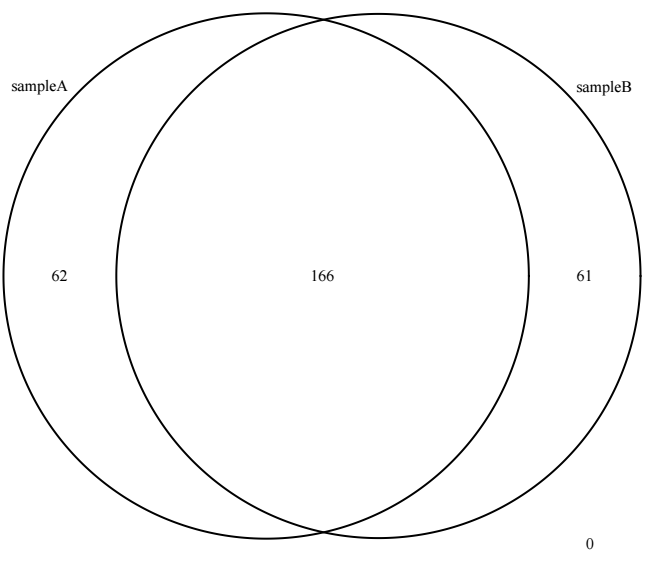
在進行venn分析時,會發現venn圖上的個數加起來并不是輸入的peak區間的總數,在默認
用法如下
library(BSgenome.Hsapiens.UCSC.hg19)
seq <- getAllPeakSequence(sampleA, upstream=20, downstream=20, genome=Hsapiens)
write2FASTA(seq, "sampleA.peaks.fa")提取到peak序列之后,可以進行motif分析,用法如下
# 用1號染色體的堿基分布當做背景
freqs <- oligoFrequency(Hsapiens$chr1, MarkovOrder=3)
# oligoLength規定了motif的長度
os <- oligoSummary(seq, oligoLength=6, MarkovOrder=3,
quickMotif=TRUE, freqs=freqs)
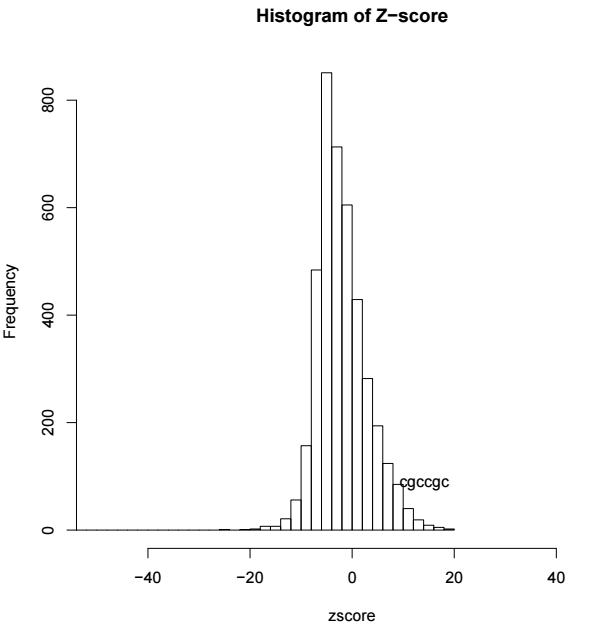
zscore <- sort(os$zscore)
# 繪制所有6個堿基組合的頻率分布圖
h <- hist(zscore, breaks=100, xlim=c(-50, 50), main="Histogram of Z-score")
# 頻率最大的堿基組合即為motif的結果
text(zscore[length(zscore)], max(h$counts)/10,
labels=names(zscore[length(zscore)]), adj=1)結果示意如下
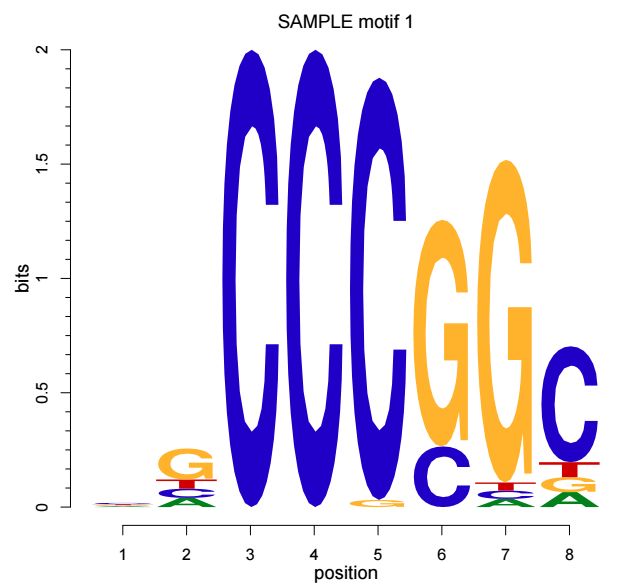
還可以通過motifStack這個R包繪制motif的sequence logo, 用法如下
library(motifStack)
pfms <- mapply(function(.ele, id)
new("pfm", mat=.ele, name=paste("SAMPLE motif", id)),
os$motifs, 1:length(os$motifs))
motifStack(pfms[[1]])輸出結果示意如下
首先是peak在基因組各個特征區間的分布比例,用法如下
library(TxDb.Hsapiens.UCSC.hg19.knownGene)
aCR<-assignChromosomeRegion(sampleA, nucleotideLevel=FALSE,
precedence=c("Promoters", "immediateDownstream",
"fiveUTRs", "threeUTRs",
"Exons", "Introns"),
TxDb=TxDb.Hsapiens.UCSC.hg19.knownGene)
barplot(aCR$percentage, las=3)輸出結果如下所示
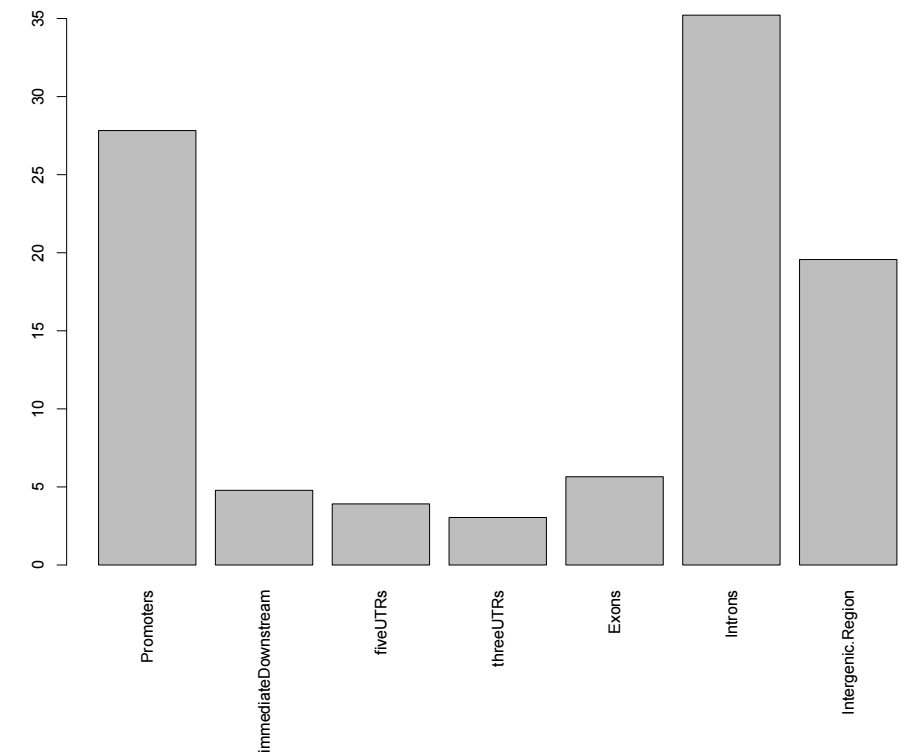
然后進行peak關聯基因的注釋,用法如下
# 準備基因組注釋信息
library(EnsDb.Hsapiens.v75)
annoData <- toGRanges(EnsDb.Hsapiens.v75, feature="gene")
# 進行
overlaps.anno <- annotatePeakInBatch(sampleA,
AnnotationData=annoData,
output="nearestLocation"
)
library(org.Hs.eg.db)
overlaps.anno <- addGeneIDs(overlaps.anno,
"org.Hs.eg.db",
IDs2Add = "entrez_id")
pie1(table(overlaps.anno$insideFeature))輸出結果示意如下
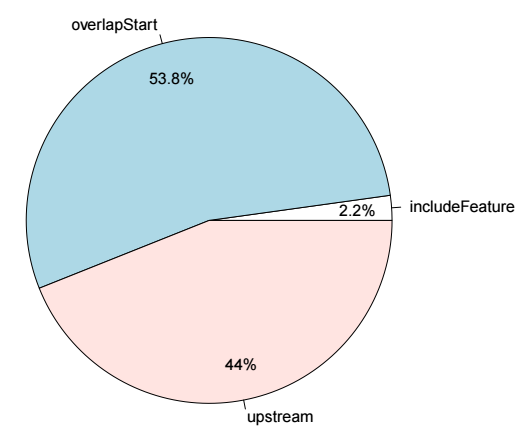
在使用annotatePeakInBatch進行注釋時,默認查找距離peak最近的基因,也可以修改output的值,overlapping代表與peak區域存在overlap的基因,設置成這個值之后就會將與peak區間存在overlap的基因作為關聯基因了,此外還有多種取值,適用不同條件,具體可以參考函數的幫助文檔。
進行完基因注釋之,得到peak關聯的基因,就可以進行后續的功能富集分析,用法如下
over <- getEnrichedGO(overlaps.anno, orgAnn="org.Hs.eg.db",
maxP=.05, minGOterm=10,
multiAdjMethod="BH", condense=TRUE)ChIPpeakAnno提供了一條完整的peak下游分析功能,包括基因注釋,富集分析,motif分析等等,是一個非常強大的工具,以上只是基本用法,更多用法和細節請參考官方文檔。
感謝各位的閱讀,以上就是“如何使用ChIPpeakAnno進行peak注釋”的內容了,經過本文的學習后,相信大家對如何使用ChIPpeakAnno進行peak注釋這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。