您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇文章為大家展示了怎么使用IRESfinder分析環狀RNA的蛋白編碼潛能,內容簡明扼要并且容易理解,絕對能使你眼前一亮,通過這篇文章的詳細介紹希望你能有所收獲。
真核生物mRNA的5’ 帽子結構可以介導核糖體的結合,從而開啟翻譯過程,circRNA由于是一個閉合的環狀結構,缺失了5’端帽子結構,所以歸類為非編碼RNA的一種。
隨著環狀RNA研究的深入,有科學家發現部分環狀RNA可以編碼蛋白。除了常見的5’帽子結構介導核糖體結合外,還存在了一種特殊情況,在一些基因上存在一段長度在150-250bp的序列,這些序列能夠折疊成類似tRNA的結構,介導核糖體與RNA結合,起始蛋白質的翻譯。這樣的位點稱之為內部核糖體進入位點序列,Internal ribosome entry site, 簡稱IRES。
能夠編碼蛋白的環狀RNA上就是通過IRES來實現翻譯過程的,為例研究環狀RNA的蛋白編碼潛能,有學者開發了識別IRES的軟件,IRESfinder就是這樣一款軟件,用于識別真核生物的IRES位點。
利用實驗驗證過的583個人類的IRES序列,挑選了19個kmer用于區分IRES和非IRES的序列,在論文中,給出了測試數據集中二種序列的kmer分布
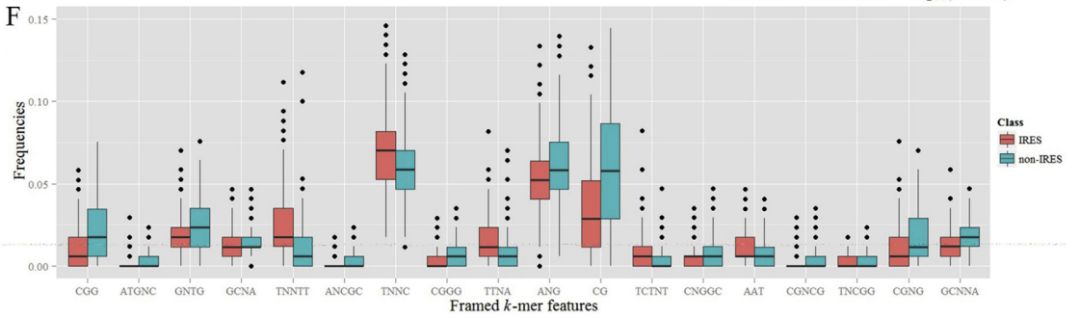
在上圖中,只包含了18個kmer,另外還有一個kmer是T,可以看到在IRES和非IRES序列之間,這些kmer的頻率分布是有差異的。
軟件采用Python進行開發,基本用法如下
python IRESfinder.py -f circRNA.fa -o IRES.out.xls
-f參數指定輸入的fasta格式的序列,-o參數指定輸出的結果文件。輸出文件的內容示意如下
ID Index Score hsa_circ_0018046 IRES 0.817344005 hsa_circ_0039868 IRES 0.795083668 hsa_circ_0089160 IRES 0.53322605 hsa_circ_0048972 IRES 0.784080068 hsa_circ_0018658 IRES 0.745230164 hsa_circ_0067857 IRES 0.704497116 hsa_circ_0019137 IRES 0.742607966 hsa_circ_0063162 IRES 0.738372532 hsa_circ_0087609 IRES 0.793042932 hsa_circ_0006254 IRES 0.64587644
通過這個軟件,可以快速分析得到RNA上的IRES序列信息,不過軟件的結果中假陽性肯定是很高的,后續還要通過實驗手段來驗證。
上述內容就是怎么使用IRESfinder分析環狀RNA的蛋白編碼潛能,你們學到知識或技能了嗎?如果還想學到更多技能或者豐富自己的知識儲備,歡迎關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。