您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關CNCI工具有什么用,小編覺得挺實用的,因此分享給大家做個參考,希望大家閱讀完這篇文章后可以有所收獲。
CPC是一款使用率非常高的lncRNA預測軟件,但是它也存在一些問題。利用二代測序得到的轉錄組數據,我們組裝得到的轉錄本往往是不完整的,基于非全長的轉錄本去預測lncRNA,如果這個lncRNA和蛋白編碼基因存在overlap,那么很容易造成誤判;其次對于沒有物種注釋的物種,其效果也很差。
為了克服上述問題,研究人員開發出了一款新的工具CNCI, 和CPC不同,該軟件基于三聯體堿基的構成來區分coding和noncoding轉錄本,論文發表在Nucleic Acids Research上,網址如下
https://academic.oup.com/nar/article/41/17/e166/2411728
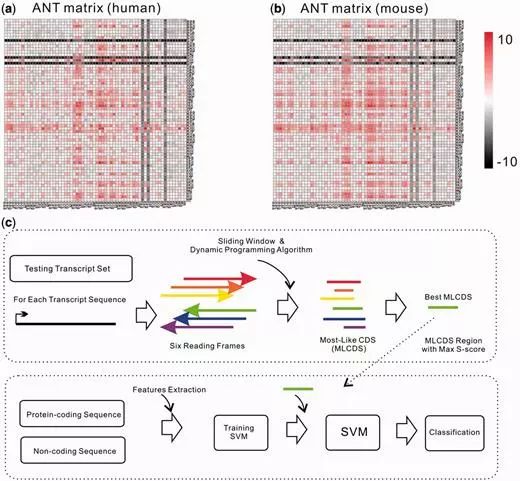
三聯體堿基指的就是三個連續的堿基,和密碼子類似,稱之為ANT, 該軟件利用人和小鼠的轉錄本數據,構建了一個支持向量機的模型,用于對脊椎動物進行分類,示意如下
對于不同長度的轉錄本序列,和其他軟件的性能比較如下
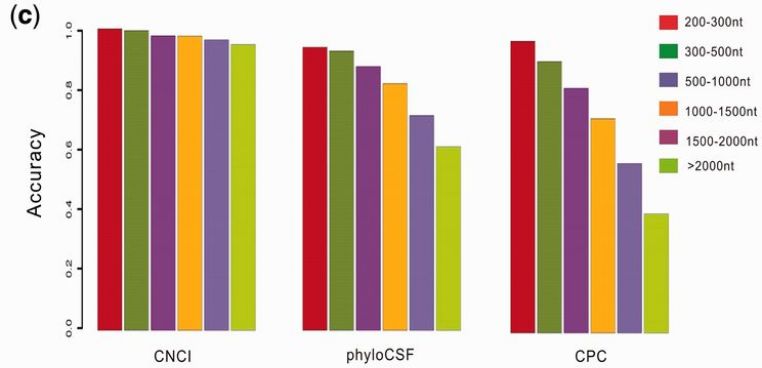
可以看到CNCI在各個長度區間性能都比較好。該軟件的源代碼保存在github上,網址如下
https://github.com/www-bioinfo-org/CNCI
安裝方式如下
git clone git@github.com:www-bioinfo-org/CNCI.git cd CNCI unzip libsvm-3.0.zip cd libsvm-3.0 make
CNCI的執行腳本是采用python開發的,直接用就可以的,但是該軟件依賴libsvm, 所以需要安裝這個庫文件。基本用法如下
python CNCI.py \ -f transcript.fasta \ -o test \ -m ve \ -p 8 \
-f指定轉錄本序列文件,可以是fasta格式,也可以是gtf格式,如果是gtf格式,需要同時指定-g和-d參數;-p參數指定并行的CPU個數;-m指定使用的模型,ve代表脊椎動
物,p代表植物;-o指定輸出結果的目錄。
在結果目錄下,有一個名為CNCI.index的文件,內容示意如下
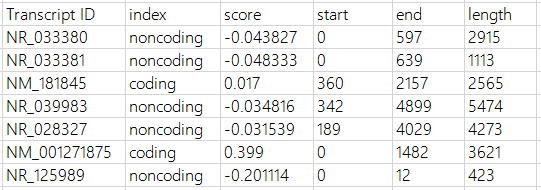
第二列表明轉錄本的分類結果。更多用法和詳細信息請參考官方文檔。
關于“CNCI工具有什么用”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,使各位可以學到更多知識,如果覺得文章不錯,請把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。