您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎樣使用Clustal進行多序列比對,相信很多沒有經驗的人對此束手無策,為此本文總結了問題出現的原因和解決方法,通過這篇文章希望你能解決這個問題。
多序列比對在保守區域鑒定,系統發育分析,motif識別等多個領域發揮重要作用,是生物信息數據分析必備的基礎技能之一。Clustal是一款經典的多序列比對工具,支持DNA, RNA, 蛋白質的比對。
clustal 有兩個版本可用,之前的版本同時提供了GUI和命令行兩種工具,GUI版的叫做ClustalX, 命令行版叫做ClustalW; 最新版本叫做Omega, 只提供了命令行版。

最新本的omega比對準確度更高,而且速度更快,適合幾千條規模的多序列比對,該軟件目前只提供了命令行版本。在官網上,提供了源代碼和編譯好的二進制文件
通常情況下,直接下載對應的二進制可執行文件就行了。軟件的基本用法如下:
clustalo -i seq.fasta > align.fa
-i指定輸入的序列文件,默認輸出結果打印在屏幕上,可以重定向到指定文件中。該軟件支持多種格式的輸出
fasta
clustal
msf
phylip
selex
stockholm
vienna
默認輸出格式為fasta, 可以通過--outfmt參數指定輸出文件的格式。多序列比對不同于Blast的地方在于,Blast是局部比對,而多序列比對是全局比對。全局比對意味著需要將輸入序列對齊到同一個水平來比對,一般是通過在輸入序列中插入堿基的方式來使序列對齊,示意如下
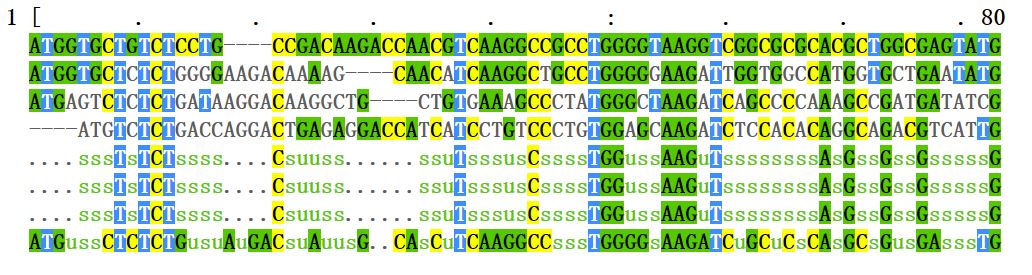
>ENA|CAA23748|CAA23748.1 Homo sapiens (human) alpha globin ATGGTGCTGTCTCCTG----CCGACAAGACCAACGTCAAGGCCGCCTGGGGTAAGGTCGG CGCGCACGCTGGCGAGTATGGTGCGGAGGCCCTGGAGAGGATGTTCCTGTCCTTCCCCAC CACCAAGACCTACTTCCCGCACTTC---GACCTGAGCCACGGCTCTGCCCAAGTTAAGGG CCACGGCAAGAAGGTGGCCGACGCGCTGACCAACGCCGTGGCGCACGTGGACGACATGCC CAACGCGCTGTCCGCCCTGAGCGACCTGCACGCGCACAAGCTTCGGGTGGACCCGGTCAA CTTCAAGCTCCTAAGCCACTGCCTGCTGGTGACCCTGGCCGCCCACCTCCCCGCCGAGTT CACCCCTGCGGTGCACGCTTCCCTGGACAAGTT---CCTGGCTTCTGTGAGCACCGTGCT GACCTCCAAATACCGTTAA >ENA|CAA24095|CAA24095.1 Mus musculus (house mouse) alpha-globin ATGGTGCTCTCTGGGGAAGACAAAAG----CAACATCAAGGCTGCCTGGGGGAAGATTGG TGGCCATGGTGCTGAATATGGAGCTGAAGCCCTGGAAAGGATGTTTGCTAGCTTCCCCAC CACCAAGACCTACTTTCCTCACTTTGATGT---AAGCCACGGCTCTGCCCAGGTCAAGGG TCACGGCAAGAAGGTCGCCGATGCGCTGGCCAGTGCTGCAGGCCACCTCGATGACCTGCC CGGTGCCTTGTCTGCTCTGAGCGACCTGCATGCCCACAAGCTGCGTGTGGATCCCGTCAA CTTCAAGCTCCTGAGCCACTGCCTGCTGGTGACCTTGGCTAGCCACCACCCTGCCGATTT CACCCCCGCGGTACATGCCTCTCTGGACAAATT---CCTTGCCTCTGTGAGCACCGTGCT GACCTCCAAGTACCGTTAA >ENA|BAA20512|BAA20512.1 Cyprinus carpio (common carp) alpha-globin ATGAGTCTCTCTGATAAGGACAAGGCTG----CTGTGAAAGCCCTATGGGCTAAGATCAG CCCCAAAGCCGATGATATCGGCGCTGAAGCTCTCGGCAGAATGCTGACCGTCTACCCTCA GACCAAGACCTACTTCGCTCACTGGGATGACCTGAGCCCTGGGTCCGGTCCTGTGAAGAA GCATGGCAAGGTTATCATGGGTGCAGTGGCCGATGCCGTTTCAAAAATAGACGACCTTGT GGGAGGTCTGGCCTCCCTGAGCGAACTTCATGCTTCCAAGCTGCGTGTTGACCCGGCCAA CTTCAAGATCCTCGCACACAATGTCATCGTGGTCATCGGCATGCTCTTCCCTGGAGACTT CCCCCCAGAGGTTCACATGTCAGTTGACAAGTTTTTCCAGAAC---TTGGCTCTGGCTCT CTCTGAGAAGTACCGCTAA
通過在序列中插入-來使得序列對齊。如果不習慣命令行的操作方式,也有在線服務可以使用。EBI提供的在線服務網址如下
https://www.ebi.ac.uk/Tools/msa/clustalo/
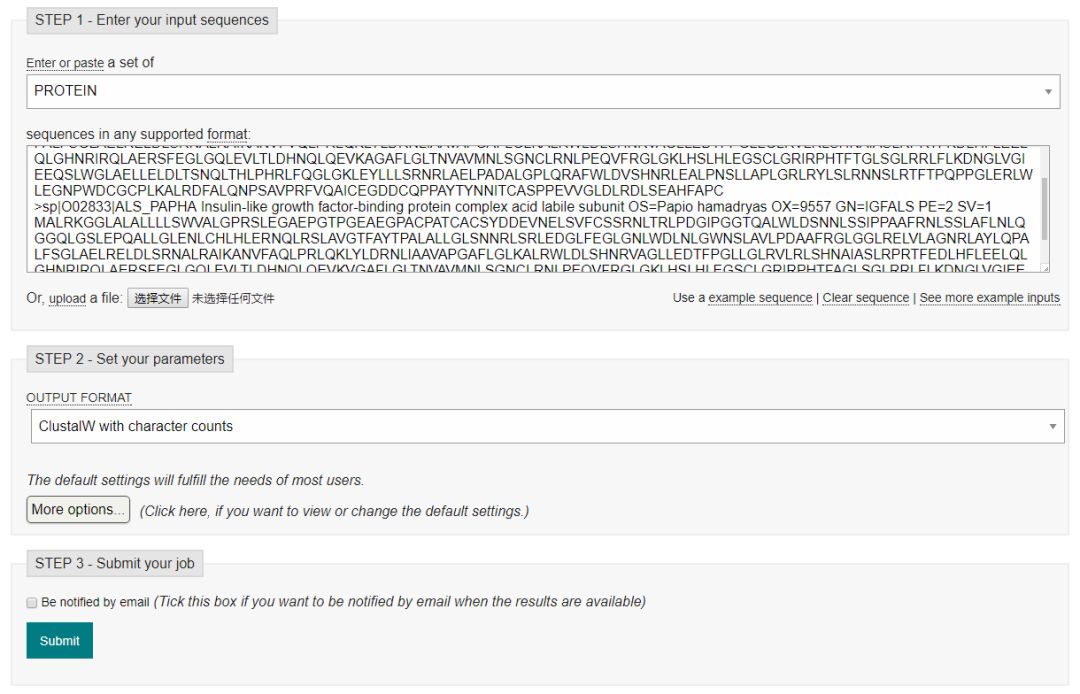
使用非常簡單,輸入序列,調整參數設置,然后提交即可。在輸出結果中,還提供了顏色標記,進化樹可視化等功能。
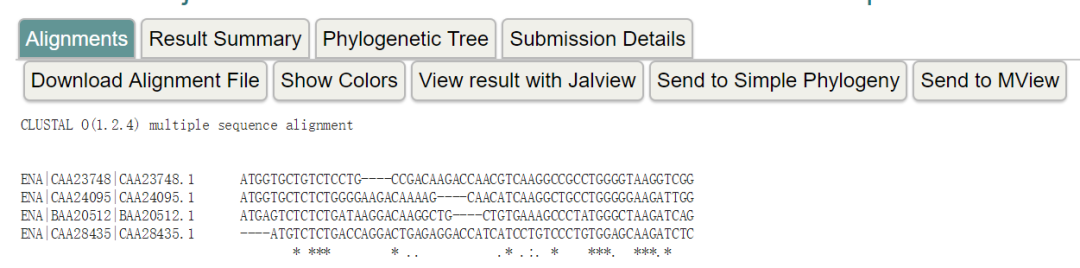
通過Mview可視化多序列比對結果,示意如下
也支持導出到Jalview軟件中進行可視化。
通過Phylogenetic Tree可以查看進化樹的結果,默認采用NJ法建樹,示意如下

也可以通過Send to Simple Phylogeny, 創建進化樹,支持NJ和UPGMA兩種建樹方式。
看完上述內容,你們掌握怎樣使用Clustal進行多序列比對的方法了嗎?如果還想學到更多技能或想了解更多相關內容,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。