您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
今天就跟大家聊聊有關R語言可視化STRING分析的蛋白互作網絡報錯的解決方法,可能很多人都不太了解,為了讓大家更加了解,小編給大家總結了以下內容,希望大家根據這篇文章可以有所收獲。
R語言可視化STRING分析的蛋白互作網絡(PPI)
有一些朋友留言說在重復
net<-graph_from_data_frame(d=links,vertices=nodes,directed = T)
遇到了報錯
Error in graph_from_data_frame(d = links, vertices = nodes, directed = T) : Some vertex names in edge list are not listed in vertex data frame
在這里記錄一下原因和解決辦法
比如我的網絡是四個節點,分別是A,B,C,D,
節點之間兩兩連線
對應的數據應該是
> nodes<-data.frame(node=c("A","B","C","D"))
> edges<-data.frame(node1=c("A","A","A","B","B","C"),
+ node2=c("B","C","D","C","D","D"))
> nodes
node
1 A
2 B
3 C
4 D
> edges
node1 node2
1 A B
2 A C
3 A D
4 B C
5 B D
6 C D
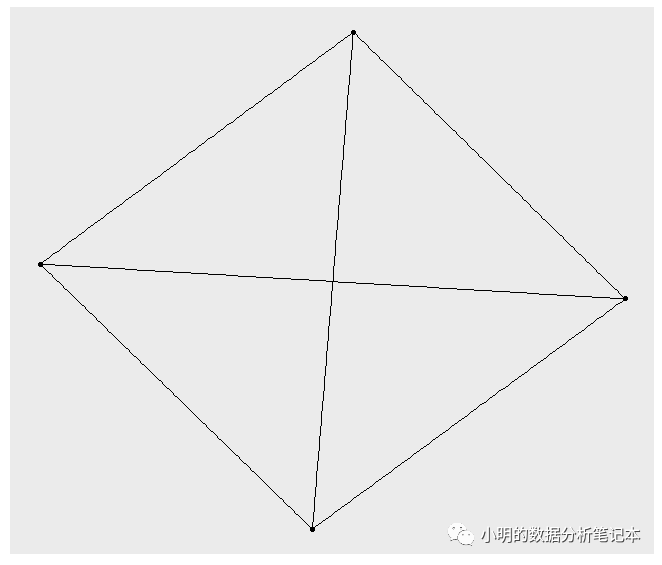
接下來是可視化
library(ggraph)
library(igraph)
net<-graph_from_data_frame(d=edges,vertices = nodes,directed = F)
ggraph(net)+
geom_edge_link()+
geom_node_point()
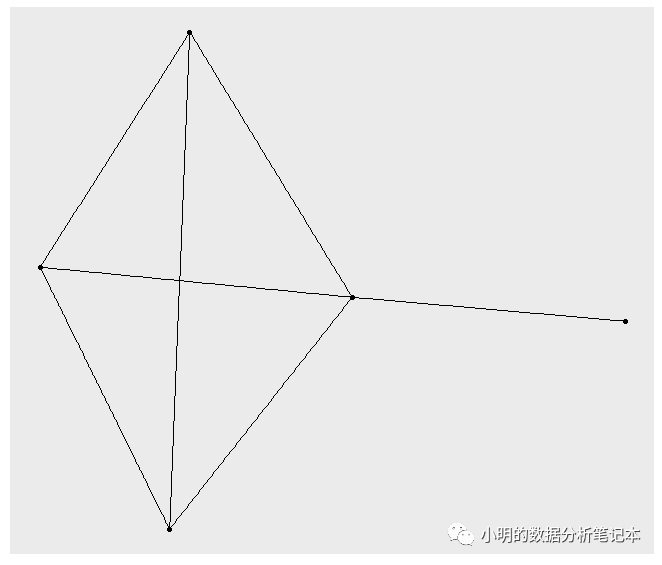
但是如果你的edges文件里出現了nodes文件里沒有的節點,
比如我在向edges這個文件里添加一個C~E的邊
df<-data.frame(node1="C",node2="E")
edges1<-rbind(edges,df)
edges1
node1 node2
1 A B
2 A C
3 A D
4 B C
5 B D
6 C D
7 C E
接下來合并節點和邊的時候就會遇到報錯
> net<-graph_from_data_frame(d=edges1,vertices = nodes,directed = F)
Error in graph_from_data_frame(d = edges1, vertices = nodes, directed = F) :
Some vertex names in edge list are not listed in vertex data frame
可以使用 %in% 來看
比如
> edges1$node1 %in% nodes$node
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE
> edges1$node2 %in% nodes$node
[1] TRUE TRUE TRUE TRUE TRUE TRUE FALSE
> which(edges1$node2 %in% nodes$node)
[1] 1 2 3 4 5 6
> which(! edges1$node2 %in% nodes$node)
[1] 7
這就說明node2這一列第7行沒有在 nodes里。
那我們就可以把這一行刪掉了
df1<-data.frame(node="E")
nodes1<-rbind(nodes,df1)
nodes1
net<-graph_from_data_frame(d=edges1,vertices = nodes1,directed = F)
ggraph(net)+
geom_edge_link()+
geom_node_point()
關于string中蛋白互作網絡輸出文件為什么會出現
邊文件中有的節點不是我們自己輸入的基因id。這個我也不清楚。
我猜,完全是猜
比如你輸入基因 a,b,c ,數據庫中某個基因 d 剛好是鏈接這三個基因的中間點 就像這種,中間是d
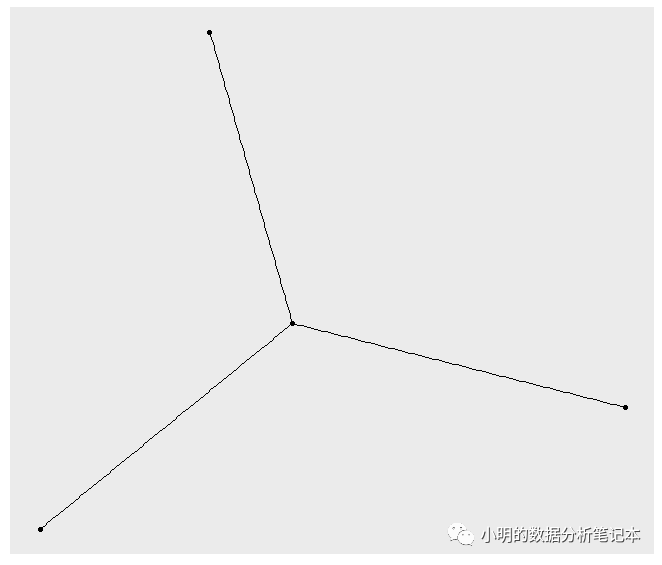
那么最終的網絡文件中就會多出來d 如果是這種情況,就不能在邊文件刪除d而是只能在節點中添加d了。
看完上述內容,你們對R語言可視化STRING分析的蛋白互作網絡報錯的解決方法有進一步的了解嗎?如果還想了解更多知識或者相關內容,請關注億速云行業資訊頻道,感謝大家的支持。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。