您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎么理解GWAS中的genotype imputation,針對這個問題,這篇文章詳細介紹了相對應的分析和解答,希望可以幫助更多想解決這個問題的小伙伴找到更簡單易行的方法。
GWAS用于尋找與疾病或者特定性狀相關聯的SNP位點,為了更加有效的挖掘信息,GWAS需要大樣本量和高密度的SNP分型結果,最佳的分型方案當然是全基因組測序,然而成百上千個樣本的全基因組測序其成本依然是巨大的,目前更加經濟有效的方案是GWAS芯片,針對特定人群,利用tag SNP的思想設計探針,覆蓋的SNP位點在幾十M的數量級。
相比全基因組測序,GWAS芯片確實更加經濟,但是其缺點也是顯而易見的,只能夠分析挖掘已知的SNP位點,而且位點數據量相對較少,要知道一個全基因組測序分析得到的SNP位點在幾百M左右。為了解決這個問題,科學家提出了基因型填充的思想。
genotype imputation,稱之為基因型填充,基本思想是利用單倍型來推斷芯片未覆蓋到的SNP位點的分型結果,在家系數據和獨立樣本的分析中都適用。家系樣本基因型填充的過程示意如下

部分樣本具有較為完整的SNP分型結果,依據這些樣本的分型結果構建在家系樣本中共享的單倍型,對應圖中方框標記的完整分型結果,針對基因型缺失的樣本,根據親緣關系推斷該樣本可能的單倍型,對于基因型缺失的位點,直接使用對應單倍型中的分型結果進行填充。
獨立樣本的基因型填充過程示意如下
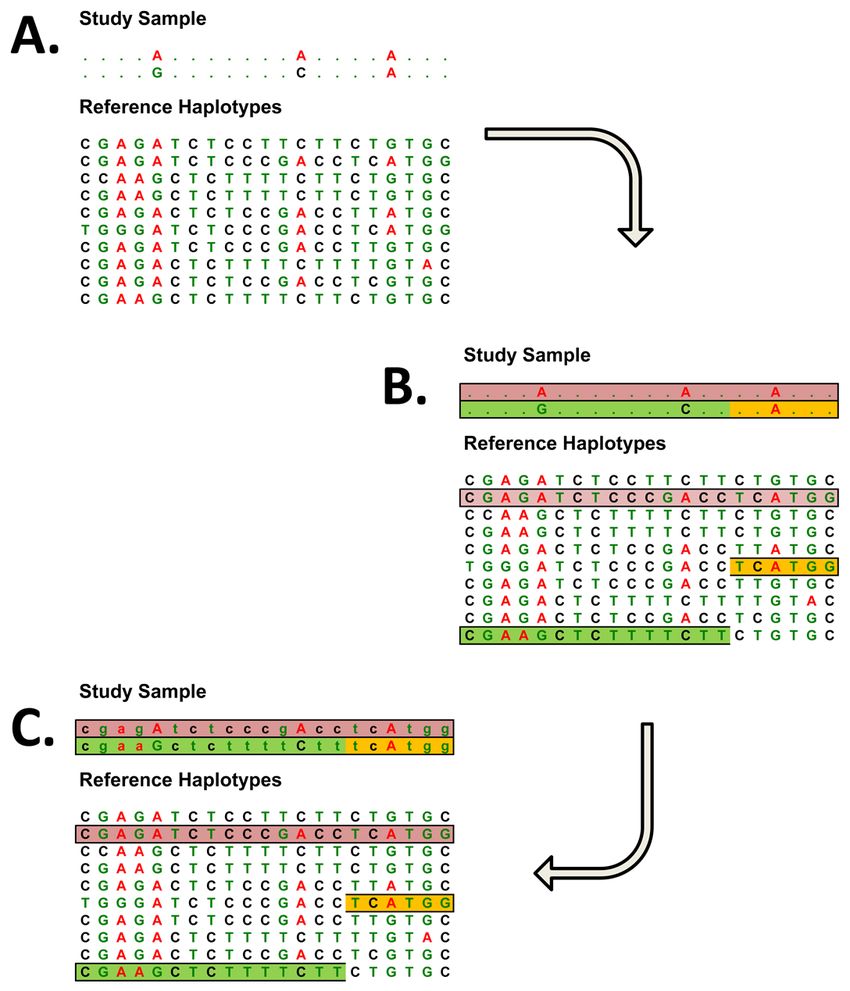
首先需要有一個參照的單倍型,根據樣本已有的分型結果,與參照的單倍型進行比較,確定其可能所屬的單倍型,然后進行填充。
以上示意圖來自下列文獻
Genotype Imputation
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2925172/
從以上示意圖可以看出,基因型填充有兩個必要條件,第一個條件就是參照的單倍型,對于獨立樣本,可以采用hapmap或者1000G等項目的單倍型作為參照,第二個條件就是已知分型結果位點的比例和分布,對于需要填充的樣本,要保證一定密度的分型結果,需要根據已有的分型結果來推斷該樣本可能的單倍型,分型結果已知的位點越多,其單倍型推斷的準確性越高,填充的準確性才會越高,根據這個條件來看,GWAS芯片最適合進行基因型填充,因為其覆蓋的SNP位點的數量和分布更有助于推測樣本的單倍型。
目前已經有很多用于基因型填充的軟件,部分列表如下
Beagle
IMPUTE2
MACH
關于怎么理解GWAS中的genotype imputation問題的解答就分享到這里了,希望以上內容可以對大家有一定的幫助,如果你還有很多疑惑沒有解開,可以關注億速云行業資訊頻道了解更多相關知識。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。