您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
小編給大家分享一下GWAS分析中如何使用PCA校正群體分層,相信大部分人都還不怎么了解,因此分享這篇文章給大家參考一下,希望大家閱讀完這篇文章后大有收獲,下面讓我們一起去了解一下吧!
GWAS通過分析case/control組之間的差異來尋找與疾病關聯的SNP位點,然而case和control兩組之間,可能本身就存在一定的差異,會影響關聯分析的檢測。
Population stratification,稱之為群體分層,是最常見的差異來源,指的是case/control組的樣本來自于不同的祖先群體,其分型結果自然是有差異的。GWAS分析的目的是尋找由于疾病導致的差異,其他的差異都屬于系統誤差,在進行分析時,需要進行校正。
對于群體分層的校正,通常采主成分分析的方法,即PCA, 對應的文章發表在nature genetics上,鏈接如下
https://www.nature.com/articles/ng1847

核心處理如下圖所示

對分型結果對應的矩陣進行PCA分析,該矩陣中行為SNP位點,列為樣本,分型結果為0,1,2。0表示沒有突變,1表示雜合突變,2表示純合突變。PCA分析之后,可以得到每個樣本在PC1,PC2等主成分軸上對應的位置。
PCA本質屬于排序分析,距離近的樣本擁有相似的屬性,根據PCA之后得到的位置信息,可以繪制如下所示的散點圖
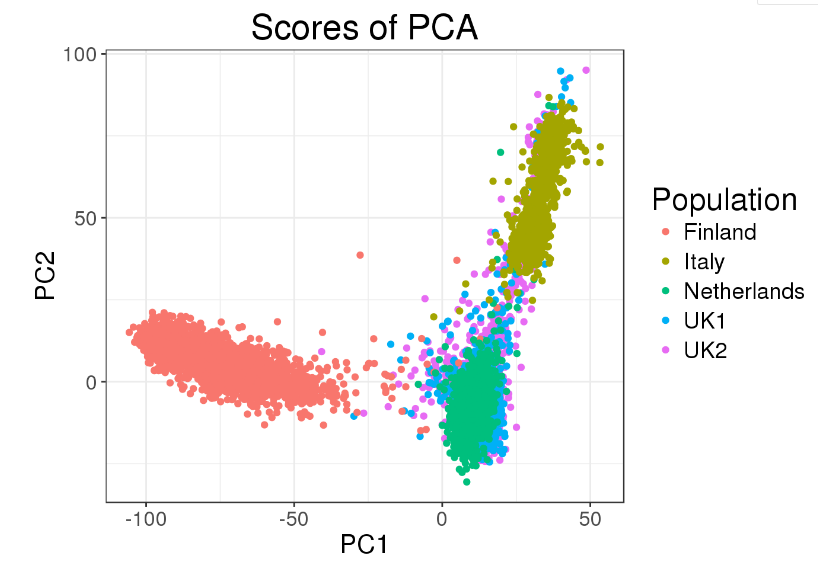
上圖中每個點代表一個樣本,繪圖使用的信息就是這些樣本在PC1和PC2兩個軸上的位置。這樣的散點圖可以直觀展示樣本的分層情況,對于顯著偏離總體的部分樣本,可以去除之后在重新進行分析。在后續進行GWAS分析時,這些PC軸上的位置信息可以作為回歸分析中的協變量,進行校正。
文章中將針對分型結果進行PCA分析的功能打包成了一個軟件,名字為EIGENSTRAT, github的網址如下
https://github.com/chrchang/eigensoft/tree/master/EIGENSTRAT
該軟件支持自動去除離群值樣本,顯示主成分的占比等很多功能,缺點就是執行速度比較慢。對于GWAS中的PCA而言,核心的信息其實就是樣本在各個主成分軸上的位置信息,我們需要這些信息來進行后續的校正。
面對GWAS規模的分型結果,運行速度是非常重要的一個因素。為此,實踐中常常采用以下兩款軟件
用法如下
plink \
--bfile sample \
--pca --out pca用法如下
gcta64 \
--bfile sample \
--make-grm \
--thread-num 5 \
--out gcta
gcta64 \
--grm gcta \
--pca 20 \
--thread-num 5 \
--out pca二者輸出結果雖然不是完全相同,但是分布的趨勢是一致的。不同之處在于,GCTA支持多線程,運行速度更快。輸出結果有多個文件,核心是一個后綴為eigenvec的文件,該文件保存了樣本在各個主成分軸上的位置信息,可以用于后續的校正。
這兩個軟件運行速度快,但是有個缺點就是不會輸出各個主成分的占比,如果想要這個信息,可以考慮類似功能的R包,比如vcfR,SNPRelate,bigsnpr等。
以上是“GWAS分析中如何使用PCA校正群體分層”這篇文章的所有內容,感謝各位的閱讀!相信大家都有了一定的了解,希望分享的內容對大家有所幫助,如果還想學習更多知識,歡迎關注億速云行業資訊頻道!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。