您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
TAD中的拓撲關聯結構域分析是怎樣的,相信很多沒有經驗的人對此束手無策,為此本文總結了問題出現的原因和解決方法,通過這篇文章希望你能解決這個問題。
利用更低分辨率的Hi-C基因組互作圖譜,科學家對染色質空間結構的了解不斷深入。主要介紹TAD這種結構,TAD全稱如下
Topologically Assocaited Domain
中文譯作拓撲關聯結構域,是一種首先在哺乳動物細胞中發現的染色質結構單元,對應的文章發表在nature上,文章標題如下
Topological Domains in Mammalian Genomes Identified by Analysis of Chromatin Interactions
pubmed的鏈接如下
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3356448/
將Hi-C基因組互作圖譜的分辨率提高到100kb以下,發現了互作圖譜中出現了一些self-interaction的區域,示意如下
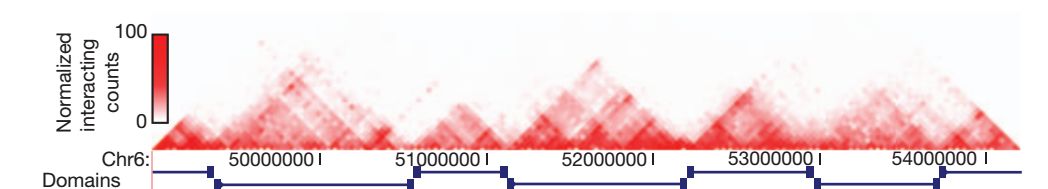
基因組互作圖譜本質是一個對稱矩陣,對角線兩側的信息是相等的。上圖中只取了原始方陣中對角線一側的信息,所以看上去是一個大的三角形,三角形的底邊對應的是原始方陣中的對角線部分。
在上圖中,互作強度由弱到強,單元格的顏色由白色過渡到紅色。可以看到,在底邊出重復出現了一些小的三角形區域,這些區域內部幾乎全部是紅色,說明這些區域內部的染色質片段間的互作頻率高,這樣的區域稱之為self-interaction區域,而相鄰的三角形區域間的互作頻率較低,如下圖所示

紅色三角形區域對應TAD內部區域的互作信息,而黑色區域對應TAD之間的互作信息。呈現到三角形的互作圖譜上,對應的就是底邊上有很多紅色的小三角形,而三角形對應的互作區域則都為白色,科學家將這種重復出現的內部互作頻率高,組間互作頻率低的domain定義為topologically assocaited domain, 簡稱TAD,對應下圖中的模型
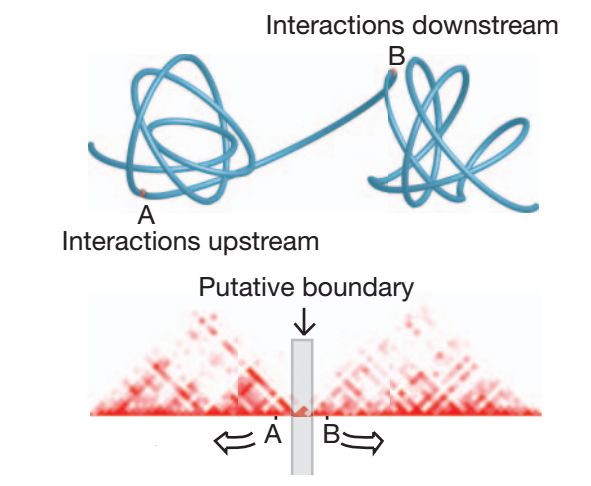
A和B對應兩個TAD, 在TAD之間存在了一個邊界,稱之為TAD doundary。為了準確地識別染色質中的TAD,定義了一種directionality index的統計量,簡稱DI,公式如下
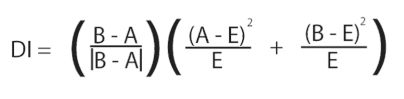
將分辨率降低到40kb,對于每個40kb的bin來說,A代表這個bin與上游2MB區域的互作reads, B代表這個bin與下游2MB區域的互作reads,E代表A和B的均值,采用類似卡方檢驗統計量的算法。空假設是這個bin與上游和下游的互作頻率相同。如果與上下游的互作頻率一致,則DI的值趨近于0。如下圖所示
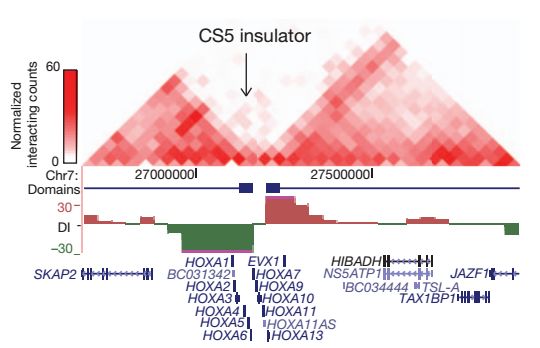
可以看到從TAD的開始到終止,DI的值會有一個從正值逐漸減小到0,然后變為負值,在不斷減小的情況。在TAD邊界處,DI的值突然趨近于0,因為邊界處與上下游的互作頻率幾乎相同,根據DI的這一分布規律,再結合隱馬爾可夫模型,最終在小鼠胚胎干細胞中識別到了2200多個TAD區域,長度的平均值為880kb。
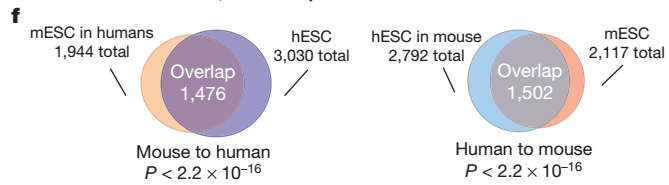
進一步分別對人和小鼠兩種不同細胞系中的TAD進行識別和分析,發現TAD在不同細胞或者組織中相對穩定,在不同物種間也具有一定的保守性,結果如下圖所示

為了進一步探究TAD在染色質上的分布特征,科學家分析了TAD邊界內各種mark的分布情況。首先是CTCF,結果如下所示
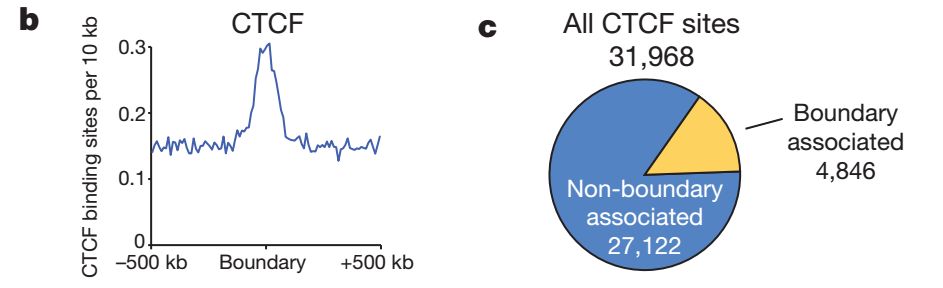
發現在TAD邊界處存在CTCF的富集,但并不是說所有的CTCF都集中出現在TAD邊界處,所以進一步由探究了其他mark,包括各種組蛋白修飾等的分布,結果如下
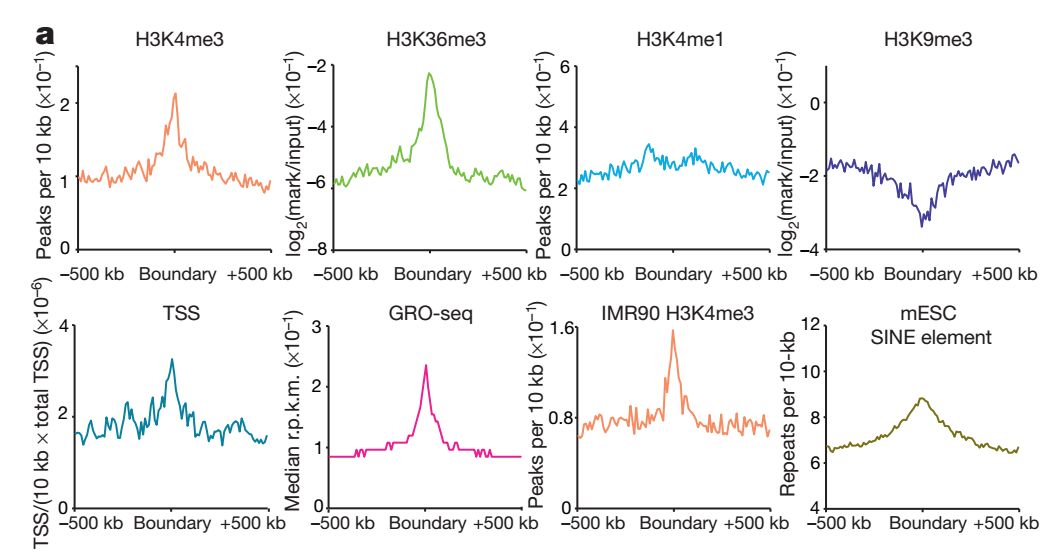
發現H3K4me3,H3K36me3, TSS, SINE重復元件等都有富集。進一步分析了基因的分布情況,結果如下
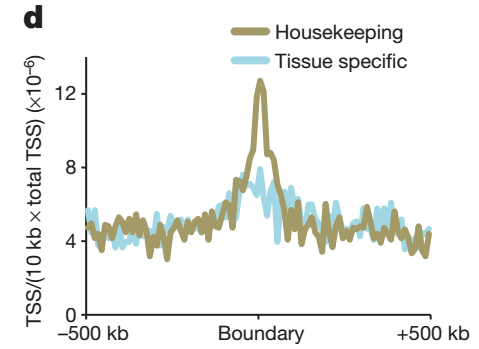
發現管家基因在TAD邊界處存在富集。
通過40kb分辨率的Hi-C互作圖譜,鑒定到了TAD這種在哺乳動物中存在的相對穩定,且具有一定進化保守性的染色質結構單元。
看完上述內容,你們掌握TAD中的拓撲關聯結構域分析是怎樣的的方法了嗎?如果還想學到更多技能或想了解更多相關內容,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。