您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容介紹了“如何利用bedtools預測chip_seq數據的靶基因”的有關知識,在實際案例的操作過程中,不少人都會遇到這樣的困境,接下來就讓小編帶領大家學習一下如何處理這些情況吧!希望大家仔細閱讀,能夠學有所成!
通常在分析peak區域對應的靶基因時,會選取轉錄起始位點TSS上下游一定長度的區域作為候選的靶基因范圍,本文介紹下如何利用bedtools來對peak與TSS區域的overlap情況進行分析,從而得到靶基因,可以分為以下幾步
以hg38為例,通過UCSC的FTP服務可以得到物種對應的refFlat文件,鏈接如下
http://hgdownload.soe.ucsc.edu/goldenPath/hg38/database/

refFLat和refGene這兩個文件記錄的信息相同,refFlat文件列數更少,這里我們選擇下載refFlat.txt.gz, 該文件的內容如下所示
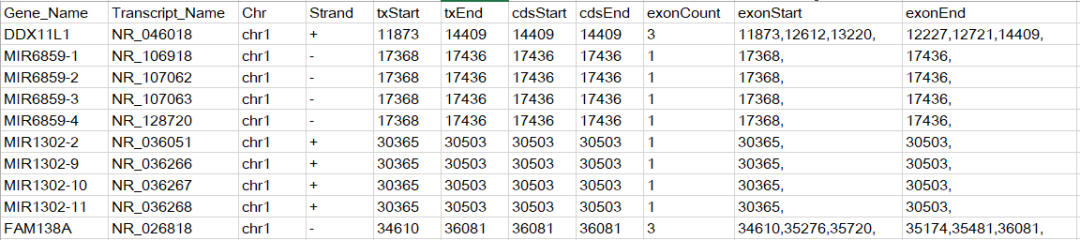
在原始文件中是沒有第一行的標題的,我手動添加的標題是為了方便描述每列的含義,從該文件中可以得到TSS位點信息。
bedtools要求輸入的文件格式為bed, gff, vcf等,這里我們需要把上述下載的原始文件轉換為bed格式,用法如下
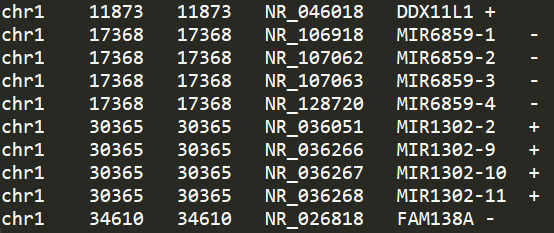
awk '{print $3"\t"$5"\t"$5"\t"$2"\t"$1"\t"$4}' > hg38.tss.bed內容如下所示
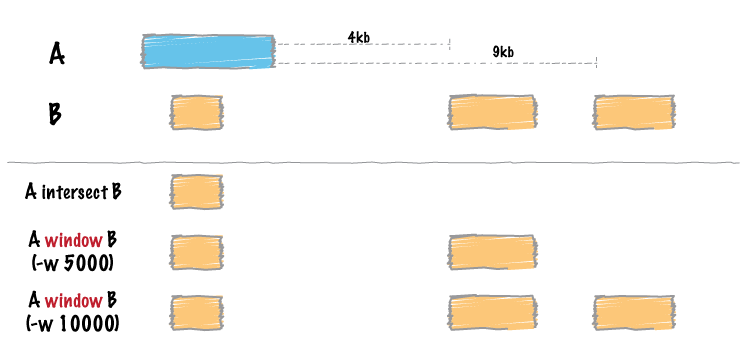
bedtools windows和intersect的功能類似,都是用于求兩個區間A和B的交集,只不過window會在A區間的上下游加上一個可以自定義的長度之后,再與B區間求交集,原理示意如下
以TSS上下游5kb為例,用法如下
bedtools window -a hg39.tss.bed -b peak.bed -w 5000 -sm > overlap.txt
通過window這個命令,可以靈活的定義TSS上下游的區間,快速得到peak對應的靶基因。
“如何利用bedtools預測chip_seq數據的靶基因”的內容就介紹到這里了,感謝大家的閱讀。如果想了解更多行業相關的知識可以關注億速云網站,小編將為大家輸出更多高質量的實用文章!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。