您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
小編給大家分享一下LncBook數據庫有什么用,希望大家閱讀完這篇文章之后都有所收獲,下面讓我們一起去探討吧!
隨著lncRNA研究的發展,lncRNA的數量越來越多,但是擁有功能注釋的lncRNA只占了其中很小一部分。為了更好的開展lncRNA的功能研究,科學家收集文獻中人類lncRNA相關的數據,包括了表達量,相關疾病,甲基化位點,SNP位點,功能描述等信息,并整理成了數據庫LncBook, 網址如下
http://bigd.big.ac.cn/lncbook/index
該數據庫中包含以下8種lncRNA相關信息
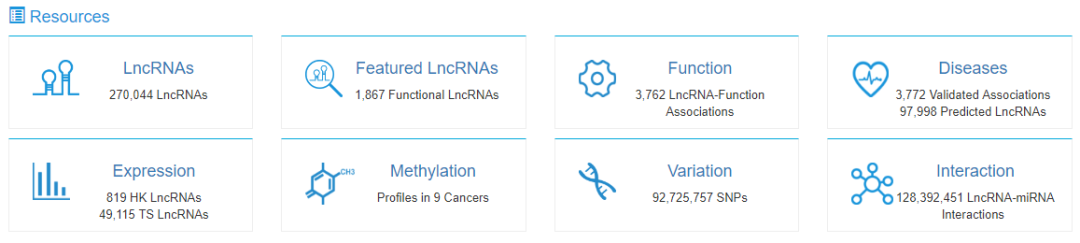
共包含277044個lncRNA, 既有實驗驗證的lncRNA,也有軟件預測的lncRNA,收集了來自Gencode, RefSeq, Noncode, Lncipedia, MiTranscriptome 等數據庫中的lncRNA, 在預測lncRNA時,采用了CPAT, PLEK, LGC3個軟件結果的交集。
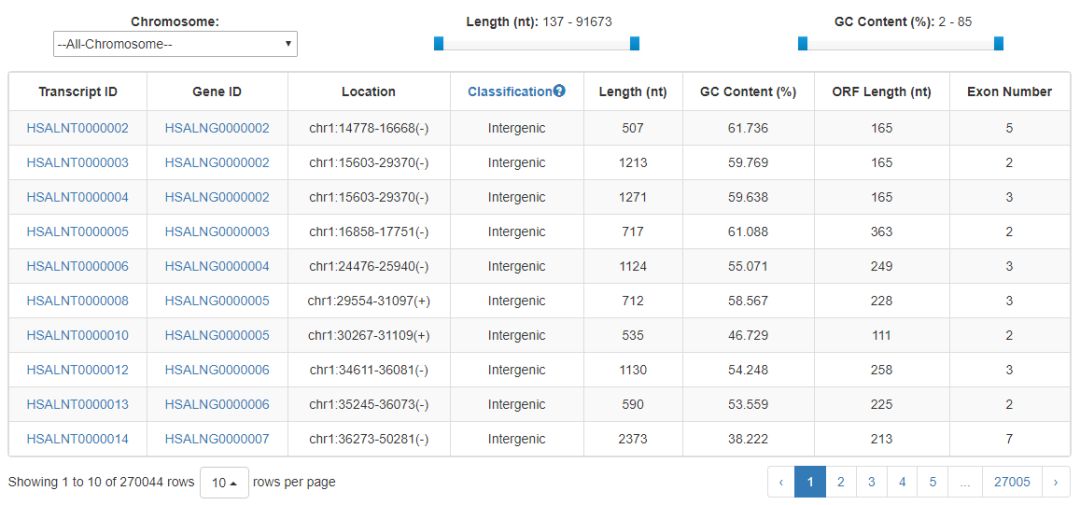
這部分提供lncRNA的ID, 染色體位置,長度,外顯子個數,類型等基本信息,示意如下
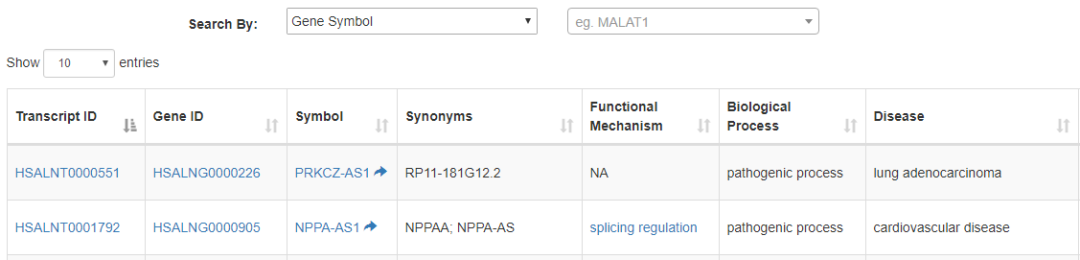
這部分只包含來自lncRNAWiki數據庫中的有功能注釋和文獻支持的lncRNA, 結果示意如下
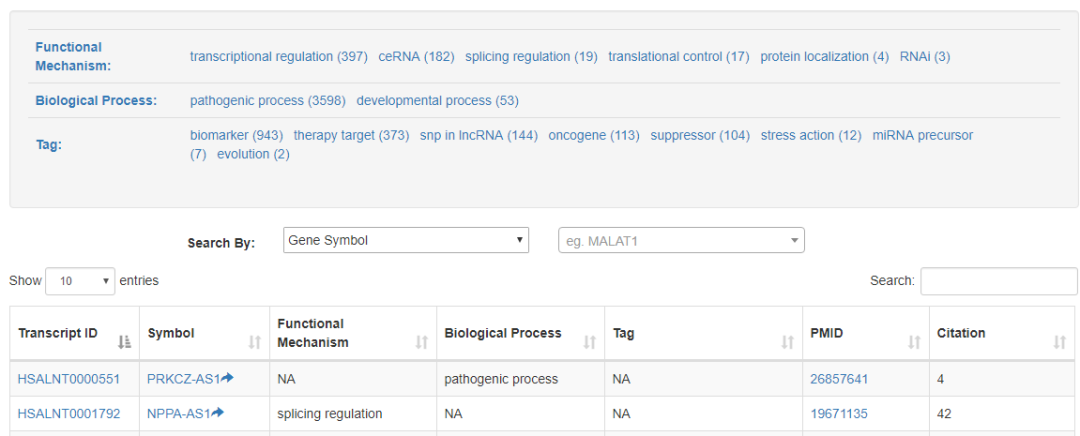
這部分給出lncRNA的生物學功能注釋和參與的生物學過程,共包含以下5種類別的功能
transcriptional regulation
ceRNA
splicing regulation
protein localization
RNAi
示意如下
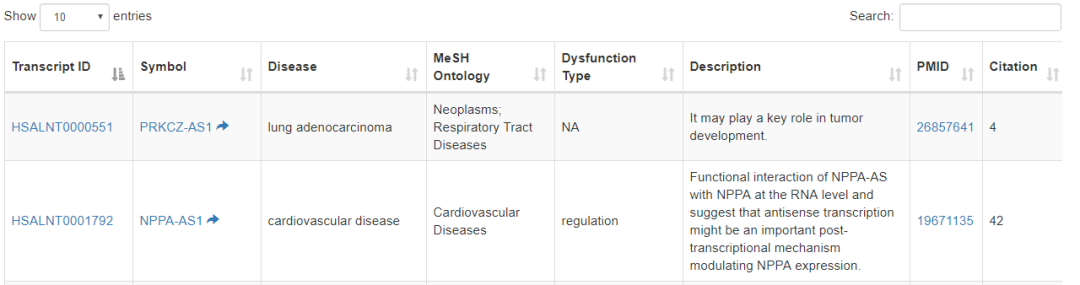
這部分給出lncRNA相關的疾病信息,包括了實驗驗證和預測兩種,實驗驗證的數據直接從lncRNADisease和lncRNAwiki這兩個數據庫得到,預測主要是結合了其他組學的數據,包括以下3種策略
相比正常樣本,如果在腫瘤樣本中某個lncRNA啟動子區存在高甲基化區域,則認為該lncRNA與疾病相關;
如果某個lncRNA區域存在于疾病相關的SNP位點,則該lncRNA與疾病相關;
如果某個lncRNA與5個以上與疾病相關miRNA(miRNA相關疾病信息來自HMDD數據庫)具有相互作用,則該lncRNA與疾病相關;
結果示意如下
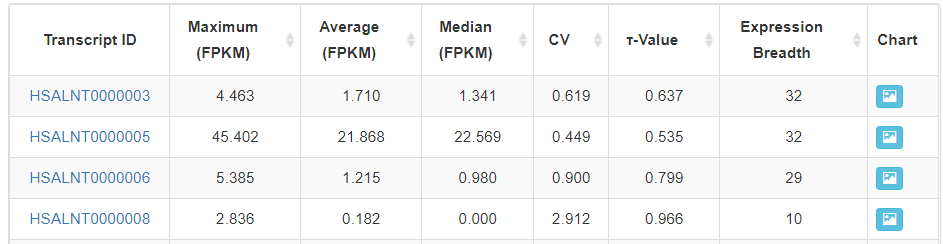
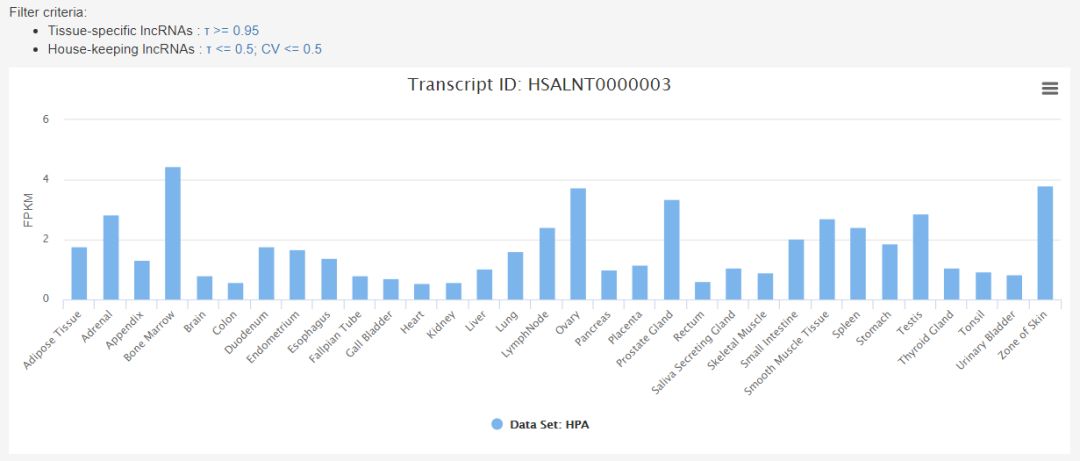
通過分析HPA和GTEx兩個公共項目的轉錄組數據,給出lncRNA在各個組織中的FPKM表達量值,示意如下
通過分析在不同組織中的表達量,可以用于判斷組織特異性lncRNA還是管家lncRNA, 計算公式如下
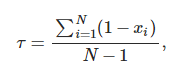
N代表組織的個數,x代表每個組織中歸一化之后的表達量,這里的歸一化是將每個組織中的表達量除以所有組織中最大的表達量。
通過分析TCGA和ENCODE數據庫的數據,給出lncRNA相關的甲基化信息,示意如下
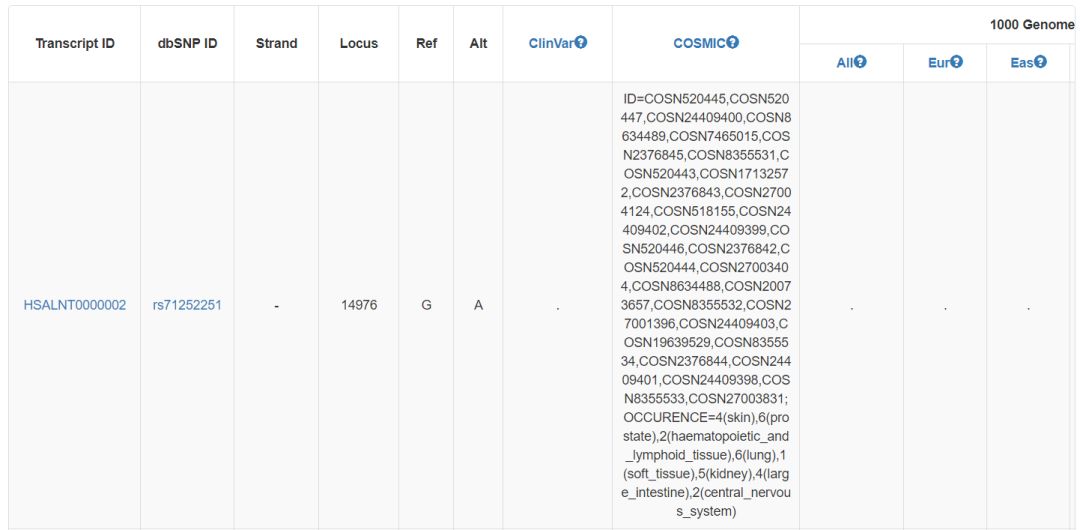
將dbSNP數據庫中的SNP位點映射到lncRNA上,同時提供了來自COSMIC和ClinVar數據庫的注釋信息,以及1000G中的頻率信息,結果示意如下
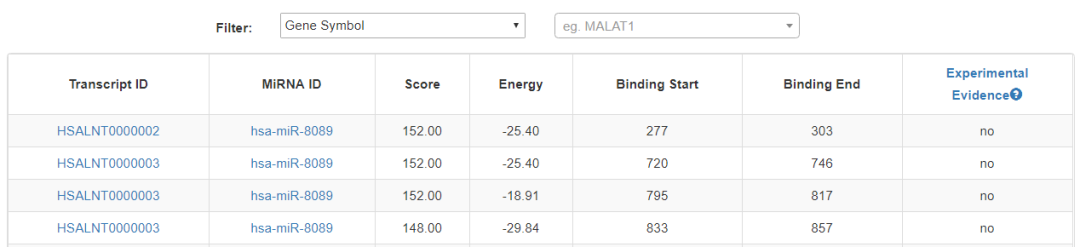
采用tagetScan和miRanda兩款軟件來預測lncRNA與miRNA的相互作用,取交集作為最終的結果,實驗證據主要來自于starbase數據庫,結果示意如下
除此之外,還提供了一下4種工具
Blast
LGC
Classification
Conversion
Blast用于將輸入序列和數據庫中的lncRNA序列進行比對,LGC用于分析序列的蛋白編碼潛能,預測lncRNA;Classification用于根據染色體位置對基因進行分類,Conversion用于提供多個lncRNA數據庫中ID的轉換,結果示意如下

lncBook和lncRNAwiki是同一個開發團隊,lncBook可以看做lncRNAwiki的升級版本。
看完了這篇文章,相信你對“LncBook數據庫有什么用”有了一定的了解,如果想了解更多相關知識,歡迎關注億速云行業資訊頻道,感謝各位的閱讀!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。