您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關eggNOG數據庫有什么用,小編覺得挺實用的,因此分享給大家做個參考,希望大家閱讀完這篇文章后可以有所收獲。
直系同源蛋白的預測在系統發育,比較基因組學等多個領域都占用重要地位,COG數據庫開創了同源蛋白數據庫的先河,后續又不斷有新的數據庫涌現,而eggNOG就是目前使用最廣泛的數據庫之一。
官網如下
http://eggnogdb.embl.de/#/app/home
eggNOG全稱evolutionary genealogy of genes:Non-supervised Orthologous, 在COG算法的基礎上進行了拓展和延伸,采用基于圖狀結構的非監督聚類算法,構建了真核,原核,病毒等不同物種的同源蛋白簇。
該數據庫最新版本為eggNOG 4.5.1, 涵蓋了2031種真核和原核生物,352種病毒,構建了19萬個同源蛋白簇。
和COG類似,eggNOG對于orthology group的功能也進行了分類整理,每個類別用一個字母表示,在以下鏈接可以查看具體的分類信息
http://eggnogdb.embl.de/download/eggnog_4.5/COG_functional_categories.txt
COG數據庫中提供了26種分類,eggNOG中提供了25種分類,缺少了如下類別
X Mobilome: prophages, transposons
其他的分類和COG數據庫完全一致。在官網的搜索框中可以對OG編號進行檢索

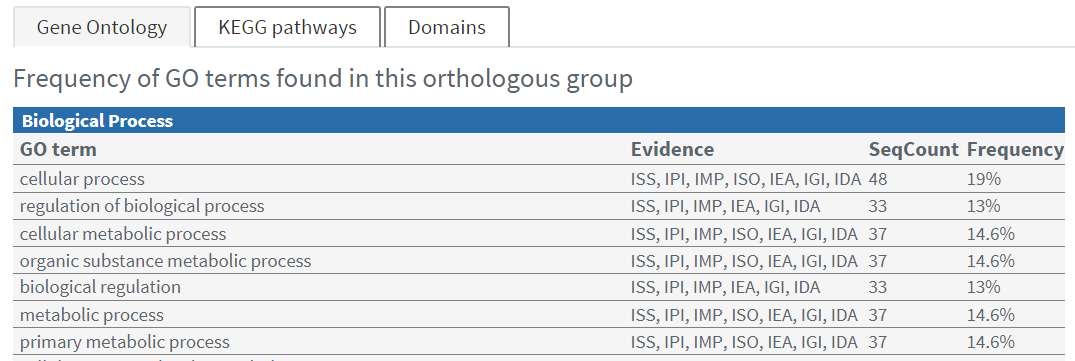
COG5157的檢索結果如下

在檢索結果中,可以查看該orthology group下包含的蛋白序列和物種信息,也可以下載對應的序列。除此之外,還提供了GO, KEGG, pfam, SMRT等相關的功能注釋信息。
除了在線檢索外,官方還提供了API服務,方便程序抓取對應數據,示例如下
http://eggnogapi.embl.de/nog_data/text/fasta/ENOG410ZSWV
通過以上API鏈接,可以獲得ENOG410ZSWV這個orthology group下的所有蛋白序列,更多用法請參考官方文檔。
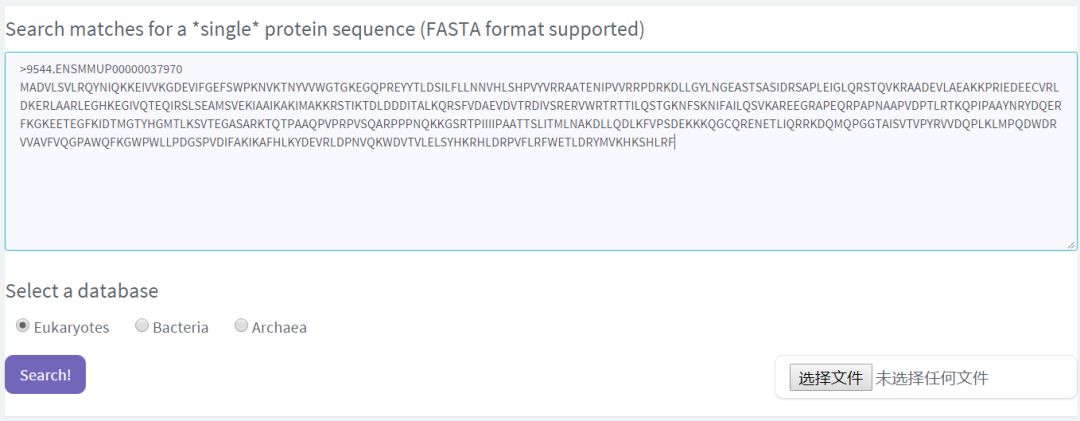
官網還提供了以下兩種注釋服務
一次只支持一條序列的查詢,輸入fasta格式的蛋白序列,選擇對應物種即可
支持多條序列的NOG注釋,提供了在線服務,也可以下載軟件到本地運行。
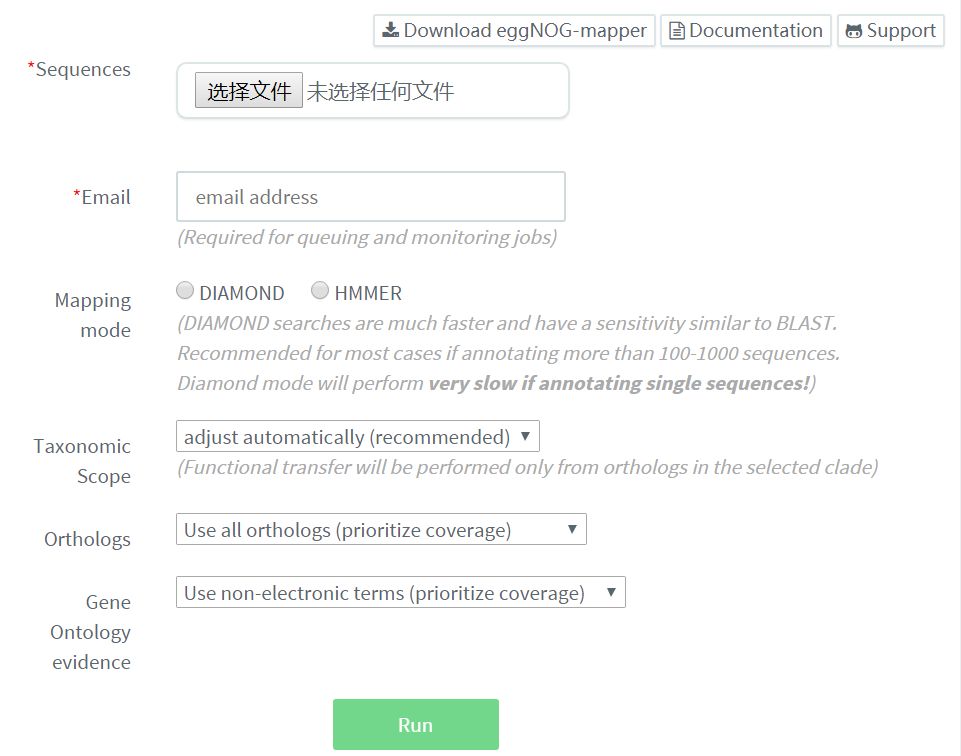
支持diamond和hmmer兩種比對軟件,當序列條數少于1000條時,選擇hmmer,當序列多余1000條時,選擇diamond, 速度更快。本地版的eggNOG-mapper 的安裝和使用可以參考如下文檔
https://github.com/jhcepas/eggnog-mapper/wiki
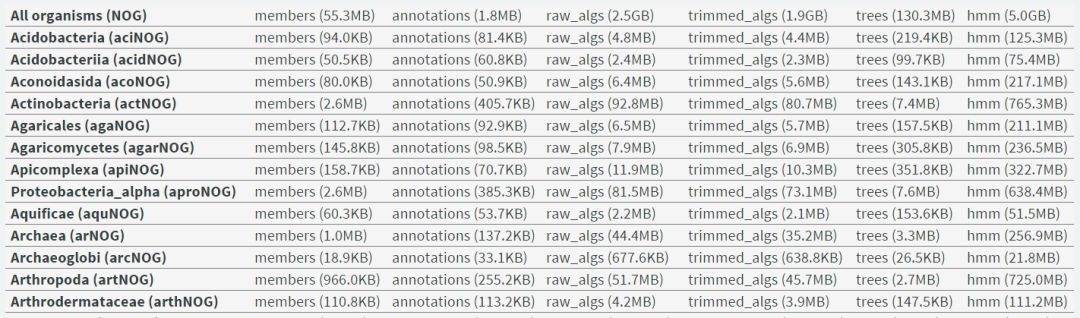
官網提供了下載服務,可以下載完整數據庫,也可以根據物種挑選合適的子數據庫進行下載,示意如下
通過eggNOG數據庫,我們可以對新的蛋白序列進行同源蛋白的注釋, 挖掘其功能。
關于“eggNOG數據庫有什么用”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,使各位可以學到更多知識,如果覺得文章不錯,請把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。