您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇“python遺傳算法之geatpy的怎么安裝使用”文章的知識點大部分人都不太理解,所以小編給大家總結了以下內容,內容詳細,步驟清晰,具有一定的借鑒價值,希望大家閱讀完這篇文章能有所收獲,下面我們一起來看看這篇“python遺傳算法之geatpy的怎么安裝使用”文章吧。
首先是安裝geatpy,使用pip3命令進行安裝即可:
pip3 install geatpy
出現如下提示即安裝成功:
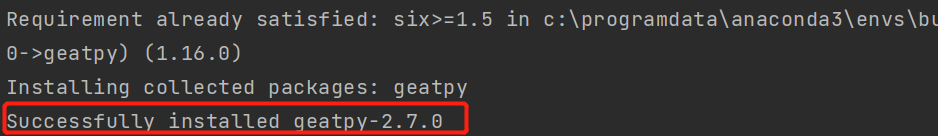
geatpy中的大部分數據都是都是使用numpy的數組進行存儲和計算的,下面我將介紹遺傳算法中的概念是如何用numpy數據表示,以及行和列的含義。
遺傳算法中最重要的就是個體的染色體表示,在geatpy中種群染色體用Chrom表示,這是一個二維數組,其中每一行對應一個個體的染色體編碼,Chrom的結構如下:其中lind表示編碼的長度,Nind表示的是種群的規模(個體數量)。

種群表現型是是指種群染色體矩陣Chrom經過解碼后得到的基因表現型矩陣Phen,每一行對應一個個體,每一列對應一個決策變量,Phen的結構如下:其中Nvar表示變量的個數
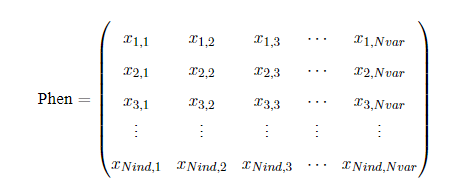
Phen的值與采用的解碼方式有關。Geatpy提供二進制/格雷碼編碼轉十進制整數或實數的解碼方式。另外,在Geatpy也可以使用不需要解碼的“實值編碼”種群,這種種群的染色體的每一位就對應著決策變量的實際值,即這種編碼方式下Phen等價Chrom。
Geatpy采用numpy的array類型矩陣來存儲種群的目標函數值。一般命名為ObjV,每一行對應每一個個體,因此它擁有與Chrom相同的行數;每一列對應一個目標函數,因此對于單目標函數,ObjV會只有1列;而對于多目標函數,ObjV會有多列, ObjV的表示形式如下:
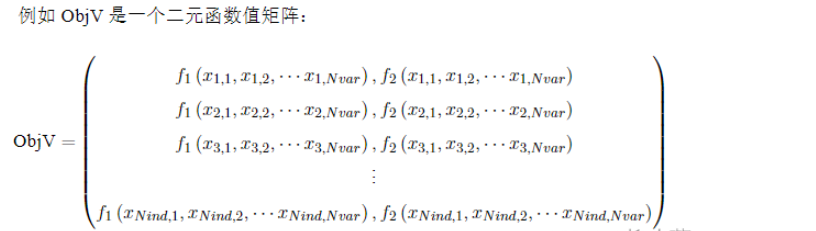
Geatpy采用列向量來存儲種群個體適應度(適應度函數計算而來)。一般命名為FitnV,它同樣是numpy的array類型,每一行對應種群矩陣的每一個個體。因此它擁有與Chrom相同的行數,FitnV的格式如下:

注意:Geatpy中的適應度遵循“最小適應度為0”的約定。
Geatpy采用numpy的array類型的矩陣CV(Constraint Violation Value)來存儲種群個體違反各個約束條件的程度。命名為CV,它的每一行對應種群的每一個個體,因此它擁有與Chrom相同的行數;每一列對應一個約束條件,因此若有一個約束條件,那么CV矩陣就會只有一列,如有多個約束條件,CV矩陣就會有多列。如果設有num個約束,則CV矩陣的結構如下圖所示:

CV矩陣的某個元素若小于或等于0,則表示該元素對應的個體滿足對應的約束條件。若大于0,則表示違反約束條件,在大于0的條件下值越大,該個體違反該約束的程度就越高。Geatpy提供兩種處理約束條件的方法,一種是罰函數法,另一種是可行性法則。在使用可行性法則處理約束條件時,需要用到CV矩陣。
所謂的譯碼矩陣,只是用來描述種群染色體特征的矩陣,如染色體中的每一位元素所表達的決策變量的范圍、是否包含范圍的邊界、采用二進制還是格雷碼、是否使用對數刻度、染色體解碼后所代表的決策變量的是連續型變量還是離散型變量等等。
在只使用工具箱的庫函數而不使用Geatpy提供的面向對象的進化算法框架時,譯碼矩陣可以單獨使用。若采用Geatpy提供的面向對象的進化算法框架時,譯碼矩陣可以與一個存儲著種群染色體編碼方式的字符串Encoding來配合使用。
目前Geatpy中有三種Encoding,分別為:
BG:(二進制/格雷碼)
RI:((實整數編碼,即實數和整數的混合編碼)
P:(排列編碼,即染色體每一位的元素都是互異)
注:’RI’和’P’編碼的染色體都不需要解碼,染色體上的每一位本身就代表著決策變量的真實值,因此“實整數編碼”和“排列編碼”可統稱為“實值編碼”
以BG編碼為例,我們展示一下編譯矩陣FieldD。FieldD的結構如下:
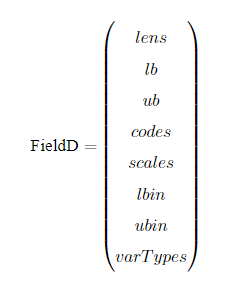
其中,lens,lb,ub,codes,scales,lbin,ubin,varTypes都是行向量,其長度等于決策變量的個數。
lens:代表以條染色體中,每個子染色體的長度。
lb:代表每個變量的上界
ub:代表每個變量的下界
codes:代表染色體字串用的編碼方式,[1,0,1]代表第一個變量用的格雷編碼,第二個變量用的二進制編碼,第3個變量用的格雷編碼。
scales:指明每個子串用的是算術刻度還是對數刻度。scales[i] = 0為算術刻度,scales[i] = 1為對數刻度(對數刻度很少用,可以忽略。)
lbin:代表變量上界是否包含其范圍邊界。0代表不包含,1代表包含。‘[ ’和 ‘(’ 的區別
ubin:代表變量下界是否包含其范圍邊界。0代表不包含,1代表包含。
varTypes:代表決策變量的類型,元素為0表示對應位置的決策變量是連續型變量;1表示對應的是離散型變量。
例如:有以下一個譯碼矩陣

它表示待解碼的種群染色體矩陣Chrom解碼后可以表示成3個決策變量,每個決策變量的取值范圍分別是[1,10], [2,9], [3,15]。其中第一第二個變量采用的是二進制編碼,第三個變量采用的是格雷編碼,且第一、第三個決策變量為連續型變量;第二個為離散型變量。
#通過種群染色體chrom和譯碼矩陣FieldD,可解碼成種群表現型矩陣。 import geatpy as ea Phen = ea.bs2ri(Chrom, FieldD)
在使用Geatpy進行進化算法編程時,常常建立一個進化追蹤器(如pop_trace)來記錄種群在進化的過程中各代的最優個體,尤其是采用無精英保留機制時,進化追蹤器幫助我們記錄種群在進化過程中的最優個體。待進化完成后,再從進化追蹤器中挑選出“歷史最優”的個體。這種進化記錄器有多種,其中一種是numpy的array類型的,結構如下:其中MAXGEN是種群進化的代數(迭代次數)。
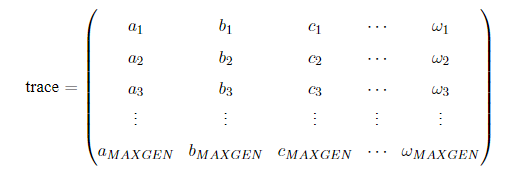
trace的每一列代表不同的指標,比如第一列記錄各代種群的最佳目標函數值,第二列記錄各代種群的平均目標函數值…trace的每一行對應每一代,如第一行代表第一代,第二行代表第二代…另外一種進化記錄器是一個列表,列表中的每一個元素都是一個擁有相同數據類型的數據。比如在Geatpy的面向對象進化算法框架中的pop_trace,它是一個列表,列表中的每一個元素都是歷代的種群對象。
在Geatpy提供的面向對象進化算法框架中,種群類(Population)是一個存儲著與種群個體相關信息的類。它有以下基本屬性:
sizes : int -種群規模,即種群的個體數目。
ChromNum : int -染色體的數目,即每個個體有多少條染色體。
Encoding : str -染色體編碼方式。
Field : array -譯碼矩陣,可以是FieldD或FieldDR。
Chrom : array -種群染色體矩陣,每一行對應一個個體的一條染色體。
Lind : int -種群染色體長度。
ObjV : array -種群目標函數值矩陣。
FitnV : array -種群個體適應度列向量。
CV : array -種群個體違反約束條件程度的矩陣。
Phen : array -種群表現型矩陣。
可以直接對種群對象進行提取個體、個體合并等操作,比如pop1和pop2是兩個種群對象,則通過語句“pop3 = pop1 + pop2”,即可把兩個種群的個體合并,得到一個新的種群。在合并的過程中,實際上是把種群的各個屬性進行合并,然后用合并的數據來生成一個新的種群(詳見Population.py)。又比如執行語句“pop3 = pop1[[0]]”,可以把種群的第0號個體抽取出來,得到一個新的只有一個個體的種群對象pop3。值得注意的是,種群的這種個體抽取操作要求下標必須為列表或是Numpy array類型的行向量,不能是標量(詳見Population.py)
PsyPopulation類是Population的子類,它提供Population類所不支持的多染色體混合編碼。它有以下基本屬性:
sizes : int -種群規模,即種群的個體數目。
ChromNum : int -染色體的數目,即每個個體有多少條染色體。
Encodings : list -存儲各染色體編碼方式的列表。
Fields : list -存儲各染色體對應的譯碼矩陣的列表。
Chroms : list -存儲種群各染色體矩陣的列表。
Linds : list -存儲種群各染色體長度的列表。
ObjV : array -種群目標函數值矩陣。
FitnV : array -種群個體適應度列向量。
CV : array -種群個體違反約束條件程度的矩陣。
Phen : array -種群表現型矩陣。
可見PsyPopulation類基本與Population類一樣,不同之處是采用Linds、Encodings、Fields和Chroms分別存儲多個Lind、Encoding、Field和Chrom。
PsyPopulation類的對象往往與帶“psy”字樣的進化算法模板配合使用,以實現多染色體混合編碼的進化優化。
遺傳算法求解以下函數的最小值:
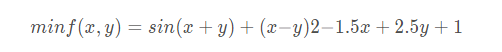
代碼實現:
#-*-coding:utf-8-*-
import numpy as np
import geatpy as ea#導入geatpy庫
import time
"""============================目標函數============================"""
def aim(Phen):#傳入種群染色體矩陣解碼后的基因表現型矩陣
x1 = Phen[:, [0]]#取出第一列,得到所有個體的第一個自變量
x2 = Phen[:, [1]]#取出第二列,得到所有個體的第二個自變量
return np.sin(x1 + x2) + (x1 - x2) ** 2 - 1.5 * x1 + 2.5 * x2+1
"""============================變量設置============================"""
x1 = [-1.5, 4]#第一個決策變量范圍
x2 = [-3, 4]#第二個決策變量范圍
b1 = [1, 1]#第一個決策變量邊界,1表示包含范圍的邊界,0表示不包含
b2 = [1, 1]#第二個決策變量邊界,1表示包含范圍的邊界,0表示不包含
#生成自變量的范圍矩陣,使得第一行為所有決策變量的下界,第二行為上界
ranges=np.vstack([x1, x2]).T
#生成自變量的邊界矩陣
borders=np.vstack([b1, b2]).T
varTypes = np.array([0, 0])#決策變量的類型,0表示連續,1表示離散
"""==========================染色體編碼設置========================="""
Encoding ='BG'#'BG'表示采用二進制/格雷編碼
codes = [1, 1]#決策變量的編碼方式,兩個1表示變量均使用格雷編碼
precisions =[6, 6]#決策變量的編碼精度,表示解碼后能表示的決策變量的精度可達到小數點后6位
scales = [0, 0]#0表示采用算術刻度,1表示采用對數刻度#調用函數創建譯碼矩陣
FieldD =ea.crtfld(Encoding,varTypes,ranges,borders,precisions,codes,scales)
"""=========================遺傳算法參數設置========================"""
NIND = 20#種群個體數目
MAXGEN = 100#最大遺傳代數
maxormins = np.array([1])#表示目標函數是最小化,元素為-1則表示對應的目標函數是最大化
selectStyle ='sus'#采用隨機抽樣選擇
recStyle ='xovdp'#采用兩點交叉
mutStyle ='mutbin'#采用二進制染色體的變異算子
Lind =int(np.sum(FieldD[0, :]))#計算染色體長度
pc= 0.9#交叉概率
pm= 1/Lind#變異概率
obj_trace = np.zeros((MAXGEN, 2))#定義目標函數值記錄器
var_trace = np.zeros((MAXGEN, Lind))#染色體記錄器,記錄歷代最優個體的染色體
"""=========================開始遺傳算法進化========================"""
start_time = time.time()#開始計時
Chrom = ea.crtpc(Encoding,NIND, FieldD)#生成種群染色體矩陣
variable = ea.bs2ri(Chrom, FieldD)#對初始種群進行解碼
ObjV = aim(variable)#計算初始種群個體的目標函數值
best_ind = np.argmin(ObjV)#計算當代最優個體的序號
#開始進化
for gen in range(MAXGEN):
FitnV = ea.ranking(maxormins * ObjV)#根據目標函數大小分配適應度值
SelCh = Chrom[ea.selecting(selectStyle,FitnV,NIND-1),:]#選擇
SelCh = ea.recombin(recStyle, SelCh, pc)#重組
SelCh = ea.mutate(mutStyle, Encoding, SelCh, pm)#變異
# #把父代精英個體與子代的染色體進行合并,得到新一代種群
Chrom = np.vstack([Chrom[best_ind, :], SelCh])
Phen = ea.bs2ri(Chrom, FieldD)#對種群進行解碼(二進制轉十進制)
ObjV = aim(Phen)#求種群個體的目標函數值
#記錄
best_ind = np.argmin(ObjV)#計算當代最優個體的序號
obj_trace[gen,0]=np.sum(ObjV)/ObjV.shape[0]#記錄當代種群的目標函數均值
obj_trace[gen,1]=ObjV[best_ind]#記錄當代種群最優個體目標函數值
var_trace[gen,:]=Chrom[best_ind,:]#記錄當代種群最優個體的染色體
# 進化完成
end_time = time.time()#結束計時
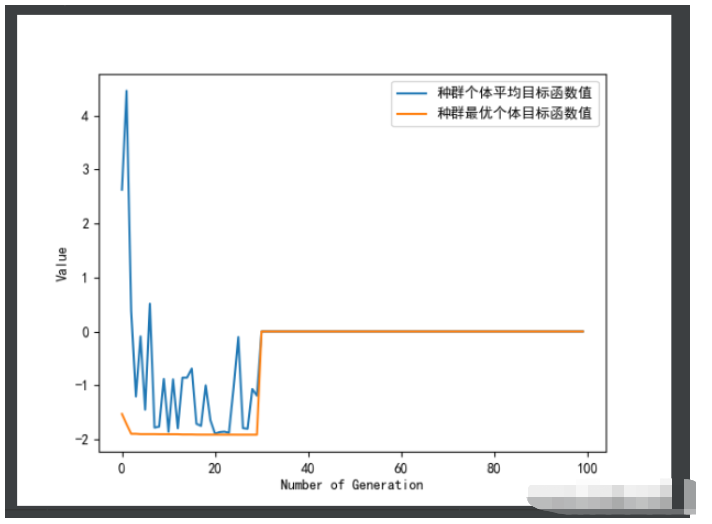
ea.trcplot(obj_trace, [['種群個體平均目標函數值','種群最優個體目標函數值']])#繪制圖像
"""============================輸出結果============================"""
best_gen = np.argmin(obj_trace[:, [1]])
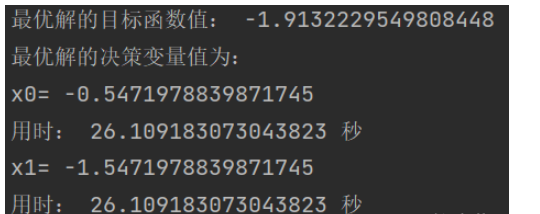
print('最優解的目標函數值:', obj_trace[best_gen, 1])
variable = ea.bs2ri(var_trace[[best_gen], :], FieldD)#解碼得到表現型(即對應的決策變量值)
print('最優解的決策變量值為:')
for i in range(variable.shape[1]):
print('x'+str(i)+'=',variable[0, i])
print('用時:', end_time - start_time,'秒')效果圖:
結果如下:
以上就是關于“python遺傳算法之geatpy的怎么安裝使用”這篇文章的內容,相信大家都有了一定的了解,希望小編分享的內容對大家有幫助,若想了解更多相關的知識內容,請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。