您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
Geatpy是一個高性能實用型的Python遺傳算法工具箱,提供一個面向對象的進化算法框架,經過全面改版后,新版Geatpy2目前由華南農業大學、暨南大學、華南理工等本碩博學生聯合團隊開發及維護。
Website (including documentation): http://www.geatpy.com
Demo : https://github.com/geatpy-dev/geatpy/tree/master/geatpy/demo
Pypi page : https://pypi.org/project/geatpy/
Contact us: http://geatpy.com/index.php/about/
Bug reports: https://github.com/geatpy-dev/geatpy/issues
Notice: http://geatpy.com/index.php/notice/
FAQ: http://geatpy.com/index.php/faq/
Geatpy提供了許多已實現的遺傳和進化算法相關算子的庫函數,如初始化種群、選擇、交叉、變異、重插入、多目標優化非支配排序等,并且提供諸多已實現的進化算法模板來實現多樣化的進化算法。其執行效率高于Matlab、Java和Python編寫的一些知名工具箱、平臺或框架等,學習成本低、模塊高度脫耦、擴展性高。
Geatpy支持二進制/格雷碼編碼種群、實數值種群、整數值種群、排列編碼種群。支持×××賭選擇、隨機抽樣選擇、錦標賽選擇。提供單點交叉、兩點交叉、洗牌交叉、部分匹配交叉(PMX)、順序交叉(OX)、線性重組、離散重組、中間重組等重組算子。提供簡單離散變異、實數值變異、整數值變異、互換變異等變異算子。支持隨機重插入、精英重插入。支持awGA、rwGA、nsga2、快速非支配排序等多目標優化的庫函數、提供進化算法框架下的常用進化算法模板等。
關于遺傳算法、進化算法的學習資料,在官網中https://www.geatpy.com 有詳細講解以及相關的學術論文鏈接。同時網上也有很多資料。
閑話少說……下面講一下怎么安裝和使用:
先說一下安裝方法:
首先是要windows系統,Python要是3.5,3.6或3.7版本 ,并且安裝了pip。只需在控制臺執行
pip install geatpy
即可安裝成功。或者到github上下載源碼進行編譯安裝:https://github.com/geatpy-dev/geatpy 。推薦是直接用pip的方式安裝。因為這樣方便后續的更新。我為了方便運行demo代碼以及查看源碼和官方教程文檔,因此另外在github上也下載了(但仍用pip方式安裝)。
有些初學Python的讀者反映還是不知道怎么安裝,或者安裝之后不知道怎么寫代碼。這里推薦安裝Anaconda,它集成了Python的許多常用的運行庫,比如Numpy、Scipy等。其內置的Spyder開發軟件的風格跟Matlab類似,給人熟悉的感覺,更容易上手。
再說一下更新方法:
Geatpy在持續更新。可以通過以下命令使電腦上的版本與官方最新版保持一致:
pip install --upgrade geatpy
若在更新過程中遇到"--user"錯誤的問題,這是用pip進行安裝時遇到的常見問題之一。意味著需要以管理員方式運行:
pip install --user --upgrade geatpy
Geatpy提供2種方式來使用進化算法求解問題。先來講一下第一種最基本的實現方式:編寫編程腳本。
1. 編寫腳本實現遺傳算法:
以一個非常簡單的單目標優化問題為例:求f(x)=x*sin(10*pi*x)+2.0 在 x∈[-1,2] 上的最大值。
直接編寫腳本如下:
"""demo.py"""
import numpy as np
import geatpy as ea # 導入geatpy庫
import matplotlib.pyplot as plt
import time
"""============================目標函數============================"""
def aim(x): # 傳入種群染色體矩陣解碼后的基因表現型矩陣
return x * np.sin(10 * np.pi * x) + 2.0
x = np.linspace(-1, 2, 200)
plt.plot(x, aim(x)) # 繪制目標函數圖像
"""============================變量設置============================"""
x1 = [-1, 2] # 自變量范圍
b1 = [1, 1] # 自變量邊界
varTypes = np.array([0]) # 自變量的類型,0表示連續,1表示離散
Encoding = 'BG' # 'BG'表示采用二進制/格雷編碼
codes = [1] # 變量的編碼方式,2個變量均使用格雷編碼
precisions =[4] # 變量的編碼精度
scales = [0] # 采用算術刻度
ranges=np.vstack([x1]).T # 生成自變量的范圍矩陣
borders=np.vstack([b1]).T # 生成自變量的邊界矩陣
"""=========================遺傳算法參數設置========================="""
NIND = 40; # 種群個體數目
MAXGEN = 25; # 最大遺傳代數
FieldD = ea.crtfld(Encoding,varTypes,ranges,borders,precisions,codes,scales) # 調用函數創建區域描述器
Lind = int(np.sum(FieldD[0, :])) # 計算編碼后的染色體長度
obj_trace = np.zeros((MAXGEN, 2)) # 定義目標函數值記錄器
var_trace = np.zeros((MAXGEN, Lind)) # 定義染色體記錄器,記錄每一代最優個體的染色體
"""=========================開始遺傳算法進化========================"""
start_time = time.time() # 開始計時
Chrom = ea.crtbp(NIND, Lind) # 生成種群染色體矩陣
variable = ea.bs2real(Chrom, FieldD) # 對初始種群進行解碼
ObjV = aim(variable) # 計算初始種群個體的目標函數值
best_ind = np.argmax(ObjV) # 計算當代最優個體的序號
# 開始進化
for gen in range(MAXGEN):
FitnV = ea.ranking(-ObjV) # 根據目標函數大小分配適應度值(由于遵循目標最小化約定,因此最大化問題要對目標函數值乘上-1)
SelCh=Chrom[ea.selecting('rws', FitnV, NIND-1), :] # 選擇,采用'rws'×××賭選擇
SelCh=ea.recombin('xovsp', SelCh, 0.7) # 重組(采用兩點交叉方式,交叉概率為0.7)
SelCh=ea.mutbin(Encoding, SelCh) # 二進制種群變異
# 把父代精英個體與子代合并
Chrom = np.vstack([Chrom[best_ind, :], SelCh])
variable = ea.bs2real(Chrom, FieldD) # 對育種種群進行解碼(二進制轉十進制)
ObjV = aim(variable) # 求育種個體的目標函數值
# 記錄
best_ind = np.argmax(ObjV) # 計算當代最優個體的序號
obj_trace[gen, 0] = np.sum(ObjV) / NIND # 記錄當代種群的目標函數均值
obj_trace[gen, 1] = ObjV[best_ind] # 記錄當代種群最優個體目標函數值
var_trace[gen, :] = Chrom[best_ind, :] # 記錄當代種群最優個體的變量值
# 進化完成
end_time = time.time() # 結束計時
"""============================輸出結果及繪圖================================"""
best_gen = np.argmax(obj_trace[:, [1]])
print('目標函數最大值:', obj_trace[best_gen, 1]) # 輸出目標函數最大值
variable = ea.bs2real(var_trace[[best_gen], :], FieldD) # 解碼得到表現型
print('對應的決策變量值為:')
print(variable[0][0]) # 因為此處variable是一個矩陣,因此用[0][0]來取出里面的元素
print('用時:', end_time - start_time)
plt.plot(variable, aim(variable),'bo')
ea.trcplot(obj_trace, [['種群個體平均目標函數值', '種群最優個體目標函數值']])
運行結果如下:
目標函數最大值: 3.850272716105895
對應的決策變量值為:
1.8505813776055176
用時: 0.02496051788330078
仔細查看上述代碼,我們會發現Geatpy的書寫風格與Matlab大同小異,有Matlab相關編程經驗的基本上可以無縫轉移到Python上利用Geatpy進行遺傳算法程序開發。
Geatpy提供了詳盡的API文檔,比如要查看上面代碼中的"ranking"函數是干什么的,可以在python中執行
import geatpy as ga
help(ga.ranking)
即可看到"ranking"函數的相關使用方法。
另外官網上也有更多詳盡的Geatpy教程:http://geatpy.com/index.php/details/
2. 利用框架實現遺傳算法。
Geatpy提供開放的面向對象進化算法框架。即“問題類”+“進化算法模板類+種群類”。對于一些復雜的進化算法,如多目標進化優化、改進的遺傳算法等,按照上面所說的編寫腳本代碼是非常麻煩的,而用框架的方法可以極大提高編程效率。
這里給出一個利用框架實現NSGA-II算法求多目標優化函數ZDT-1的帕累托前沿面的例子:
第一步:首先編寫ZDT1的問題類,寫在“MyProblem.py”文件中:
# -*- coding: utf-8 -*-
"""MyProblem.py"""
import numpy as np
import geatpy as ea
class MyProblem(ea.Problem): # 繼承Problem父類
def __init__(self):
name = 'ZDT1' # 初始化name(函數名稱,可以隨意設置)
M = 2 # 初始化M(目標維數)
maxormins = [1] * M # 初始化maxormins(目標最小最大化標記列表,1:最小化該目標;-1:最大化該目標)
Dim = 30 # 初始化Dim(決策變量維數)
varTypes = [0] * Dim # 初始化varTypes(決策變量的類型,0:實數;1:整數)
lb = [0] * Dim # 決策變量下界
ub = [1] * Dim # 決策變量上界
lbin = [1] * Dim # 決策變量下邊界
ubin = [1] * Dim # 決策變量上邊界
# 調用父類構造方法完成實例化
ea.Problem.__init__(self, name, M, maxormins, Dim, varTypes, lb, ub, lbin, ubin)
def aimFunc(self, pop): # 目標函數
Vars = pop.Phen # 得到決策變量矩陣
ObjV1 = Vars[:, 0]
gx = 1 + 9 * np.sum(Vars[:, 1:30], 1)
hx = 1 - np.sqrt(ObjV1 / gx)
ObjV2 = gx * hx
pop.ObjV = np.array([ObjV1, ObjV2]).T # 把結果賦值給ObjV
def calBest(self): # 計算全局最優解
N = 10000 # 生成10000個參考點
ObjV1 = np.linspace(0, 1, N)
ObjV2 = 1 - np.sqrt(ObjV1)
globalBestObjV = np.array([ObjV1, ObjV2]).T
return globalBestObjV
上面代碼中,問題類的構造函數__init__()是用于定義與ZDT1測試問題相關的一些參數,如決策變量范圍、類型、邊界等等。aimFunc()是待優化的目標函數。calBest()可以用來計算理論的全局最優解,這個理論最優解可以是通過計算得到的,也可以是通過導入外部文件的數據得到的,如果待求解的問題沒有或尚不知道理論最優解是多少,則這個calBest()函數可以省略不寫。
第二步:在同一個文件夾下編寫執行腳本,實例化上述問題類的對象,然后調用Geatpy提供的nsga2算法的進化算法模板(moea_NSGA2_templet),最后結合理論全局最優解PF(即俗稱的“真實前沿點”)通過計算GD、IGD、HV等指標來分析優化效果:
# -*- coding: utf-8 -*-
import geatpy as ea # import geatpy
from MyProblem import MyProblem
"""================================實例化問題對象============================="""
problem = MyProblem() # 生成問題對象
"""==================================種群設置================================"""
Encoding = 'RI' # 編碼方式
NIND = 50 # 種群規模
Field = ea.crtfld(Encoding, problem.varTypes, problem.ranges, problem.borders) # 創建區域描述器
population = ea.Population(Encoding, Field, NIND) # 實例化種群對象(此時種群還沒被初始化,僅僅是完成種群對象的實例化)
"""================================算法參數設置==============================="""
myAlgorithm = ea.moea_NSGA2_templet(problem, population) # 實例化一個算法模板對象`
myAlgorithm.MAXGEN = 200 # 最大進化代數
myAlgorithm.drawing = 1 # 設置繪圖方式(0:不繪圖;1:繪制結果圖;2:繪制過程動畫)
"""===========================調用算法模板進行種群進化===========================
調用run執行算法模板,得到帕累托最優解集NDSet。NDSet是一個種群類Population的對象。
NDSet.ObjV為最優解個體的目標函數值;NDSet.Phen為對應的決策變量值。
詳見Population.py中關于種群類的定義。
"""
NDSet = myAlgorithm.run() # 執行算法模板,得到非支配種群
NDSet.save() # 把結果保存到文件中
# 輸出無錫婦科檢查多少錢 http://www.120csfkyy.com/
print('用時:%f 秒'%(myAlgorithm.passTime))
print('評價次數:%d 次'%(myAlgorithm.evalsNum))
print('非支配個體數:%d 個'%(NDSet.sizes))
print('單位時間找到帕累托前沿點個數:%d 個'%(int(NDSet.sizes // myAlgorithm.passTime)))
# 計算指標
PF = problem.getBest() # 獲取真實前沿,詳見Problem.py中關于Problem類的定義
if PF is not None and NDSet.sizes != 0:
GD = ea.indicator.GD(NDSet.ObjV, PF) # 計算GD指標
IGD = ea.indicator.IGD(NDSet.ObjV, PF) # 計算IGD指標
HV = ea.indicator.HV(NDSet.ObjV, PF) # 計算HV指標
Spacing = ea.indicator.Spacing(NDSet.ObjV) # 計算Spacing指標
print('GD',GD)
print('IGD',IGD)
print('HV', HV)
print('Spacing', Spacing)
"""=============================進化過程指標追蹤分析============================"""
if PF is not None:
metricName = [['IGD'], ['HV']]
[NDSet_trace, Metrics] = ea.indicator.moea_tracking(myAlgorithm.pop_trace, PF, metricName, problem.maxormins)
# 繪制指標追蹤分析圖
ea.trcplot(Metrics, labels = metricName, titles = metricName)
運行結果如下:
種群信息導出完畢。
用時:0.503653 秒
評價次數:10000 次
非支配個體數:50 個
單位時間找到帕累托前沿點個數:99 個
GD 0.0011025023611967554
IGD 0.15098973339777405
HV 0.624906599521637
Spacing 0.009326105831814594
正在進行進化追蹤指標分析,請稍后......
指標追蹤分析結束,進化記錄器中有效進化代數為: 200
上述代碼中已經對各個流程進行了詳細的注釋。其中進化算法的核心邏輯是寫在進化算法模板內部的,可前往查看對應的源代碼。此外,我們還可以參考Geatpy進化算法模板的源代碼來自定義算法模板,以實現豐富多樣的進化算法,如各種各樣的改進的進化算法等:
最后值得注意的是:目標函數aimFunc()那一塊地方最容易寫錯。aimFunc()的輸入參數pop是一個種群對象(有關種群對象可以查看工具箱中的Population.py類源碼,或者查看Geatpy數據結構)。pop.Phen是種群的表現型矩陣,意思是種群染色體解碼后得到的表現型矩陣,它對應的即為問題類中的決策變量。Phen是一個矩陣,每一行對應種群中的一個個體的表現型。在計算目標函數時,可以把這個Phen拆成一行一行,即逐個逐個個體地計算目標函數值,然后再拼成一個矩陣賦值給pop對象的ObjV屬性。也可以利用Numpy的矩陣化計算來“一口氣”把種群所有個體的目標函數值計算出來。無論采用的是哪種計算方法,最后得到的目標函數值是要保存在pop對象的ObjV屬性中的,這個ObjV是“種群目標函數值矩陣”,每一行對應一個個體的所有目標函數值,每一列對應一個目標。比如:
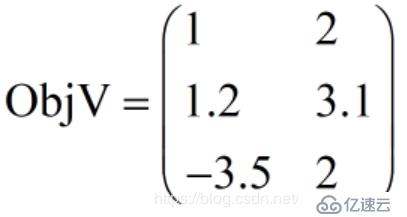
它表示有種群3個個體,待優化目標有2個。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。