您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這期內容當中小編將會給大家帶來有關如何分析基因型填充的在線工具Michigan Imputation Server,文章內容豐富且以專業的角度為大家分析和敘述,閱讀完這篇文章希望大家可以有所收獲。
得益于分析軟件運行速度的不斷提高,硬件資源消耗的不斷優化,基因型填充這一計算量巨大的任務也出現了web服務,Michigan Imputation Server就是其中之一。
采用Eagle進行pre-phasing, minimac4進行填充的分析策略,保證了運行速度。該平臺支持hg19和hg38兩個基因組版本,支持的reference panel列表如下
HRC (Version r1.1 2016)
1000 Genomes Phase 1 (Version 3)
1000 Genomes Phase 3 (Version 5)
CAAPA - African American Panel
HapMap 2
要求輸入文件為VCF格式,可以通過以下方法轉換得到
# plink將ped/map轉換為vcf格式
plink --file sample --recode vcf --chr 1 --out sample.chr1
# vcftools對VCF文件排序
# bgzip壓縮VCF文件
vcf-sort sample.chr1.vcf | bgzip -c > sample.chr1.vcf.gz按照染色體進行拆分,每條染色體一個VCF文件,然后通過提交頁面上傳即可,示意如下
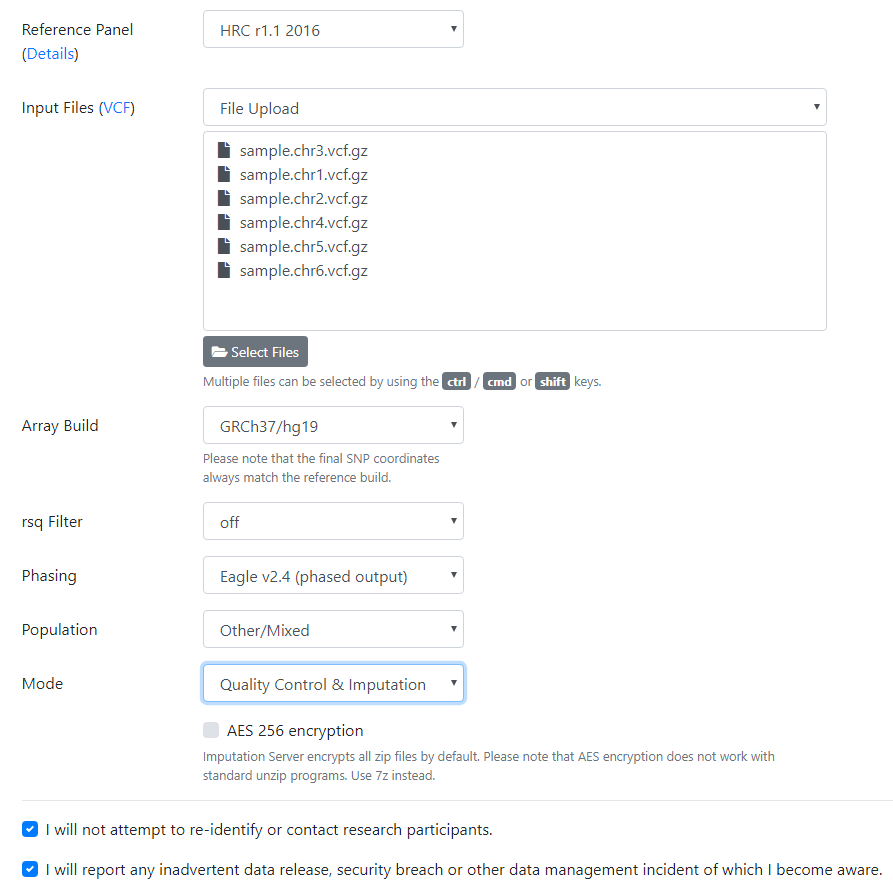
選擇參考的panel, 上傳VCF格式的輸入文件,設置參考基因組版本,然后提交即可。運行的步驟如下
按照20M的長度將染色體拆分成chunk, 統計輸入文件的樣本數,染色體條數,位點數,chunk數,reference panel等基本信息,結果示意如下
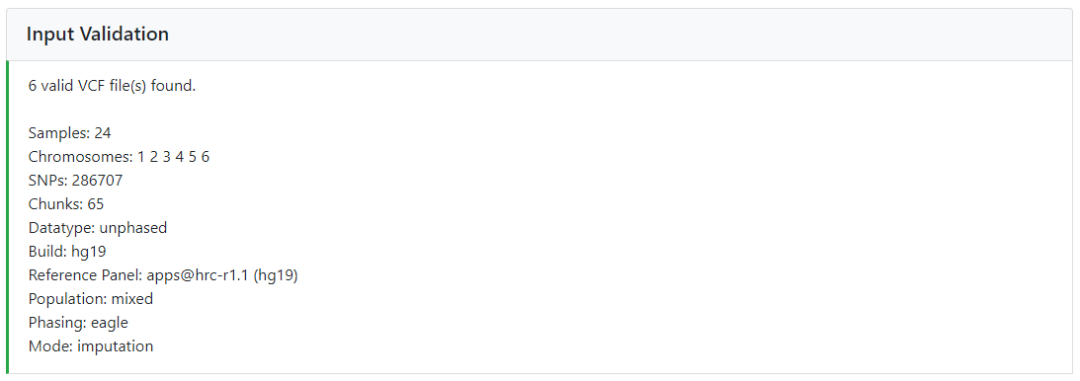
從snp和sample兩個方面進行指控,結果統計如下

對于每個chunks, 進行pre-phasing和imputation
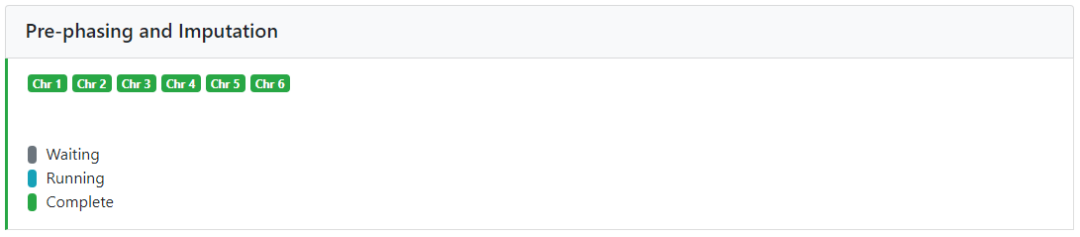
運行完成后,合并同一染色體的不同chunks,然后將結果打包,并加密,輸出結果示意如下

該網站內置了HRC reference panel這個最大規模的單倍型數據集,在運行速度快的同時也保證了填充的準確性,盡管其填充準確率不會像impute2那么高。
上述就是小編為大家分享的如何分析基因型填充的在線工具Michigan Imputation Server了,如果剛好有類似的疑惑,不妨參照上述分析進行理解。如果想知道更多相關知識,歡迎關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。