您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“如何通過GDC Legacy Archive下載TCGA原始數據”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“如何通過GDC Legacy Archive下載TCGA原始數據”吧!
在2016年之前,TCGA項目的相關結果文件存放在CGhub和TCGA Data Coordinating Center簡稱DCC提供的TCGA Data Portal中,當時的結果是以hg19或者hg18為參考得到的。
在DCC中,將數據劃分為了3個等級。level 1代筆原始的,未經處理的數據的,比如芯片下機數據;level2 代表處理的中間結果,比如測序深度對應的wig文件;level 3 代表處理完成后的最終結果,比如基因的定量結果。
2016年之后,CGhub和DCC相繼關閉,所有的數據統一遷移到現在用的GDC數據庫,而且通過GDC的pipeline將原有的結果轉換為hg38參考基因組版本。目前在GDC中檢索到的結果都是經過了GDC pipeline處理過后的,從這里也可以看出,遷移到hg38是一個大的趨勢。
當然目前使用hg19的還是挺多的,如果你需要基于hg19版本的TCGA數據,在GDC中也可以找到。其實GDC中的數據可以分為以下兩個部分
GDC harmonized data
GDC legacy archive
在R包TCGAbiolinks中,介紹了二者的區別,如下圖所示

第一部分就是默認使用的基于hg38版本的數據,第二部分則是對原始的TCGA結果的一個存儲,通過GDC首頁的GDC APPs, 可以找到CDC Legacy Archive的入口,鏈接如下
https://portal.gdc.cancer.gov/legacy-archive
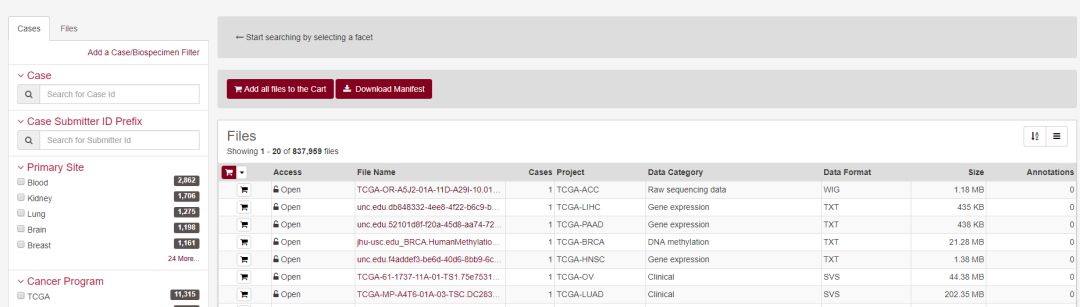
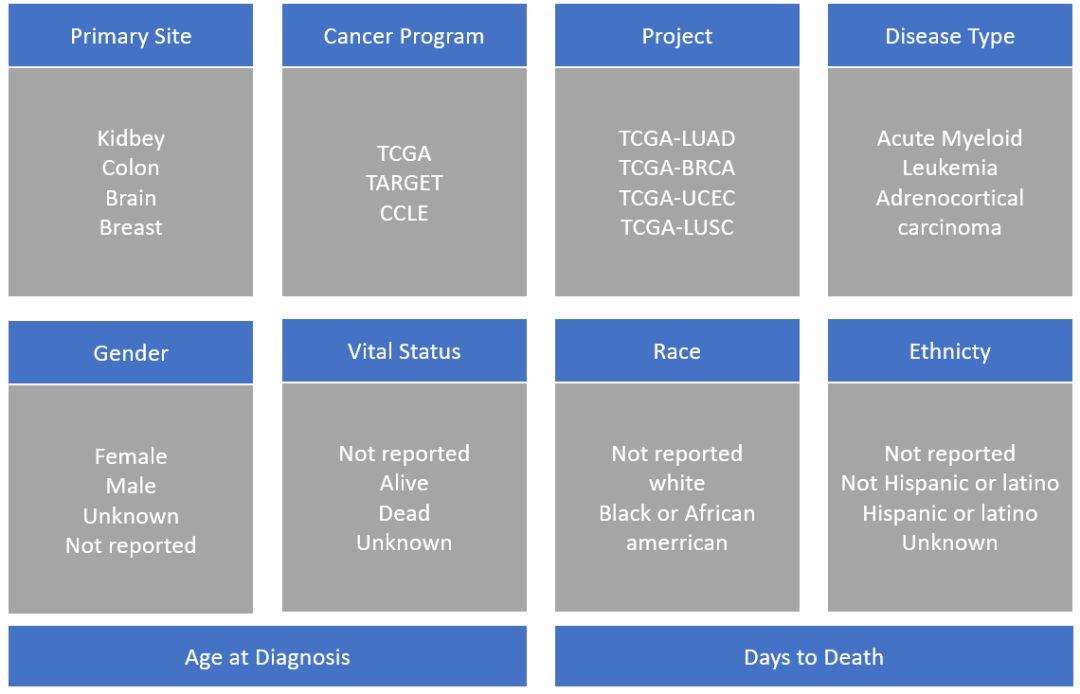
在左側的面板可以根據相關屬性對Cases和Files進行篩選,Cases相關的屬性如下
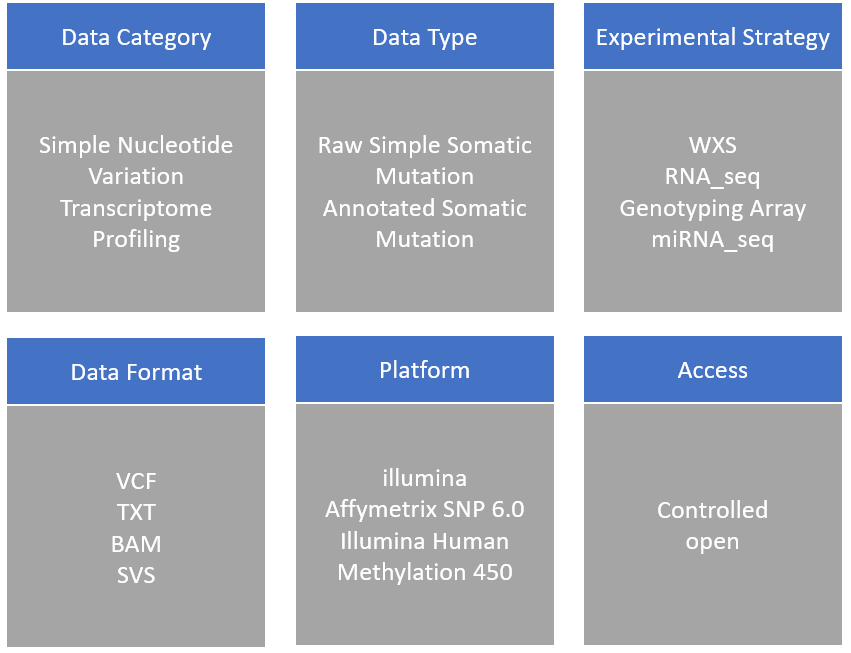
Files相關的屬性如下
數據的下載方式和前面文章中介紹的相同,這里不贅述,從文件名稱可以看到對應的level, 不同level的文件示意如下
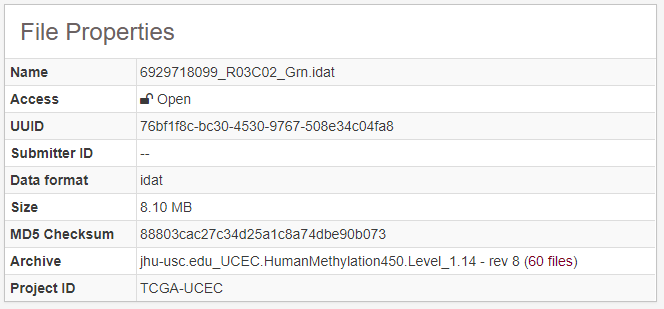
通過Data Type為Raw intensitites進行篩選,得到芯片的原始數據, 示意如下
通過Data Type為Coverage WIG進行篩選,得到比對的測序深度數據, 示意如下
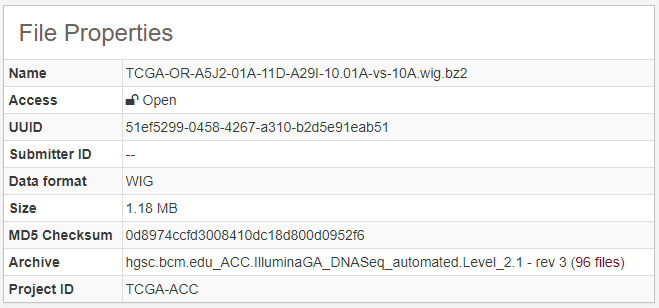
通過Data Type為miRNA gene quantification進行篩選,得到miRNA表達定量數據, 示意如下
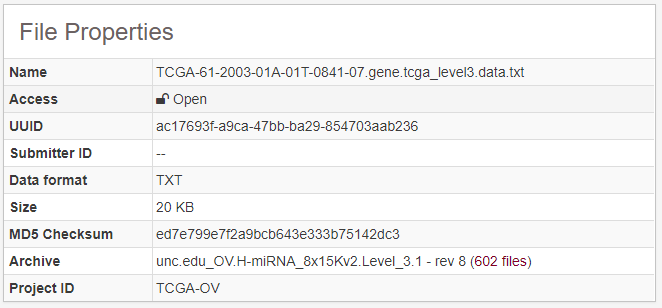
通過GDC Legacy Archive, 可以找到基于hg19的數據結果文件,但是由于相關的網站已經關閉,無法確認該數據分析的pipieline等細節信息,所以需要謹慎使用。
到此,相信大家對“如何通過GDC Legacy Archive下載TCGA原始數據”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。