您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容主要講解“narrow,broad, gapped peak三種格式之間的區別與聯系”,感興趣的朋友不妨來看看。本文介紹的方法操作簡單快捷,實用性強。下面就讓小編來帶大家學習“narrow,broad, gapped peak三種格式之間的區別與聯系”吧!
在進行peak calling分析時,經常會接觸到以下3種peak格式
narrow peaks format
broad peaks fotmat
gapped peaks format
peak被定義為基因組上一段reads富集的區域,核心信息是在染色體上的起始和終止位置,除此之外,還有軟件對于該peak區域的打分,比如常見的pvalue, qvalue, fold_enrichment等值。
和基因組比對信息用BAM格式來存儲類似,為了標準化不同peak calling軟件的輸出,特意制定了以上3種數據格式。這三種格式本質上都是bed文件,只不過列數不太類似。
該格式又稱之為point-source peaks format, macs2默認輸出就是這種格式,是一種BED6+4的格式,列數為10列,示意如下
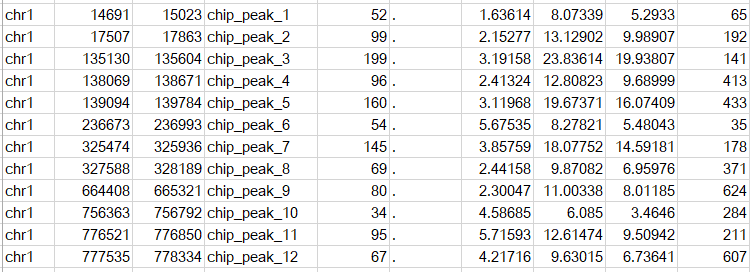
前四列分別代表chrom, chromStart, chromEnd, name, 用于描述peak區間和名稱,注意bed格式中起始位置從0開始計數。
第五列代表score,在macs2的輸出結果中為int(-10*log10qvalue),第六列代表strand, 在macs2的輸出結果中為.,第七列代表signalvalue, 通常使用fold_enrichment的值,第八列代表pvalue, 在macs2的輸出結果中為-log10(pvalue),第九列代表qvalue, 在macs2的輸出結果中為-log10(qvalue),第十列代表peak, 在macs2的輸出結果中為peak的中心,即summit距離peak起始位置的距離。
這種格式就是在narrow peaks format的基礎上丟掉了最后一列的信息,為BED6+3的格式, 列數為9列。
前兩種格式都是由于描述連續的peak區間,適用于DNA水平上的富集區域信息的存儲,比如chip_seq, ATAC_seq鑒定到的peak區間,而gapped peaks format用于描述非連續的peak區間,這里的非連續通常指的是在peak的區間內會包含多個exon區域,適用于RNA水平上的富集區域信息的存儲,比如m6A_seq鑒定到的peak區間。
該格式在BED12的基礎上進行延伸,演變為BED12+3的格式,列數為15列,每列的含義示意如下

前6列的含義和上述兩種peak格式完全相同,后3列的含義和broad peak完全相同,為了專區表示peak區間內包含的exon信息,借鑒轉錄本的BED12格式,引入了以下6列
thickStart
thickEnd
itemRgb
blockCount
blockSizes
blockStarts
thickStart和thickEnd有點類似轉錄本中CDS的起始和終止位置,在存儲peak信息時,通常的做法是將這兩列的值和chromStart和chromEnd的值設置成相同的,itemRgb是一個RGB顏色值,比如255,0,0, 如果沒有對應的顏色信息,則用0來表示。
blockCount代表該peak區間包含的exon的個數,blockSizes代表每個exon區間的長度,多個exon用逗號連接,blockStarts代表每個exon區間在基因組上的起始位置,多個exon用逗號連接。
關于這三種格式的相關介紹請參考以下鏈接
https://genome.ucsc.edu/FAQ/FAQformat.html#format13
到此,相信大家對“narrow,broad, gapped peak三種格式之間的區別與聯系”有了更深的了解,不妨來實際操作一番吧!這里是億速云網站,更多相關內容可以進入相關頻道進行查詢,關注我們,繼續學習!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。