您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關FastTree軟件有什么用,小編覺得挺實用的,因此分享給大家做個參考,希望大家閱讀完這篇文章后可以有所收獲。
FastTree 是基于最大似然法構建進化樹的軟件,它最大的特點就是運行速度快,支持幾百萬條序列的建樹任務。官方的說法是,對于大的比對數據集,FastTree 比phyml或者RAxML 快100到1000倍。官網如下
http://www.microbesonline.org/fasttree/
FastTree 支持核酸和蛋白的進化樹構建,對于核酸,可選的替換模型包括以下幾種
JC
GTR
默認的模型為JC。
對于蛋白質,可選的替換模型包括以下幾種
JTT
LG
WAG
默認的模型為JTT。
利用不同的測試數據集,比較了fastTree 不同替換模型和RAxML, PhyML 運行速度的差異。結果如下
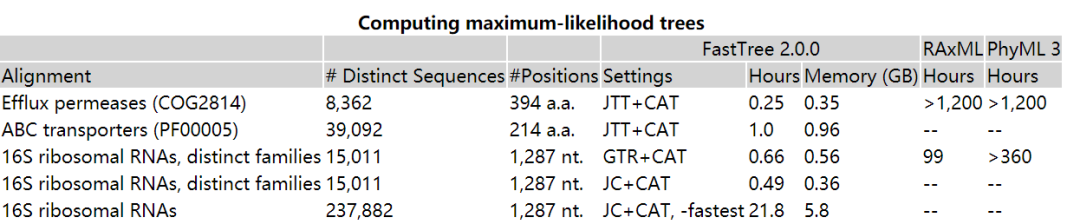
對于蛋白序列而言,FastTree 的運行速度比其他兩款軟件快了1000多倍,而且對于幾萬條序列的比對,其他兩款軟件的運行時間太久,超過了可以忍受的范圍;對于核酸序列而言,默認的JC模型的速度最快, GTR模型速度少稍差一籌,其他兩款軟件同樣運行速度慢的不行。
FastTree 除了運行速度快之外,準確度也令人滿意,比較的結果如下

對于幾萬條的核酸序列,只有FastTree, NJ, Clearcut 這3個軟件有結果,而FastTree 的準確度是最高的,從此可以看出,對于幾萬條核酸序列的進化樹分析,FastTree 是最佳選擇之一;對于蛋白序列,在可以運行出結果的前提下,FastTree 的準確度相比RAxML, PhyML 都稍差一點。
綜合運行速度和建樹的準確性,FastTree 都是最佳的進化樹構建軟件之一。
我們可以直接從官網下載可執行文件

FastTree要求輸入的多序列比對結果為FASTA或者Phylip格式,對于蛋白質的進化樹構建,基本用法如下
FastTree protein.fasta > tree
也可以選擇LG或者WAG替換模型,用法如下
FastTree -lg protein.fasta > tree FastTree -wag protein.fasta > tree
對于核酸序列,基本用法如下
FastTree -nt nucleotide.fasta > tree
也可以選擇GTR替換模型,用法如下
FastTree -nt -gtr nucleotide.fasta > tree
默認生成的tree 文件是 Newick格式, 可以導入 figTree 或者 TreeViewer等軟件中進行查看。
關于“FastTree軟件有什么用”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,使各位可以學到更多知識,如果覺得文章不錯,請把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。