您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章給大家介紹seq2HLA如何利用RNA_seq數據進行HLA分型,內容非常詳細,感興趣的小伙伴們可以參考借鑒,希望對大家能有所幫助。
對于不同的HLA Allel來說,exon2和exon3 序列的差異性尤為明顯,很多的HLA 分型軟件都會根據這部分序列,整理出HLA Allel序列參考數據庫。
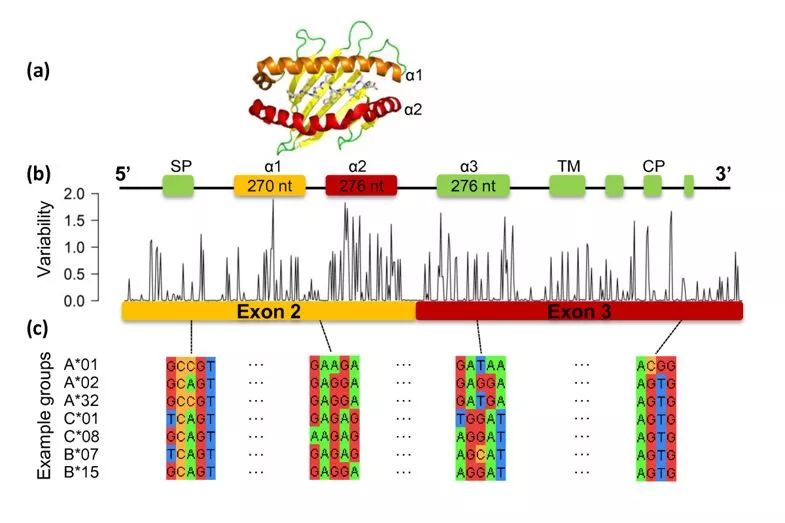
seq2HLA也采用了類似的策略,通過HLA不同Allel的exon2和exon3的序列,整理了一份HLA參考數據庫,通過將RNA_seq的reads與該數據庫比對,確定HLA分型結果,原理示意圖如下
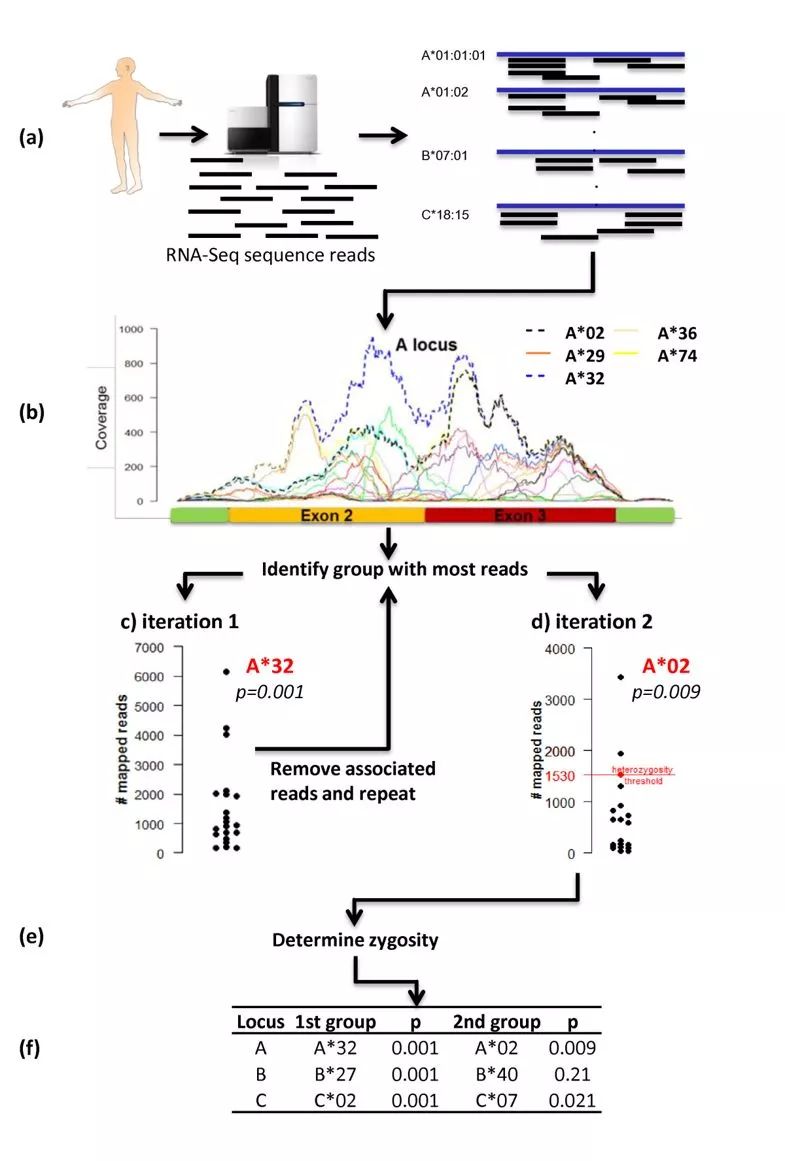
迭代兩次,每次挑選出覆蓋度最高的Allel 作為分型結果。
seq2HLA采用python和R進行開發,安裝過程較為簡單,直接下載源代碼即可,安裝過程如下
git clone https://github.com/TRON-Bioinformatics/seq2HLA cd seq2HLA/
用法如下:
python seq2HLA.py -1 R1.fastq -2 R2.fastq -r test -p 10
-1和-2參數分別指定輸入的R1和R2端的fastq格式的序列; -r參數指定輸出文件名稱的前綴,-p指定線程數,主要是bowtie比對時的線程。
輸出文件非常多,詳細列表如下
test.ambiguity test-ClassI-class.bowtielog test-ClassI-class.expression test-ClassI-class.HLAgenotype2digits test-ClassI-class.HLAgenotype4digits test-ClassII.bowtielog test-ClassII.expression test-ClassII.HLAgenotype2digits test-ClassII.HLAgenotype4digits test-ClassI-nonclass.bowtielog test-ClassI-nonclass.expression test-ClassI-nonclass.HLAgenotype2digits test-ClassI-nonclass.HLAgenotype4digits
我們主要關注后綴為HLAgenotype4digits的結果文件,可以看到,同時體用了HLA Clas I 和 Class II 兩種類型基因的分型結果。以HLA I型基因的4位分型結果為例,文件內容如下
#Locus Allele 1 Confidence Allele 2 Confidence A A*02:65 0.008687167 A*02:65 NA B B*39:05' 0.3821314 B*13:48 0.09848174 C C*08:02' NA C*08:02 NA
對于HlA I型基因,給出了A, B, C 三個基因的分型結果,每個基因給出了兩個Allel, 對于每個Allel, 會給出對應的打分值。
關于seq2HLA如何利用RNA_seq數據進行HLA分型就分享到這里了,希望以上內容可以對大家有一定的幫助,可以學到更多知識。如果覺得文章不錯,可以把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。