您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章將為大家詳細講解有關如何分析breakdancer檢測結構變異,文章內容質量較高,因此小編分享給大家做個參考,希望大家閱讀完這篇文章后對相關知識有一定的了解。
breakdancer 是一款結構變異檢測軟件, 專門針對雙端測序數據進行開發,github地址如下
https://github.com/genome/breakdancer
分析原理圖如下
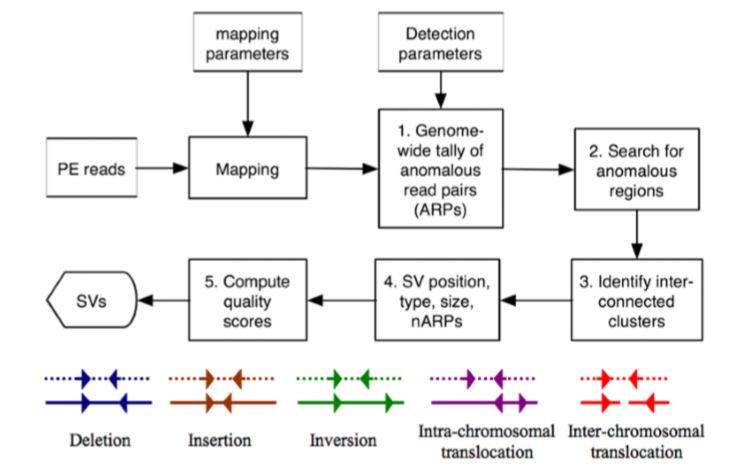
從原理圖可以看出,breakdancer 會根據雙端reads的比對情況,檢測以下5種類型的結構變異
insertions
deletions
inversions
inter-chromosomal translocations
intra-chromosomal translocations
軟件的安裝過程如下
git clone --recursive https://github.com/genome/breakdancer.git cd breakdancer mkdir build cd build cmake .. -DCMAKE_BUILD_TYPE=release -DCMAKE_INSTALL_PREFIX=/usr/local make
最終會生成一個可執行的二進制文件,breakdancer-max。軟件的使用也比較簡單,共兩個步驟。
輸入文件為比對基因組產生的bam文件, 用法如下
bam2cfg.pl tumor.bam normal.bam > config.txt
配置文件中,每個樣本對應一行記錄,包含以下特征值
readgroup:tumor platform:illumina map:tumor.bam readlen:144.84 lib:YL num:10000 lower:0.00 upper:519.05 mean:210.24 std:65.87 SWnormality:-40.54 exe:samtools view
用法如下
breakdancer_max -t -q 10 -d sv.reads config.txt > sv.out
結構變異的檢測計算量較大,所以需要的時間也很久。輸出文件的列數很多,共有14列。
每一列的含義如下
Chromosome 1
Position 1
Orientation 1
Chromosome 2
Position 2
Orientation 2
Type of a SV
Size of a SV
Confidence Score
Total number of supporting read pairs
Total number of supporting read pairs from each map file
Estimated allele frequency
Software version
The run parameters
1到6列描述的是斷裂點的位置信息;第7列描述結構變異的類型,DEL代表缺失,INS代表插入,INV代表倒位,ITX代表同一染色體上的易位,CTX代表不同染色體之間的易位;第8列代表結構變異的長度,對于染色體間的易位,這個數值沒有含義;第9列代表該結構變異可信度的打分值,數值越大,可靠性越高。
關于如何分析breakdancer檢測結構變異就分享到這里了,希望以上內容可以對大家有一定的幫助,可以學到更多知識。如果覺得文章不錯,可以把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。