溫馨提示×
您好,登錄后才能下訂單哦!
點擊 登錄注冊 即表示同意《億速云用戶服務條款》
您好,登錄后才能下訂單哦!
本篇文章給大家分享的是有關R語言ggplot2如何實現坐標軸放到右邊、更改繪圖邊界和數據分組排序,小編覺得挺實用的,因此分享給大家學習,希望大家閱讀完這篇文章后可以有所收獲,話不多說,跟著小編一起來看看吧。
正常坐標軸都是位于左邊和下邊,如果要改成上邊或者右邊可以使用如下代碼
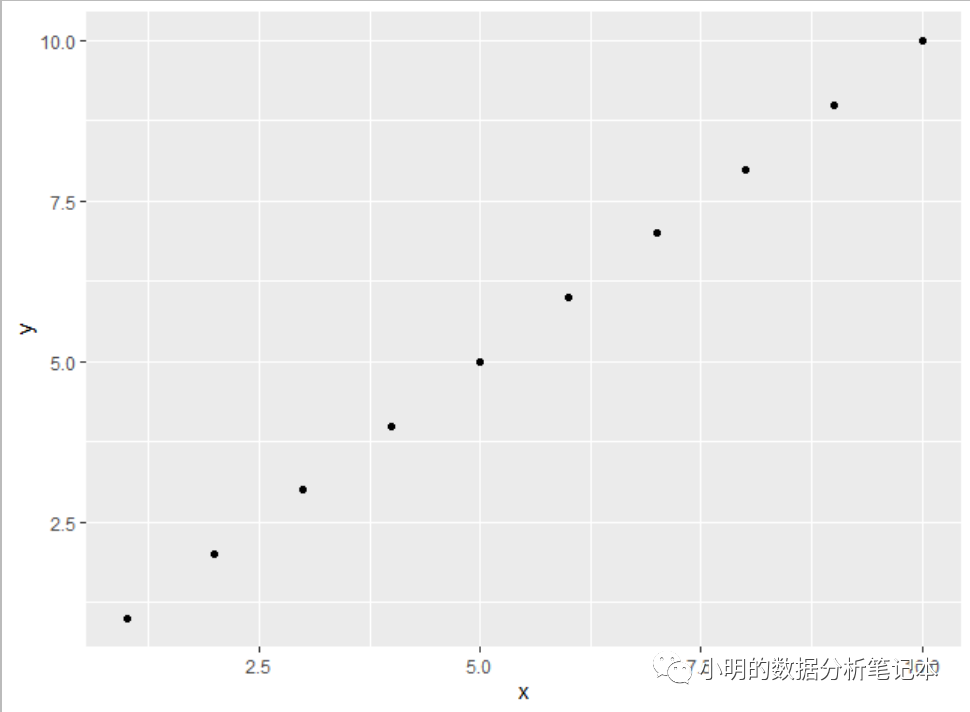
正常
library(ggplot2)
df<-data.frame(x=1:10,y=1:10)
ggplot(df,aes(x=x,y=y))+
geom_point()
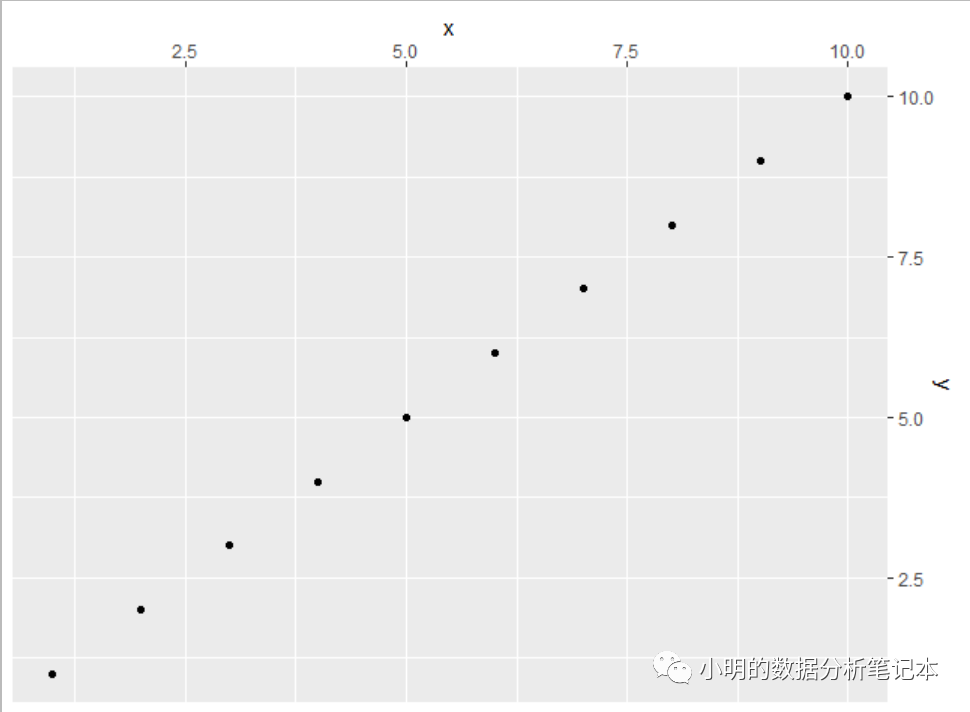
不正常
ggplot(df,aes(x=x,y=y))+
geom_point()+
scale_x_continuous(position = "top")+
scale_y_continuous(position = "right")
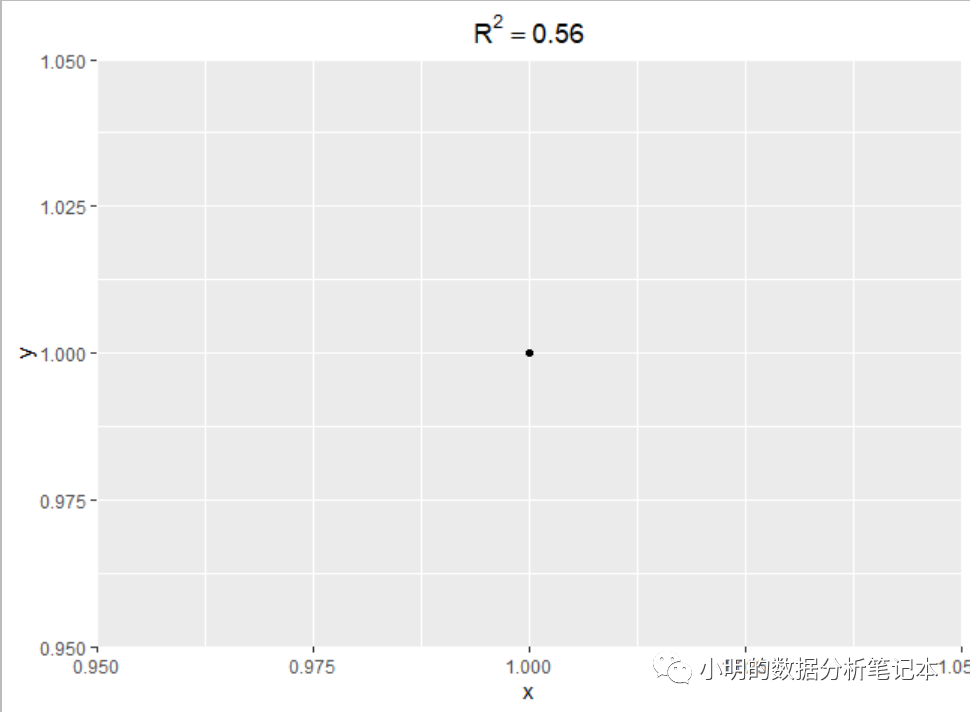
library(ggplot2)
ggplot()+
geom_point(aes(x=1,y=1))+
labs(title=expression(R^2==0.56))+
theme(plot.title = element_text(hjust=0.5))
library(ggplot2)
ggplot()+
geom_point(aes(x=1,y=1))+
labs(title=expression(R^2==0.56))+
theme(plot.title = element_text(hjust=0.5),
plot.margin = unit(c(1,4,1,1),"lines"))
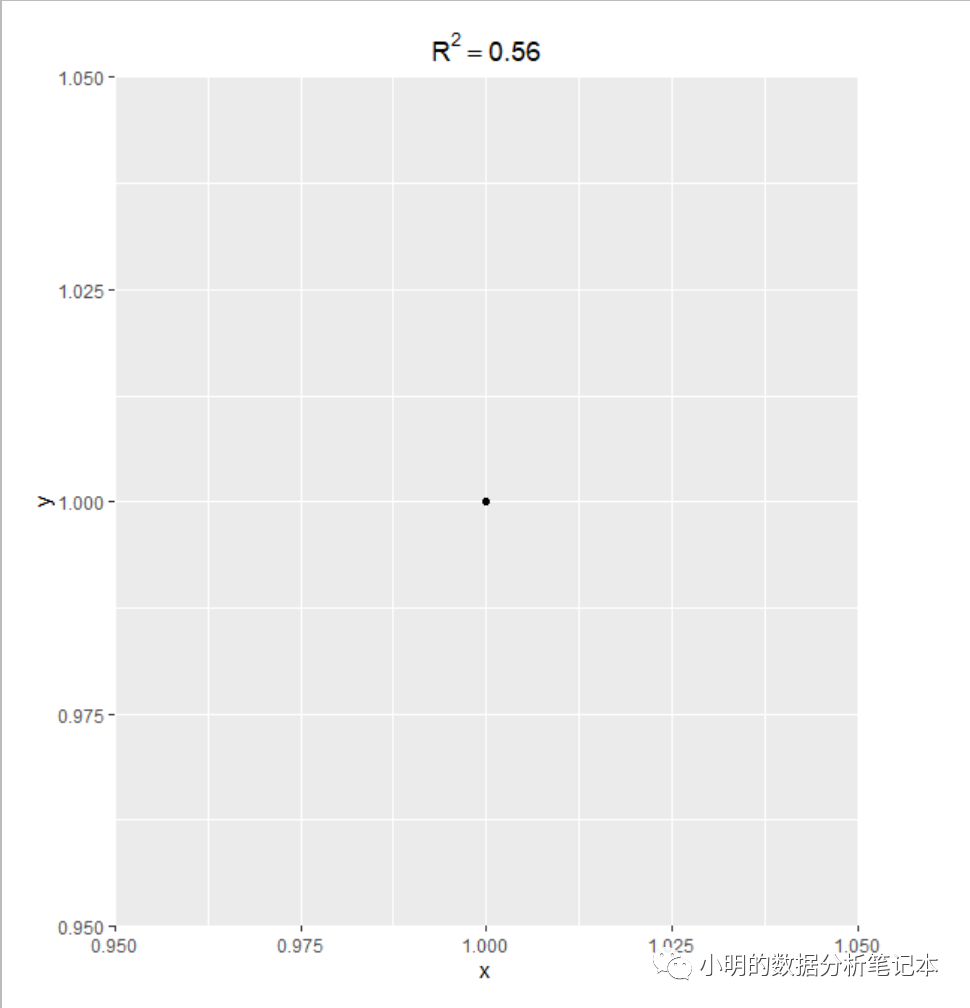
四個位置 控制的分別是上右下 左
> df<-data.frame(group=c("A","A","A","B","B","B"),
+ value=c(5,4,7,2,6,4))
> library(dplyr)
Attaching package: ‘dplyr’
The following objects are masked from ‘package:stats’:
filter, lag
The following objects are masked from ‘package:base’:
intersect, setdiff, setequal, union
> df%>%
+ group_by(group)%>%
+ arrange(group,value)
# A tibble: 6 x 2
# Groups: group [2]
group value
<chr> <dbl>
1 A 4
2 A 5
3 A 7
4 B 2
5 B 4
6 B 6
GO注釋的結果通常是兩列,第一列是GO號,第二列是好多基因名,用逗號分隔。就是下面這種
GO0001 gene1,gene2
GO0002 gene5,gene3,gene4
GO0003 gene3,gene10
有時候我們需要把它整理成,單個基因對應一個GO號的形式
就是這種
V1 V2
<chr> <chr>
1 GO0001 gene1
2 GO0001 gene2
3 GO0002 gene5
4 GO0002 gene3
5 GO0002 gene4
6 GO0003 gene3
7 GO0003 gene10
我最開始的解決辦法是寫簡單的python腳本,昨天在一個微信群里看到有人給出的R語言代碼,很好用,記錄在這里
#install.packages("tidyr")
library(tidyr)
df<-read.table("../Some_data/pra.txt",header=F,sep=" ")
df%>%
separate_rows(V2,sep=",")以上就是R語言ggplot2如何實現坐標軸放到右邊、更改繪圖邊界和數據分組排序,小編相信有部分知識點可能是我們日常工作會見到或用到的。希望你能通過這篇文章學到更多知識。更多詳情敬請關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。