您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要講解了“qt MAF過濾的方法”,文中的講解內容簡單清晰,易于學習與理解,下面請大家跟著小編的思路慢慢深入,一起來研究和學習“qt MAF過濾的方法”吧!
經過去掉缺失,去掉錯誤的性別信息,得到的文件為:
HapMap_3_r3_6.bed HapMap_3_r3_6.fam HapMap_3_r3_6.log
HapMap_3_r3_6.bim HapMap_3_r3_6.hh
這里,我們根據最小等位基因頻率(MAF)去篩選。
「為什么要根據MAF去篩選?」
?最小等位基因頻率怎么計算?比如一個位點有AA或者AT或者TT,那么就可以計算A的基因頻率和T的基因頻率,qA + qT = 1,這里誰比較小,誰就是最小等位基因頻率,比如qA = 0.3, qT = 0.7, 那么這個位點的MAF為0.3. 之所以用這個過濾標準,是因為MAF如果非常小,比如低于0.02,那么意味著大部分位點都是相同的基因型,這些位點貢獻的信息非常少,增加假陽性。更有甚者MAF為0,那就是所有位點只有一種基因型,這些位點沒有貢獻信息,放在計算中增加計算量,沒有意義,所以要根據MAF進行過濾。
?
「思路:」
「提取常染色體上的位點名稱:」
因為這里是人的數據,所以染色體只需要去1~22的常染色體,提取它的家系ID和個體ID,后面用于提取。
awk '{ if($1 >=1 && $1 <= 22) print $2}' HapMap_3_r3_6.bim > snp_1_22.txt
wc -l snp_1_22.txt
1399306 snp_1_22.txt 常染色體上共有1399306位點。
這里,用到了位點提取參數--extract
plink --bfile HapMap_3_r3_6 --extract snp_1_22.txt --make-bed --out HapMap_3_r3_7
「查看日志:」
PLINK v1.90b6.5 64-bit (13 Sep 2018) www.cog-genomics.org/plink/1.9/
(C) 2005-2018 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to HapMap_3_r3_7.log.
Options in effect:
--bfile HapMap_3_r3_6
--extract snp_1_22.txt
--make-bed
--out HapMap_3_r3_7
515185 MB RAM detected; reserving 257592 MB for main workspace.
1431211 variants loaded from .bim file.
163 people (79 males, 84 females) loaded from .fam.
112 phenotype values loaded from .fam.
--extract: 1399306 variants remaining.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 112 founders and 51 nonfounders present.
Calculating allele frequencies... done.
Total genotyping rate is 0.998153.
1399306 variants and 163 people pass filters and QC.
Among remaining phenotypes, 56 are cases and 56 are controls. (51 phenotypes
are missing.)
--make-bed to HapMap_3_r3_7.bed + HapMap_3_r3_7.bim + HapMap_3_r3_7.fam ...
done.
可以看到,共有163個基因型,共有1399306個SNP
首先,通過參數--freq,計算每個SNP的MAF頻率,通過直方圖查看整體分布。可視化會更加直接。
plink --bfile HapMap_3_r3_7 --freq --out MAF_check
結果文件:MAF_check.frq預覽:
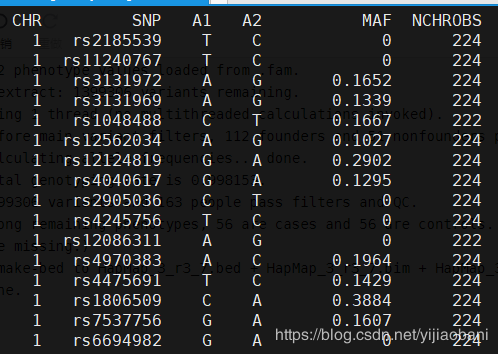
「R代碼:」
maf_freq <- read.table("MAF_check.frq", header =TRUE, as.is=T)
pdf("MAF_distribution.pdf")
hist(maf_freq[,5],main = "MAF distribution", xlab = "MAF")
dev.off() 「圖形如下:」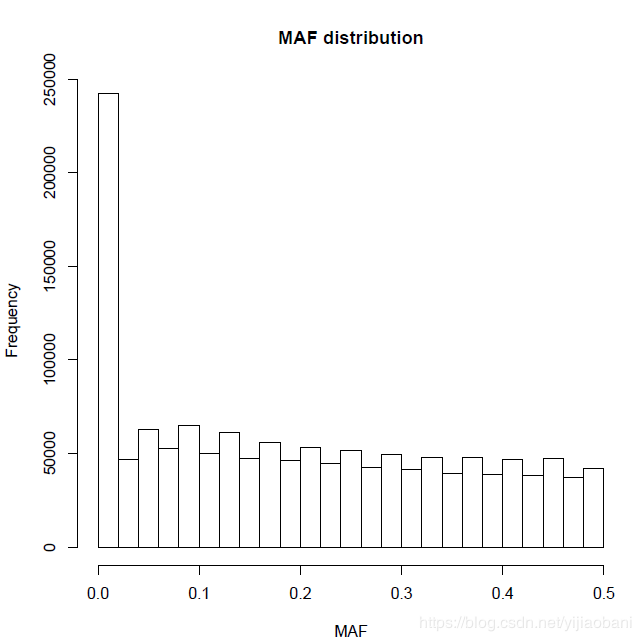
可以看出,很多基因頻率為0,說明沒有分型,這些位點需要刪掉。
plink --bfile HapMap_3_r3_7 --maf 0.05 --make-bed --out HapMap_3_r3_8 日志:
PLINK v1.90b6.5 64-bit (13 Sep 2018) www.cog-genomics.org/plink/1.9/
(C) 2005-2018 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to HapMap_3_r3_8.log.
Options in effect:
--bfile HapMap_3_r3_7
--maf 0.05
--make-bed
--out HapMap_3_r3_8
515185 MB RAM detected; reserving 257592 MB for main workspace.
1399306 variants loaded from .bim file.
163 people (79 males, 84 females) loaded from .fam.
112 phenotype values loaded from .fam.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 112 founders and 51 nonfounders present.
Calculating allele frequencies... done.
Total genotyping rate is 0.998153.
325518 variants removed due to minor allele threshold(s)
(--maf/--max-maf/--mac/--max-mac).
1073788 variants and 163 people pass filters and QC.
Among remaining phenotypes, 56 are cases and 56 are controls. (51 phenotypes
are missing.)
--make-bed to HapMap_3_r3_8.bed + HapMap_3_r3_8.bim + HapMap_3_r3_8.fam ...
done.
可以看到,325518個位點被刪掉了,剩余1073788 個位點。
「結果文件:」
HapMap_3_r3_8.bed HapMap_3_r3_8.bim HapMap_3_r3_8.fam HapMap_3_r3_8.log感謝各位的閱讀,以上就是“qt MAF過濾的方法”的內容了,經過本文的學習后,相信大家對qt MAF過濾的方法這一問題有了更深刻的體會,具體使用情況還需要大家實踐驗證。這里是億速云,小編將為大家推送更多相關知識點的文章,歡迎關注!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。