您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這期內容當中小編將會給大家帶來有關可以做structure的R語言包LEA是怎樣的,文章內容豐富且以專業的角度為大家分析和敘述,閱讀完這篇文章希望大家可以有所收獲。
關于分群的軟件,之前寫了structure 2.3.4 軟件使用指南,軟件雖然有windows版本,但是操作太麻煩了,也寫了Admixture使用說明文檔cookbook,但是只有Linux版本,使用起來有難度。難道不能使用R語言進行structure繪圖么?結果來了:LEA!
LEA: An R package for landscape and ecological association studies
使用說明文檔
不同格式的數據使用LEA
This short tutorial explains how population structure analyses reproducing the results of the widely-used computer program structure can be performed using commands in the R language. The method works for any operating systems, and it does not require the installation
of structure or additional computer programs. The R program allows running population structure inference algorithms, choosing the number of clusters, and showing admixture coefficient bar-plots using a few commands. The methods used by R are fast and accurate, and they
are free of standard population genetic equilibrium hypotheses. In addition, these methods allow their users to play with a large panel of graphical functions for displaying pie-charts and interpolated admixture coefficients on geographic maps.
劃重點:
可以在R語言中實現軟件Structure的功能
可以做類似admixture的圖
簡單操作, 幾個命令實現相關功能
C語言開發, 可以處理大數據
install.packages(c("fields","RColorBrewer","mapplots"))
source("http://bioconductor.org/biocLite.R")
biocLite("LEA")如果安裝不成功, 也可以通過CRAN把軟件包下載到本地, 進行安裝:
install.packages("LEA_1.4.0_tar.gz", repos = NULL, type ="source")載入兩個函數, 進行格式轉化以及可視化:
source("http://membres-timc.imag.fr/Olivier.Francois/Conversion.R")
source("http://membres-timc.imag.fr/Olivier.Francois/POPSutilities.R")plink格式的ped文件, 具體格式參考:plink格式的ped和map文件及轉化為012的方法
1 SAMPLE0 0 0 2 2 1 2 3 3 1 1 2 1
2 SAMPLE1 0 0 1 2 2 1 1 3 0 4 1 1
3 SAMPLE2 0 0 2 1 2 2 3 3 1 4 1 1前六列為:
家系ID
個體ID
父本
母本
性別
表型值
SNP1-1(SNP1的第一個位點)
SNP1-2(SNP的第二個位點)
測試數據采用admixture的示例數據, 使用plink將其轉化為ped文件
library(LEA)
# 結果會生成test.geno文件的數據.
output = ped2lfmm("test.ped")# 使用LEA進行structure進行分析
library(LEA)
obj.snmf = snmf("test.geno", K = 3, alpha = 100, project = "new")
qmatrix = Q(obj.snmf, K = 3)
head(qmatrix)
barplot(t(qmatrix), col = rainbow(3), border = NA, space = 0,
xlab = "Individuals", ylab = "Admixture coefficients")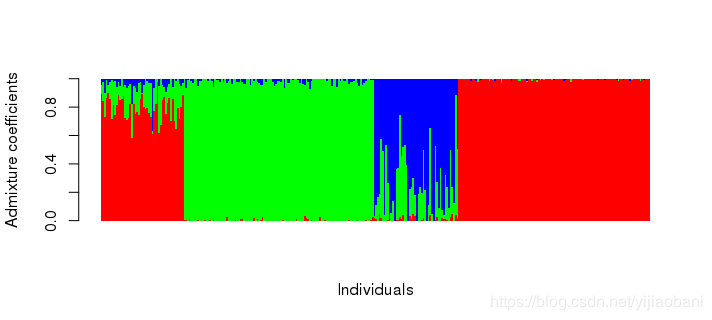
對比admixture的結果
# 對比admixture結果
qad = read.table("test.3.Q")
head(qad)
barplot(t(qad), col = rainbow(3), border = NA, space = 0,
xlab = "Individuals", ylab = "Admixture coefficients")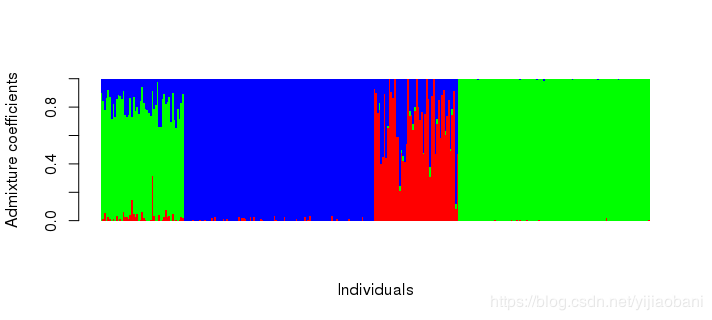
snmf選擇最優K值# 繪制折線圖, 選擇最優K值.
plot(project, col = "blue", pch = 19, cex = 1.2)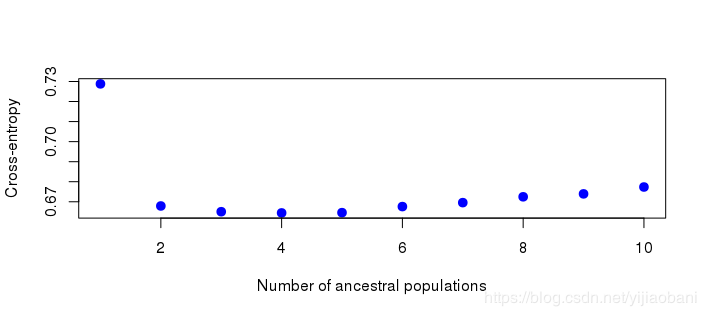
可以看出, K=3時, 最小, 因此選擇K=3.
上述就是小編為大家分享的可以做structure的R語言包LEA是怎樣的了,如果剛好有類似的疑惑,不妨參照上述分析進行理解。如果想知道更多相關知識,歡迎關注億速云行業資訊頻道。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。