您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章給大家介紹如何理解chromatin loops染色質,內容非常詳細,感興趣的小伙伴們可以參考借鑒,希望對大家能有所幫助。
利用5kb分辨率的Hi-C基因組互作圖譜,科學家識別到了chromatin loop這種染色質結構,
通過對不同分辨率的Hi-C圖譜進行分析,檢測到了不同層級的染色質結構,從高到低依次為A/B compartments, subcompartments, TAD, chromation loop。Hi-C圖譜和染色質結構模型的對應關系如下
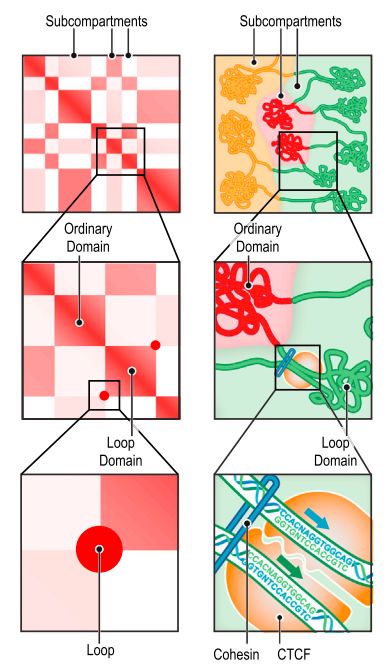
早期研究中利用1MB的Hi-C圖譜 ,定義了每條染色質包含了A和B兩個compartments。該文章中對100kb分辨率的HI-C圖譜進行聚類分析,發現A/B compartments進一步分成了6個子類,即subcompartments。對每條染色質的Hi-C圖譜進行不同算法的聚類分析,除了19號染色質外,都得到了5個cluster,對于19號染色質,得到了6個cluster。
通過分析這些subcompartments與A/B compartments的關系,發現其中2個屬于A compartments, 標記為A1和A2, 另外4個屬于B comprtmants, 標記為B1, B2, B3, B4。
對于TAD拓撲關聯結構域的識別,首創了一種ArrowHead算法,核心是對歸一化之后的交互矩陣進行變換,變化的公式如下
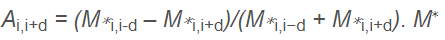
和DI的算法類似,i-d和i+d分別代表上下游的兩個bin, 如果i和上游bin互作頻率高,則A值為正數,如果和下游bin互作頻率高,則A值為負數,如果相等,則為0。示意如下
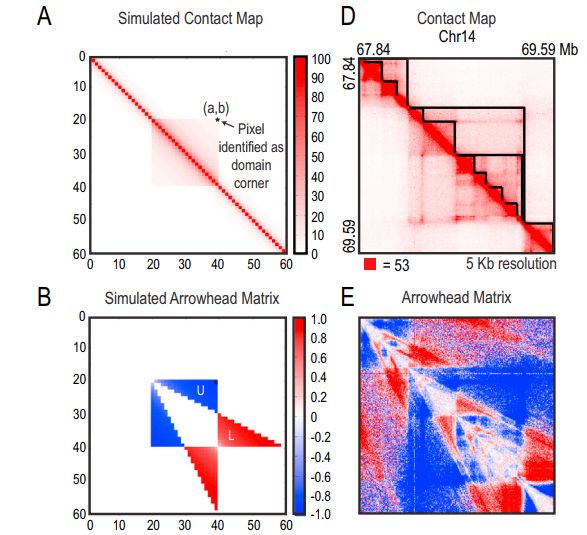
A圖中的正方形區域為一個TAD domain, 經過轉換后,變成了圖B所示的形狀。對應到整個互作矩陣上,看上去就是圖E所示的箭頭型。利用動態規劃算法,識別變換后矩陣中的箭頭區域,就可以預測TAD domain。文章中識別到的TAD domain,長度在40kb-3Mb,中位數為185kb。
對于染色質環,定義為Hi-C圖譜中互作頻率比周圍相鄰區域都高的格子區域,這樣的區域稱之為peak , 而對應的染色質區域稱之為peak loci,如下圖中藍色圓點標記的區域
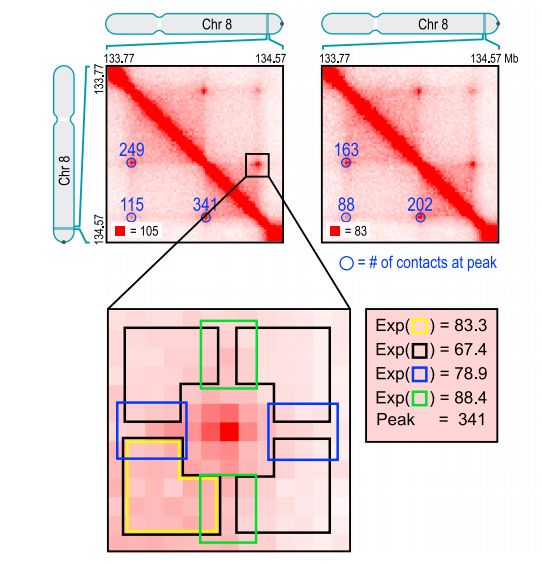
如上圖所示,通過與四周區域的交互頻率進行比較鑒定peak區域,這要去HI-C圖譜的分辨率在5kb以下。對于全基因組互作圖譜而言,這個計算量是非常大的,文章作者也提供了一種名為HiCCUPS的算法,集成在了開發的juicer軟件中。
進一步比較不同細胞系和不同物種間的染色質環區域,結果如下
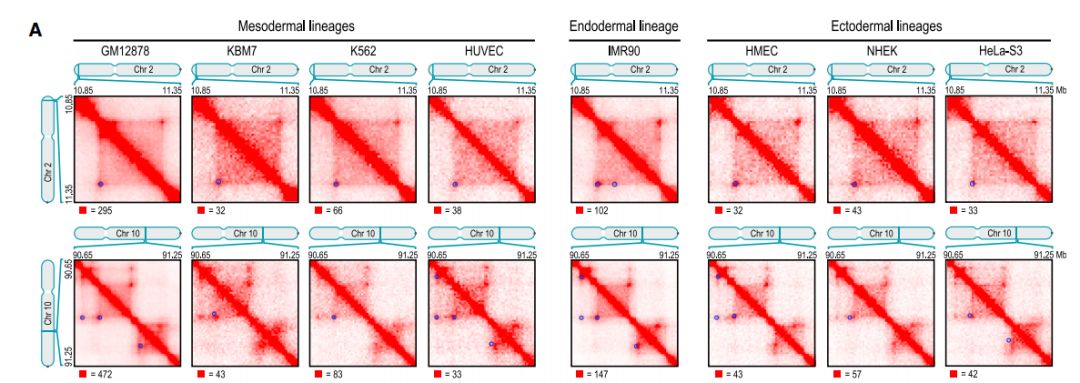
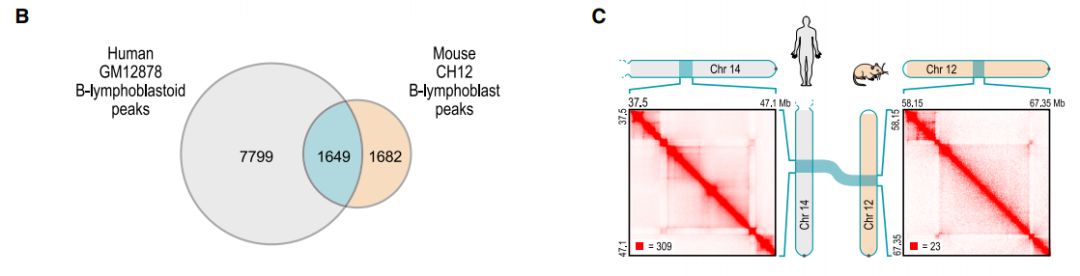
發現在不同細胞系間相對穩定,在物種間也具有一定進化保守性。對染色質環的分布位置進行分析,發現其處于TAD邊界處。進一步分析發現染色質環中有很大部分為promoter-enhancer loops, 這也解釋了增強子對靶基因的調控機制,雖然增強子與靶基因線性距離很遠,但是增強子與靶基因啟動子位于一個染色質環上,空間距離近,通過與啟動子結合調控靶基因。
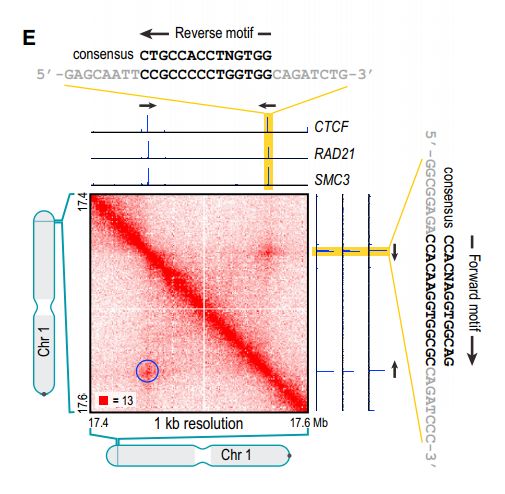
對染色質環對應區域富集的各種mark進行分析,發現其富集CTCF等轉錄因子, 如下圖所示
對于染色質環的空間結構,提出了如下模型
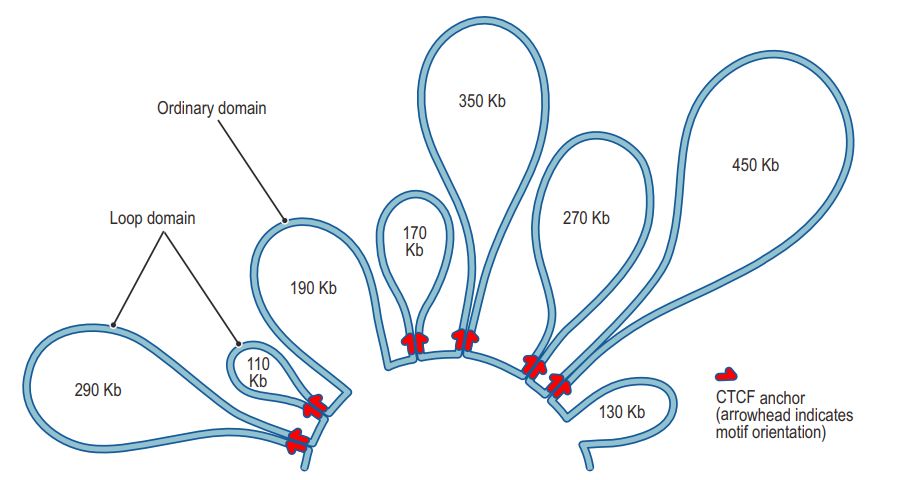
通過構建5kb以下分辨率的Hi-C圖譜,可以識別染色質環這種染色質結構單元。
關于如何理解chromatin loops染色質就分享到這里了,希望以上內容可以對大家有一定的幫助,可以學到更多知識。如果覺得文章不錯,可以把它分享出去讓更多的人看到。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。