您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
怎樣使用Mfuzz進行時間序列表達模式聚類分析,很多新手對此不是很清楚,為了幫助大家解決這個難題,下面小編將為大家詳細講解,有這方面需求的人可以來學習下,希望你能有所收獲。
在之前的文章中,我們介紹了STEM軟件,針對時間序列的數據,可以進行基因表達模式聚類分析。這里介紹另外一個功能相同的R包Mfuzz。這個R包的地址如下
https://www.bioconductor.org/packages/release/bioc/html/Mfuzz.html
Mfuzz采用了一種新的聚類算法fuzzy c-means algorithm,在文獻中稱這種聚類算法為soft clustering算法,相比K-means等hard clustering算法,一定程度上降低了噪聲對聚類結果的干擾,而且這種算法有效的定義了基因和cluster之間的關系,即基因是否屬于某個cluster, 對應的值為memebership。
對于分析而言,我們只需要提供基因表達量的數據就可以了,需要注意的是,Mfuzz默認你提供的數據是歸一化之后的表達量,這意味著表達量必須可以直接在樣本間進行比較,對于FPKM, TPM這兩種定量方式而言,是可以直接在樣本間進行比較的,但是對于count的定量結果,我們必須先進行歸一化,可以使用edgeR或者DESeq先得到歸一化之后的數據在進行后續分析。
假設你已經有歸一化之后的表達量了,通過以下幾個步驟就可以得到聚類的結果
預處理包括讀取數據,去除表達量太低或者在不同時間點間變化太小的基因等步驟,代碼如下
x <- read.table(
"normalisation.count.txt",
row.names = 1,
sep = "\t",
header = T)
count <- data.matrix(x)
eset <- new("ExpressionSet",exprs = count)
# 根據標準差去除樣本間差異太小的基因
eset <- filter.std(eset,min.std=0)需要注意的是,Mfuzz聚類時要求是一個ExpressionSet類型的對象,所以需要先用表達量構建這樣一個對象。
聚類時需要用一個數值來表征不同基因間的距離,Mfuzz中采用的是歐式距離,由于普通歐式距離的定義沒有考慮不同維度間量綱的不同,所以需要先進行標準化,代碼如下
eset <- standardise(eset)
Mfuzz中的聚類算法需要提供兩個參數,第一個參數為希望最終得到的聚類的個數,這個參數由我們直接指定;第二個參數稱之為fuzzifier值,用小寫字母m表示,可以通過函數評估一個最佳取值,代碼如下
# 聚類個數 c <- 6 # 評估出最佳的m值 m <- mestimate(eSet) # 聚類 cl <- mfuzz(eSet, c = c, m = m)
在cl這個對象中就保存了聚類的完整結果,對于這個對象的常見操作如下
# 查看每個cluster中的基因個數 cl$size # 提取某個cluster下的基因 cl$cluster[cl$cluster == 1] # 查看基因和cluster之間的membership cl$membership
代碼如下
mfuzz.plot( eSet, cl, mfrow=c(2,3), new.window= FALSE)
生成的圖片如下
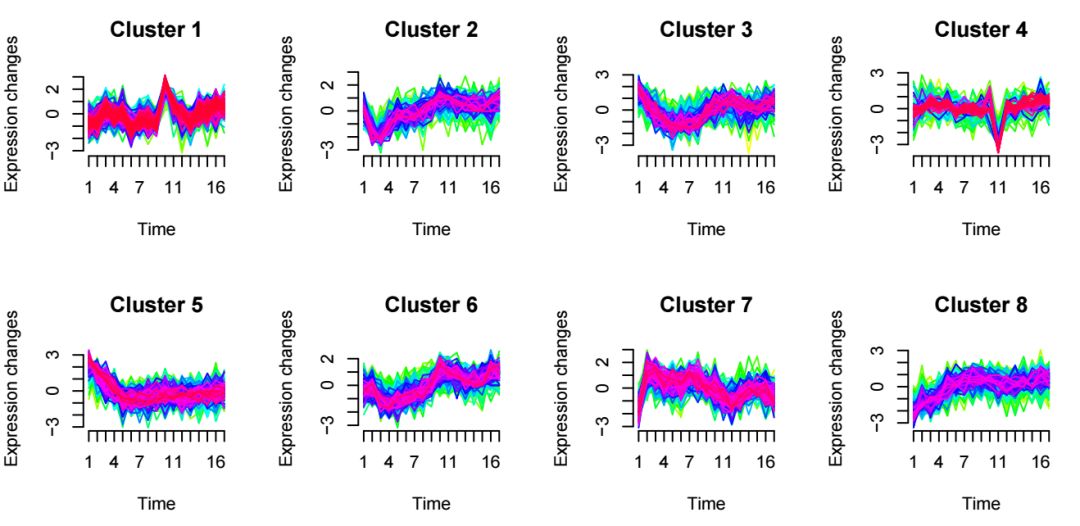
對于感興趣的表達模式,可以用上述提到的用法提取出該cluster下的基因列表,通過GO/KEGG等功能富集分析進行深入挖掘。
看完上述內容是否對您有幫助呢?如果還想對相關知識有進一步的了解或閱讀更多相關文章,請關注億速云行業資訊頻道,感謝您對億速云的支持。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。