您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要為大家展示了“MSigDB是什么數據庫”,內容簡而易懂,條理清晰,希望能夠幫助大家解決疑惑,下面讓小編帶領大家一起研究并學習一下“MSigDB是什么數據庫”這篇文章吧。
Gene Set Enrichment Analysis,中文名稱為基因集富集分析,是由Broad Institute研究所的科學家提出的一種富集方法,在提出該方法的同時還對應提供了分析的軟件GSEA和一個基因集數據庫MSigdb。本章主要介紹這個數據庫,官網如下
http://software.broadinstitute.org/gsea/msigdb/index.jsp
對于human的基因,從位置,功能,代謝途徑,靶標結合等多種角度出發,構建出了許多的基因集合,一個基因集合中就是具有相近位置或類似功能的許多基因的,Broad Institute研究所將它們構建的基因集合保存在MSigDB數據庫中。
該數據庫是不斷更新和完善的,目前最新版本為v6.2, 更新于2018年7月,共收錄了17810個基因集。不同版本如下收錄的基因集數目變化如下
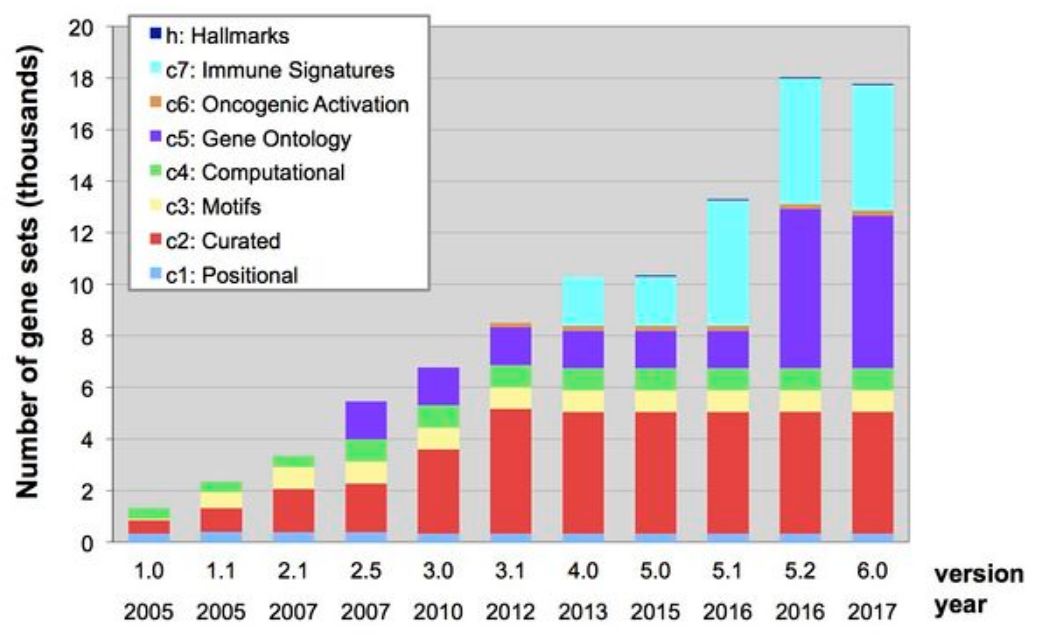
如此多的數據,肯定需要分門別類的整理,在MSigDB中,將所有的基因集劃分為以下8大類別
該類別包含了由多個已知的基因集構成的超基因集,每個H類別的基因集都對應多個基礎的其他類別的基因集。比如HALLMARK_ADIPOGENESIS對應36個基因集。
該類別包含人類每條染色體上的不同cytoband區域對應的基因集合。根據不同染色體編號進行二級分類。
該類別包含了已知數據庫,文獻和專家支持的基因集信息,包含下面5個二級分類

以KEGG為例,包含了186個基因集,每個基因集本質上都對應pathway 數據庫里的一條通路。比如基因集KEGG_ABC_TRANSPORTERS對應pathway數據庫中的hsa02010。
該類別包含了miRNA靶基因和轉錄因子結合區域等基因集合,示意如下

無論是轉錄因子還是miRNA, 都是通過特定的motif序列來識別可以結合的區域,這些基因集合,本質上為具有相同motif序列的基因集,比如AAACCAC_MIR140這個基因集, 具有相同的AAACCACmotif, 而hsa-miR-140可以識別該motif然后進行結合,所以AAACCAC_MIR140是hsa-miR-140靶標基因的集合。
該類別包含計算機軟件預測出來的基因集合,主要是和癌癥相關的基因,示意如下

該類別包含了Gene Ontology對應的基因集合,分為以下3大類別

每個基因集對應一個GO term, 比如基因集GO_MOLTING_CYCLE對應GO:0042303。
該類別包含已知條件處理后基因表達量發生變化的基因,比如AKT_UP.V1_DN對應RAD001試劑處理后表達量下調的基因。
該類別包含了免疫系統功能相關的基因集合。
在官網上,可以方便的檢索這些基因集,鏈接如下
http://software.broadinstitute.org/gsea/msigdb/genesets.jsp
選擇感興趣的類別,然后在頁面最下方就可以看到該類別下的所有基因集,示意如下

我選擇的是C1大類,2號染色體上的基因集,chr2p這種信息就是每個基因集的名字,點擊可以查看具體信息,示例如下
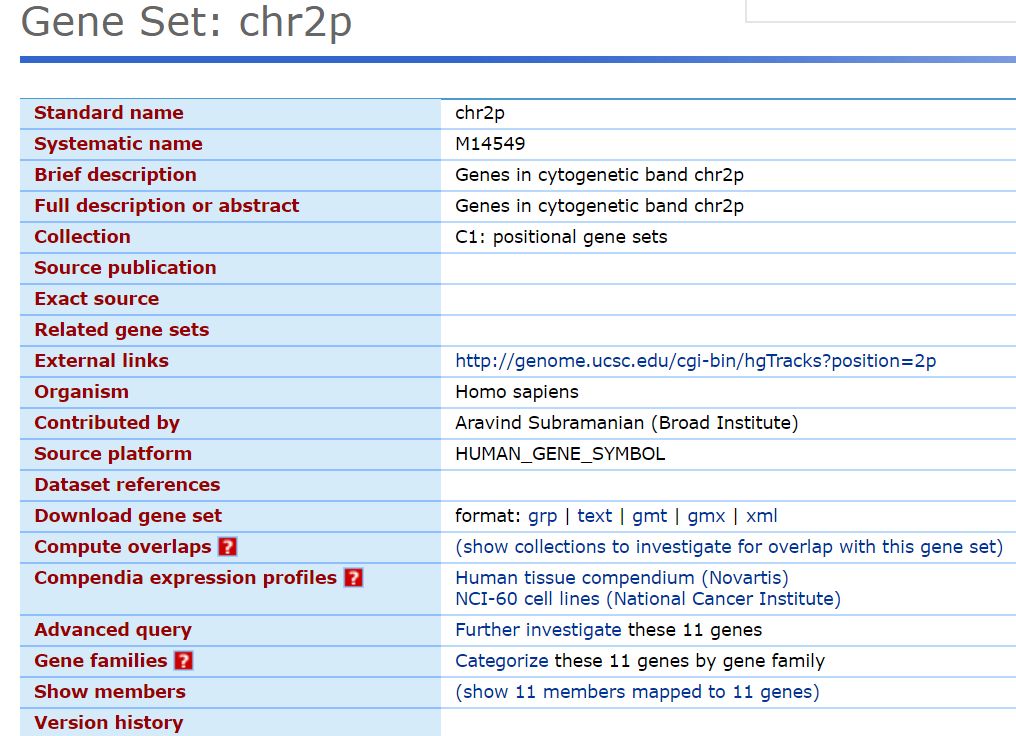
結果頁面可以看到該基因集的名稱,描述信息等,也可以直接下載,有多種格式供選擇。官網也提供了下載功能,一次下載所有的基因集,需要注冊登錄才能使用該功能。
對于GSEA而言,不僅是富集分析算法的一次提升,更是研究角度的高度升華。傳統的富集分析只會對GO, pathway等功能數據庫進行分析,而MSigDB提供了多方位的研究思路,不僅從功能出發,也可以從位置,表達量變化趨勢等角度進行探究,極大的豐富和擴展了富集分析的研究對象。
以上是“MSigDB是什么數據庫”這篇文章的所有內容,感謝各位的閱讀!相信大家都有了一定的了解,希望分享的內容對大家有所幫助,如果還想學習更多知識,歡迎關注億速云行業資訊頻道!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。