您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章主要介紹“如何使用Eagle2進行單倍型分析”,在日常操作中,相信很多人在如何使用Eagle2進行單倍型分析問題上存在疑惑,小編查閱了各式資料,整理出簡單好用的操作方法,希望對大家解答”如何使用Eagle2進行單倍型分析”的疑惑有所幫助!接下來,請跟著小編一起來學習吧!
對應的文章發表在nature genetics上,鏈接如下
https://www.nature.com/articles/ng.3679

核心算法圖示如下

對于reference haplotype, 通過PBWT轉換之后構建前綴樹,樹枝的寬度代表了單倍型的頻率,頻率越高,樹枝越寬。對于study樣本的分型結果,將可能的單倍型映射到前綴樹中,結合HMM模型來預測對應的單倍型。
和shapiet2等軟件進行比較,結果如下所示
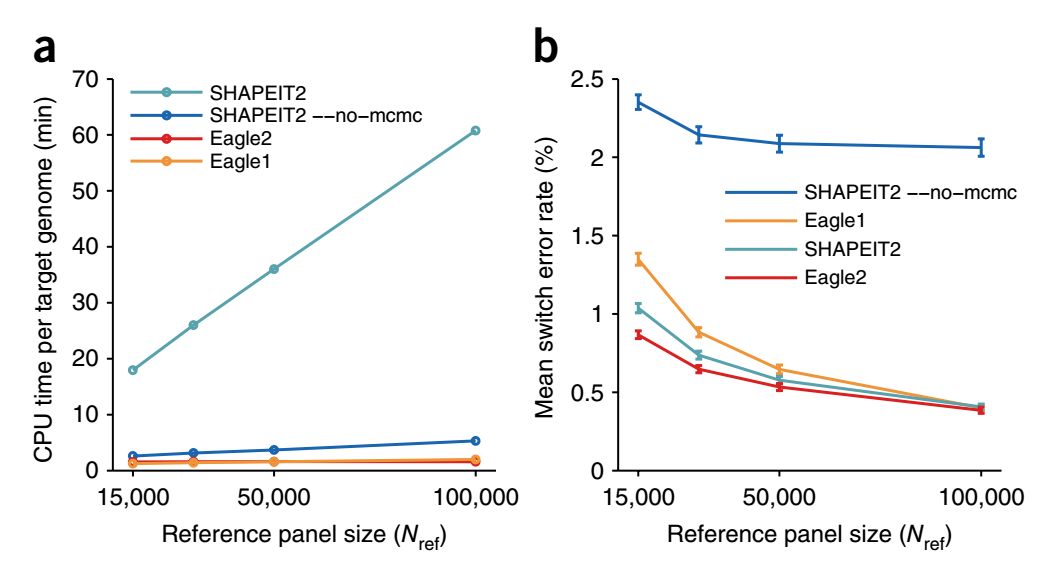
從圖a可以看出,Eagle2的運行時間最快,而且非常恒定,并不會隨著reference panel中單倍型的增多而加大運行時間, shapeit2的運行時間和panel size則基本是一個線性關系。
從圖b可以看出,panel size的增加有助于降低錯誤率,而不同軟件之間的比較可以發現,Eagle2的錯誤率最低。利用1000G和HRC兩個reference panel進行比較,可以得出相同的結論,結果如下
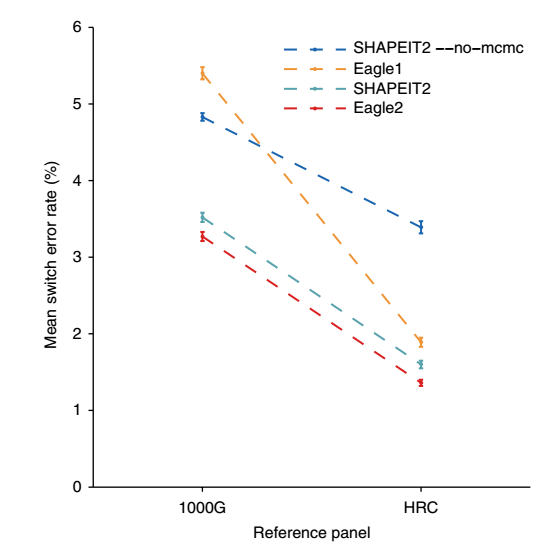
HRC的單倍型比1000G多很多,利用HRC進行phasing的錯誤率顯著降低。study樣本的對運行時間和準確率的影響如下
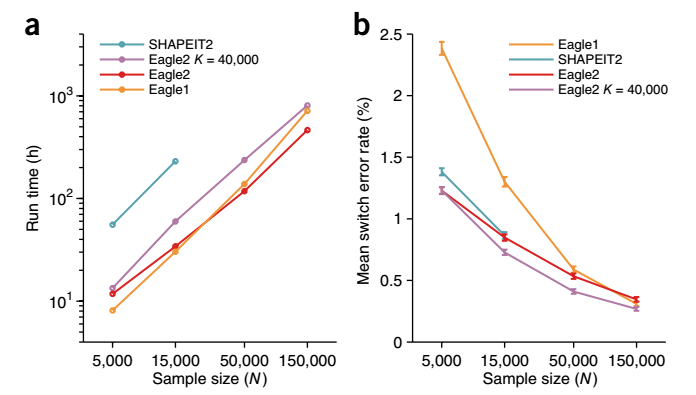
可以看到,樣本越多,運行時間越久,錯誤率越低。相比shapeit2, Eagle2的運行速度更快,錯誤率更低。
該軟件的基本用法如下
eagle \
--vcfRef HRC.r1-1.GRCh47.chr20.shapeit3.mac5.aa.genotypes.bcf \
--vcfTarget sample.chr1.vcf.gz \
--geneticMapFile genetic_map_chr1_b37.txt
--outPrefix chr1.phased \要求輸入的study樣本和reference panel的格式為VCF/BCF, 而且需要tabix的索引,如果是plink格式,可以通過plink2轉換成VCF, 官方推薦使用bcftools進行VCF和BCF的格式轉換和建立索引操作。
鑒于Eagle2運行速度和準確率的優勢,基因型填充的web服務會使用該軟件來進行phasing, 以保證運行速度和用戶體驗。
到此,關于“如何使用Eagle2進行單倍型分析”的學習就結束了,希望能夠解決大家的疑惑。理論與實踐的搭配能更好的幫助大家學習,快去試試吧!若想繼續學習更多相關知識,請繼續關注億速云網站,小編會繼續努力為大家帶來更多實用的文章!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。