您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容介紹了“如何用Python寫出心血管疾病預測模型”的有關知識,在實際案例的操作過程中,不少人都會遇到這樣的困境,接下來就讓小編帶領大家學習一下如何處理這些情況吧!希望大家仔細閱讀,能夠學有所成!
01 數據理解
數據取自于kaggle平臺分享的心血管疾病數據集,共有13個字段299 條病人診斷記錄。具體的字段概要如下:
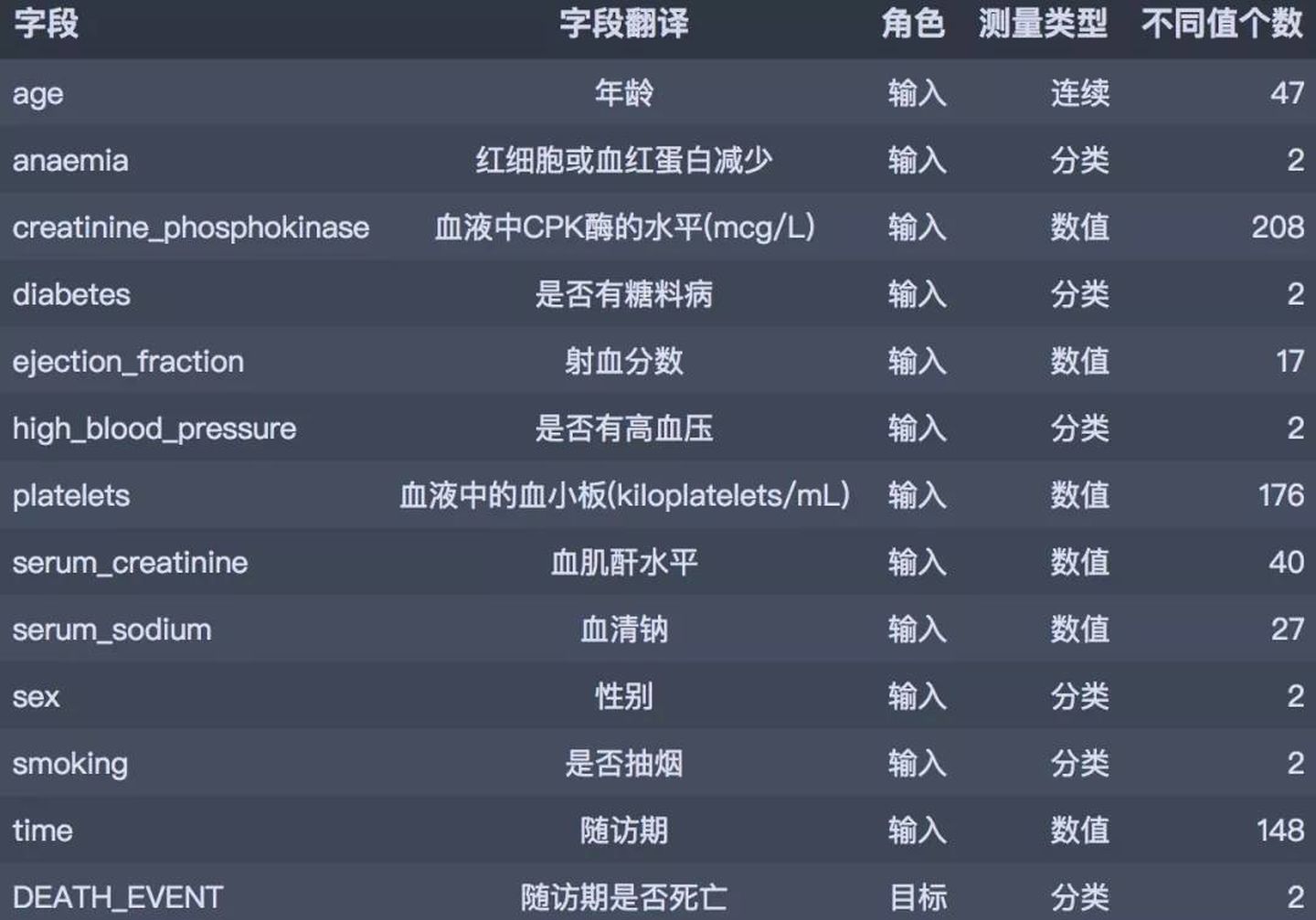
02數據讀入和初步處理
首先導入所需包。
# 數據整理 import numpy as np import pandas as pd # 可視化 import matplotlib.pyplot as plt import seaborn as sns import plotly as py import plotly.graph_objs as go import plotly.express as px import plotly.figure_factory as ff # 模型建立 from sklearn.linear_model import LogisticRegression from sklearn.tree import DecisionTreeClassifier from sklearn.ensemble import GradientBoostingClassifier, RandomForestClassifier import lightgbm # 前處理 from sklearn.preprocessing import StandardScaler # 模型評估 from sklearn.model_selection import train_test_split, GridSearchCV from sklearn.metrics import plot_confusion_matrix, confusion_matrix, f1_score
加載并預覽數據集:
# 讀入數據
df = pd.read_csv('./data/heart_failure.csv')
df.head()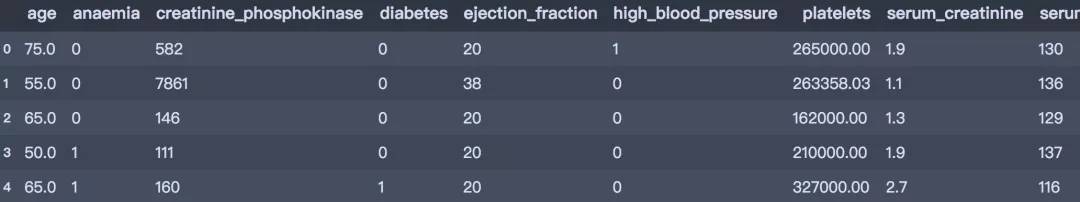
03探索性分析
1. 描述性分析
df.describe().T
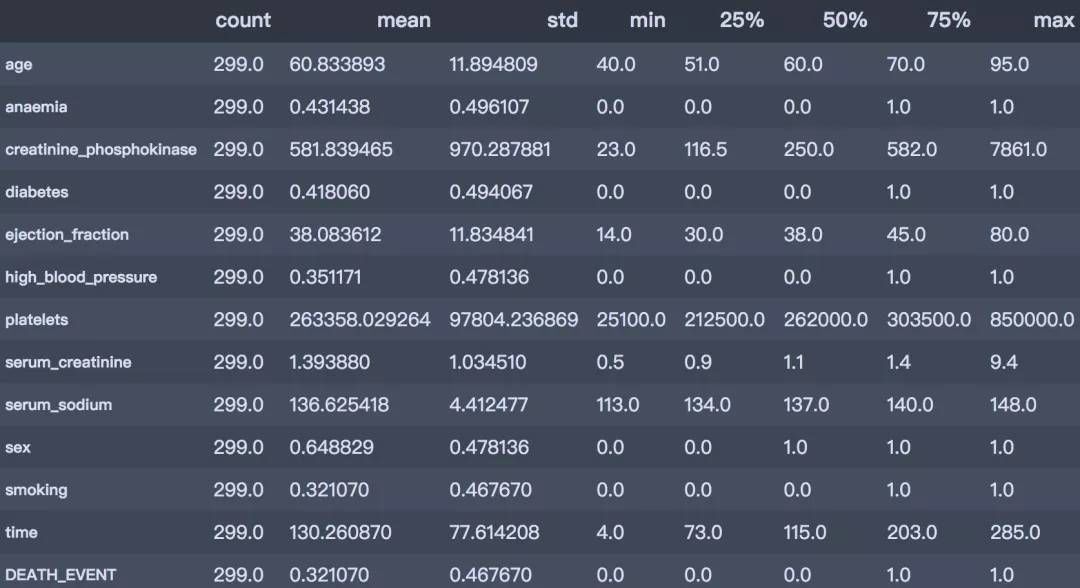
從上述描述性分析結果簡單總結如下:
是否死亡:平均的死亡率為32%;
年齡分布:平均年齡60歲,最小40歲,最大95歲
是否有糖尿病:有41.8%患有糖尿病
是否有高血壓:有35.1%患有高血壓
是否抽煙:有32.1%有抽煙
2. 目標變量
# 產生數據 death_num = df['DEATH_EVENT'].value_counts() death_num = death_num.reset_index() # 餅圖 fig = px.pie(death_num, names='index', values='DEATH_EVENT') fig.update_layout(title_text='目標變量DEATH_EVENT的分布') py.offline.plot(fig, filename='./html/目標變量DEATH_EVENT的分布.html')
總共有299人,其中隨訪期未存活人數96人,占總人數的32.1%
3. 貧血
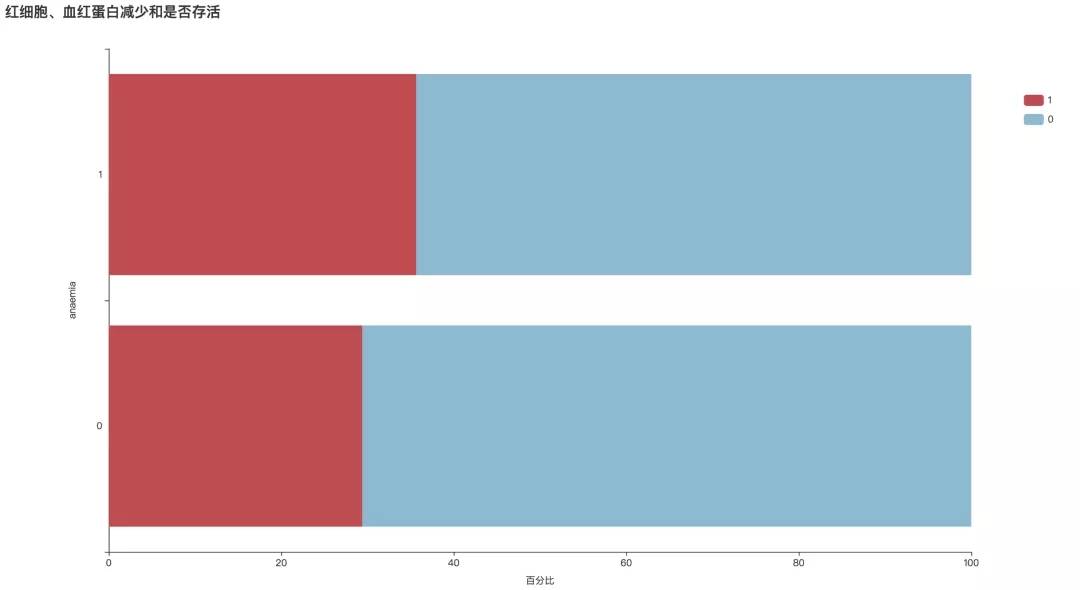
從圖中可以看出,有貧血癥狀的患者死亡概率較高,為35.66%。
bar1 = draw_categorical_graph(df['anaemia'], df['DEATH_EVENT'], title='紅細胞、血紅蛋白減少和是否存活')
bar1.render('./html/紅細胞血紅蛋白減少和是否存活.html')4. 年齡
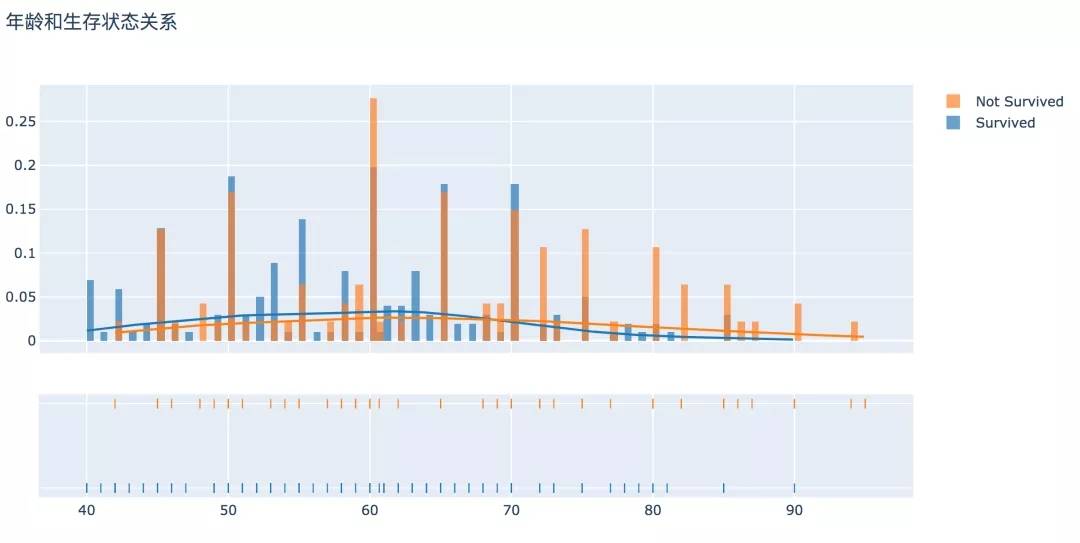
從直方圖可以看出,在患心血管疾病的病人中年齡分布差異較大,表現趨勢為年齡越大,生存比例越低、死亡的比例越高。
# 產生數據 surv = df[df['DEATH_EVENT'] == 0]['age'] not_surv = df[df['DEATH_EVENT'] == 1]['age'] hist_data = [surv, not_surv] group_labels = ['Survived', 'Not Survived'] # 直方圖 fig = ff.create_distplot(hist_data, group_labels, bin_size=0.5) fig.update_layout(title_text='年齡和生存狀態關系') py.offline.plot(fig, filename='./html/年齡和生存狀態關系.html')
5. 年齡/性別
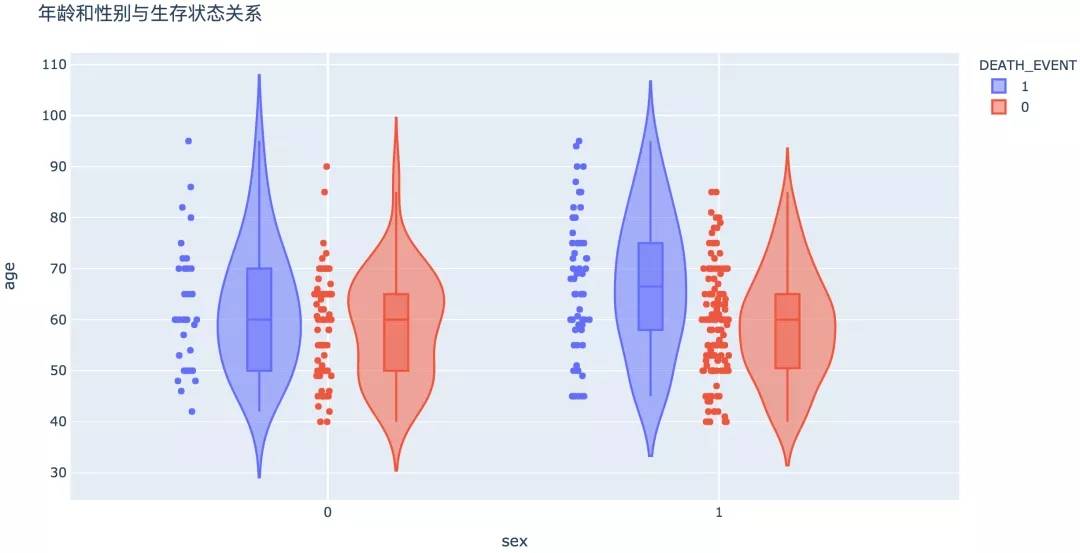
從分組統計和圖形可以看出,不同性別之間生存狀態沒有顯著性差異。在死亡的病例中,男性的平均年齡相對較高。
6. 年齡/抽煙
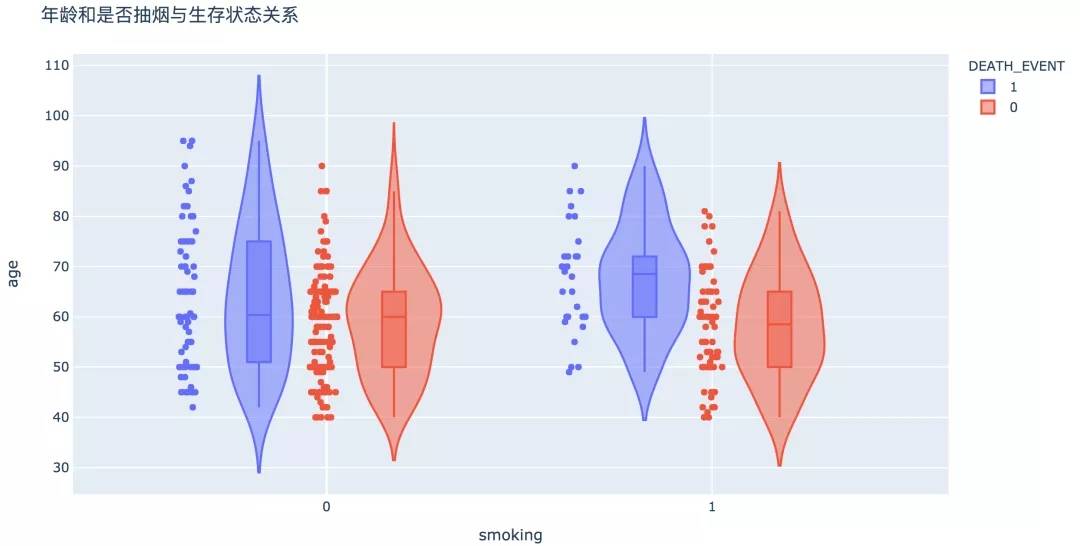
數據顯示,整體來看,是否抽煙與生存與否沒有顯著相關性。但是當我們關注抽煙的人群中,年齡在50歲以下生存概率較高。
7. 磷酸肌酸激酶(CPK)
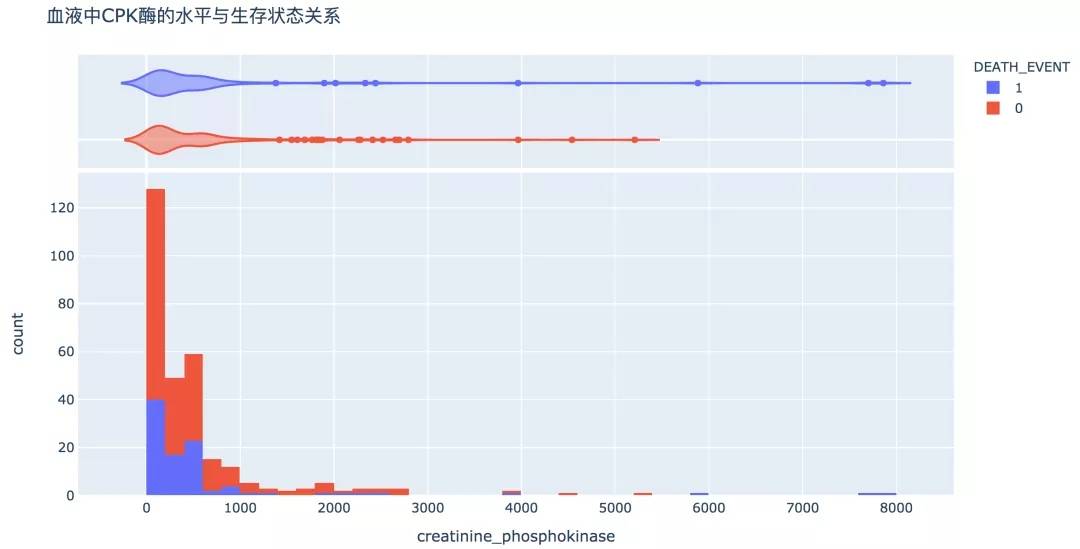
從直方圖可以看出,血液中CPK酶的水平較高的人群死亡的概率較高。
8. 射血分數
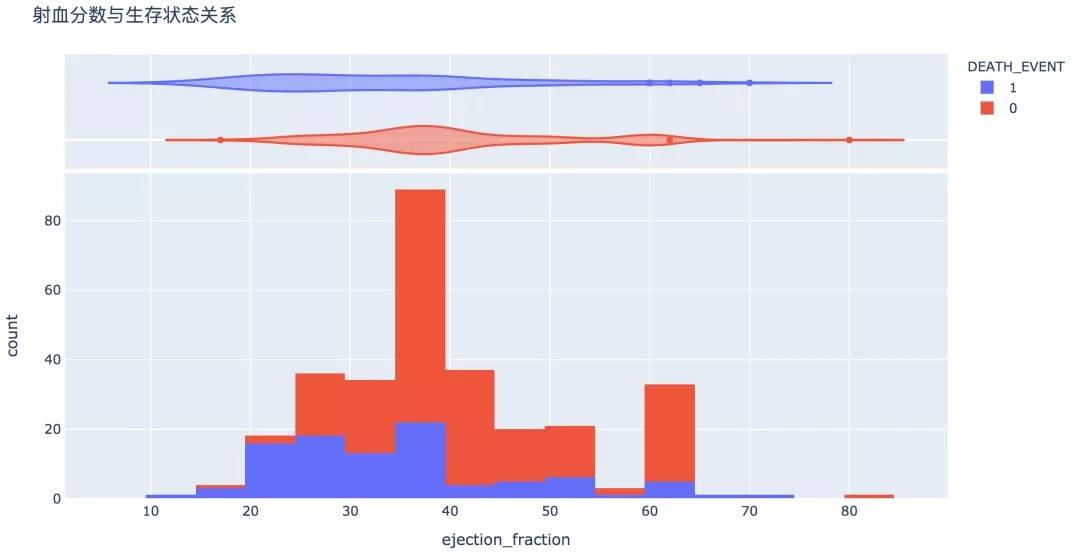
射血分數代表了心臟的泵血功能,過高和過低水平下,生存的概率較低。
9. 血小板
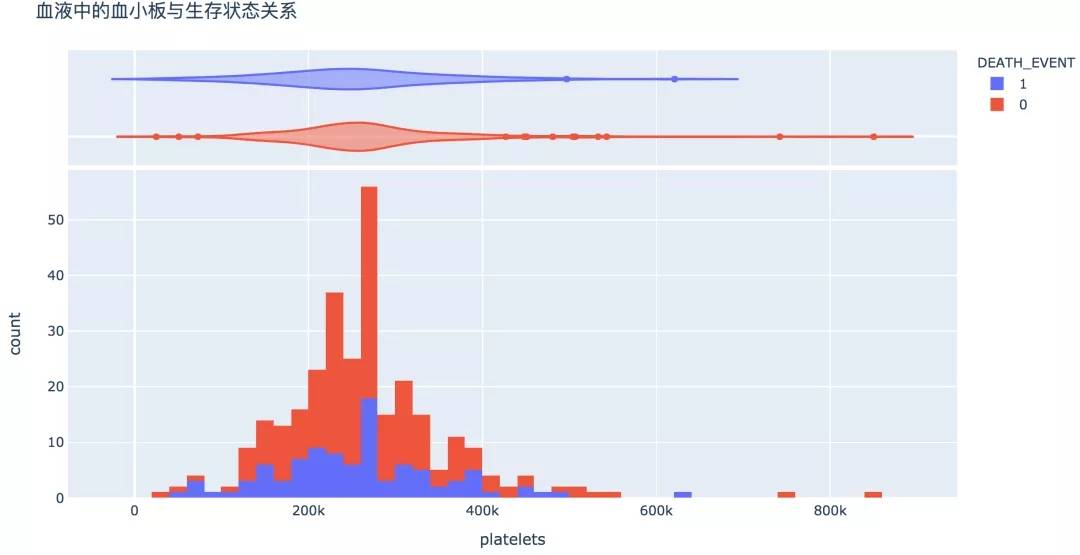
血液中血小板(100~300)×10^9個/L,較高或較低的水平則代表不正常,存活的概率較低。
10. 血肌酐水平
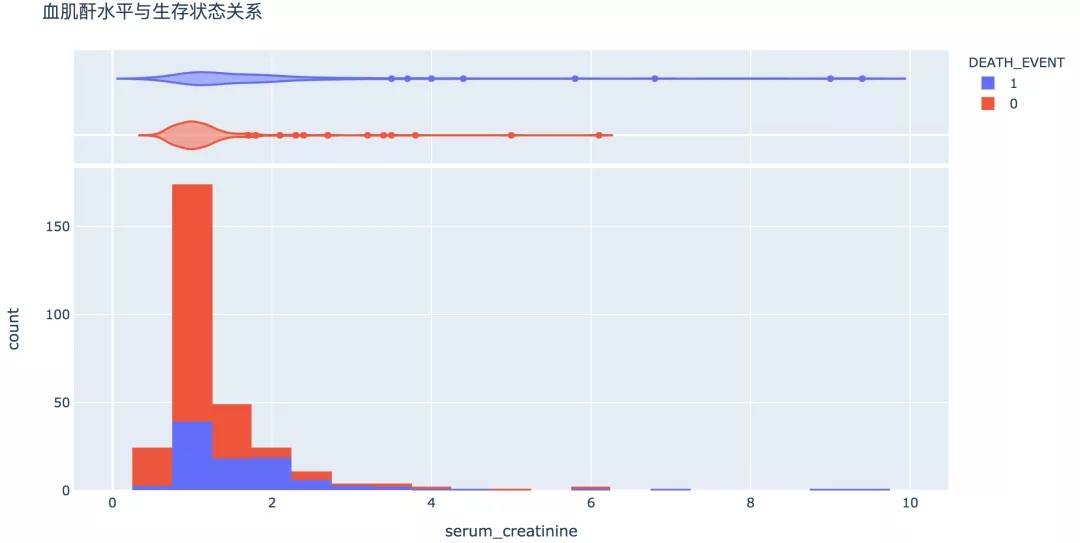
血肌酐是檢測腎功能的最常用指標,較高的指數代表腎功能不全、腎衰竭,有較高的概率死亡。
11. 血清鈉水平
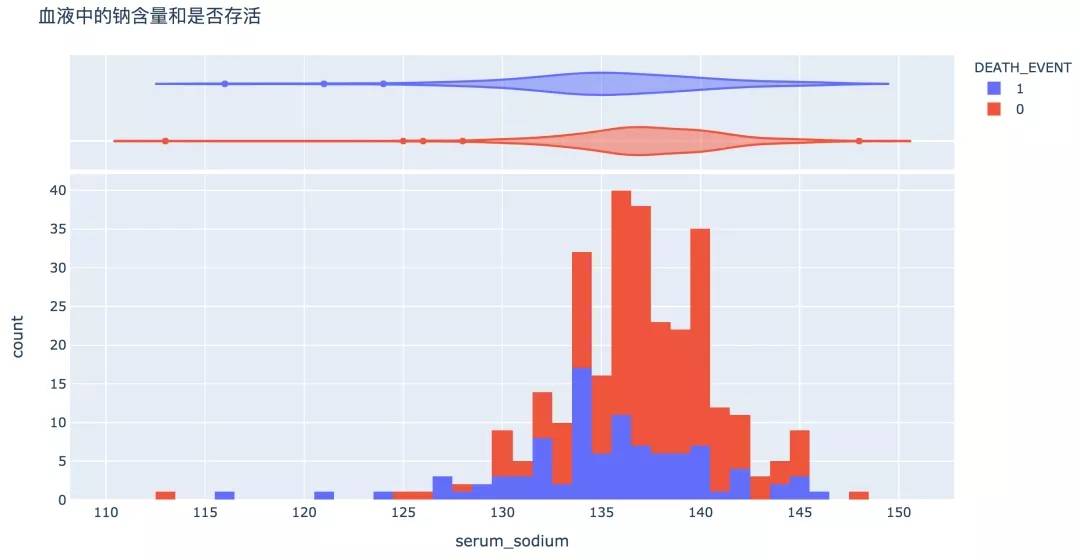
圖形顯示,血清鈉較高或較低往往伴隨著風險。
12. 相關性分析
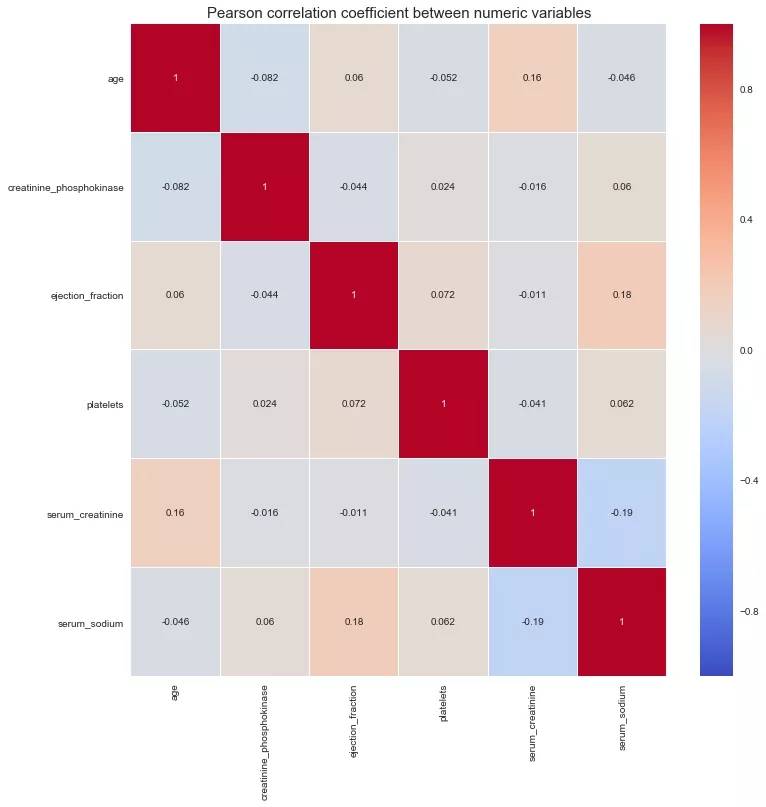
從數值型屬性的相關性圖可以看出,變量之間沒有顯著的共線性關系。
num_df = df[['age', 'creatinine_phosphokinase', 'ejection_fraction', 'platelets',
'serum_creatinine', 'serum_sodium']]
plt.figure(figsize=(12, 12))
sns.heatmap(num_df.corr(), vmin=-1, cmap='coolwarm', linewidths=0.1, annot=True)
plt.title('Pearson correlation coefficient between numeric variables', fontdict={'fontsize': 15})
plt.show()04特征篩選
我們使用統計方法進行特征篩選,目標變量DEATH_EVENT是分類變量時,當自變量是分類變量,使用卡方鑒定,自變量是數值型變量,使用方差分析。
# 劃分X和y
X = df.drop('DEATH_EVENT', axis=1)
y = df['DEATH_EVENT']
from feature_selection import Feature_select
fs = Feature_select(num_method='anova', cate_method='kf')
X_selected = fs.fit_transform(X, y)
X_selected.head()
2020 17:19:49 INFO attr select success!
After select attr: ['serum_creatinine', 'serum_sodium', 'ejection_fraction', 'age', 'time']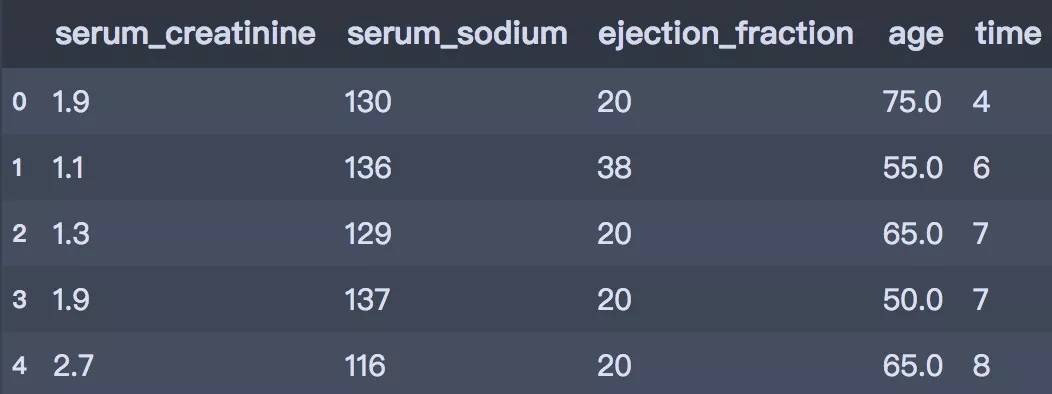
05數據建模
首先劃分訓練集和測試集。
# 劃分訓練集和測試集
Features = X_selected.columns
X = df[Features]
y = df["DEATH_EVENT"]
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, stratify=y,
random_state=2020)
# 標準化
scaler = StandardScaler()
scaler_Xtrain = scaler.fit_transform(X_train)
scaler_Xtest = scaler.fit_transform(X_test)
lr = LogisticRegression()
lr.fit(scaler_Xtrain, y_train)
test_pred = lr.predict(scaler_Xtest)
# F1-score
print("F1_score of LogisticRegression is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))我們使用決策樹進行建模,設置特征選擇標準為gini,樹的深度為5。輸出混淆矩陣圖:在這個案例中,1類是我們關注的對象。
# DecisionTreeClassifier
clf = DecisionTreeClassifier(criterion='gini', max_depth=5, random_state=1)
clf.fit(X_train, y_train)
test_pred = clf.predict(X_test)
# F1-score
print("F1_score of DecisionTreeClassifier is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))
# 繪圖
plt.figure(figsize=(10, 7))
plot_confusion_matrix(clf, X_test, y_test, cmap='Blues')
plt.title("DecisionTreeClassifier - Confusion Matrix", fontsize=15)
plt.xticks(range(2), ["Heart Not Failed","Heart Fail"], fontsize=12)
plt.yticks(range(2), ["Heart Not Failed","Heart Fail"], fontsize=12)
plt.show()
F1_score of DecisionTreeClassifier is : 0.61
<Figure size 720x504 with 0 Axes>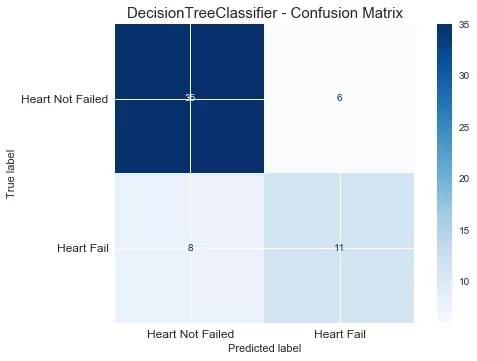
使用網格搜索進行參數調優,優化標準為f1。
parameters = {'splitter':('best','random'),
'criterion':("gini","entropy"),
"max_depth":[*range(1, 20)],
}
clf = DecisionTreeClassifier(random_state=1)
GS = GridSearchCV(clf, param_grid=parameters, cv=10, scoring='f1', n_jobs=-1)
GS.fit(X_train, y_train)
print(GS.best_params_)
print(GS.best_score_)
{'criterion': 'entropy', 'max_depth': 3, 'splitter': 'best'}
0.7638956305132776使用最優的模型重新評估測試集效果:
test_pred = GS.best_estimator_.predict(X_test)
# F1-score
print("F1_score of DecisionTreeClassifier is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))
# 繪圖
plt.figure(figsize=(10, 7))
plot_confusion_matrix(GS, X_test, y_test, cmap='Blues')
plt.title("DecisionTreeClassifier - Confusion Matrix", fontsize=15)
plt.xticks(range(2), ["Heart Not Failed","Heart Fail"], fontsize=12)
plt.yticks(range(2), ["Heart Not Failed","Heart Fail"], fontsize=12)
plt.show()
使用隨機森林
# RandomForestClassifier
rfc = RandomForestClassifier(n_estimators=1000, random_state=1)
parameters = {'max_depth': np.arange(2, 20, 1) }
GS = GridSearchCV(rfc, param_grid=parameters, cv=10, scoring='f1', n_jobs=-1)
GS.fit(X_train, y_train)
print(GS.best_params_)
print(GS.best_score_)
test_pred = GS.best_estimator_.predict(X_test)
# F1-score
print("F1_score of RandomForestClassifier is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))
{'max_depth': 3}
0.791157747481277
F1_score of RandomForestClassifier is : 0.53使用Boosting
gbl = GradientBoostingClassifier(n_estimators=1000, random_state=1)
parameters = {'max_depth': np.arange(2, 20, 1) }
GS = GridSearchCV(gbl, param_grid=parameters, cv=10, scoring='f1', n_jobs=-1)
GS.fit(X_train, y_train)
print(GS.best_params_)
print(GS.best_score_)
# 測試集
test_pred = GS.best_estimator_.predict(X_test)
# F1-score
print("F1_score of GradientBoostingClassifier is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))
{'max_depth': 3}
0.7288420428900305
F1_score of GradientBoostingClassifier is : 0.65使用LGBMClassifier
lgb_clf = lightgbm.LGBMClassifier(boosting_type='gbdt', random_state=1)
parameters = {'max_depth': np.arange(2, 20, 1) }
GS = GridSearchCV(lgb_clf, param_grid=parameters, cv=10, scoring='f1', n_jobs=-1)
GS.fit(X_train, y_train)
print(GS.best_params_)
print(GS.best_score_)
# 測試集
test_pred = GS.best_estimator_.predict(X_test)
# F1-score
print("F1_score of LGBMClassifier is : ", round(f1_score(y_true=y_test, y_pred=test_pred),2))
{'max_depth': 2}
0.780378102289867
F1_score of LGBMClassifier is : 0.74
以下為各模型在測試集上的表現效果對比:
LogisticRegression:0.63
DecisionTree Classifier:0.73
Random Forest Classifier: 0.53
GradientBoosting Classifier: 0.65
LGBM Classifier: 0.74“如何用Python寫出心血管疾病預測模型”的內容就介紹到這里了,感謝大家的閱讀。如果想了解更多行業相關的知識可以關注億速云網站,小編將為大家輸出更多高質量的實用文章!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。