您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
今天就跟大家聊聊有關如何理解CGA中的分析結果,可能很多人都不太了解,為了讓大家更加了解,小編給大家總結了以下內容,希望大家根據這篇文章可以有所收獲。
TCGA對于不同類型的數據,有著獨特的處理流程,具體如下
TCGA中的DNA測序主要用來分析腫瘤患者中的體細胞突變,和GATK的體細胞突變流程類似,前期都經過了一個預處理步驟,這里稱之為co-cleanning, 流程示意如下
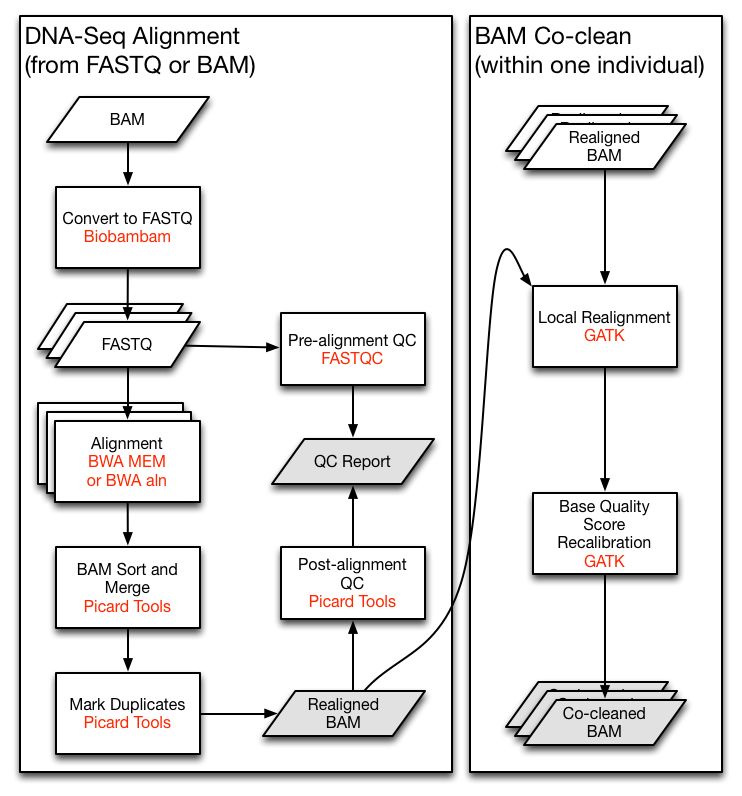
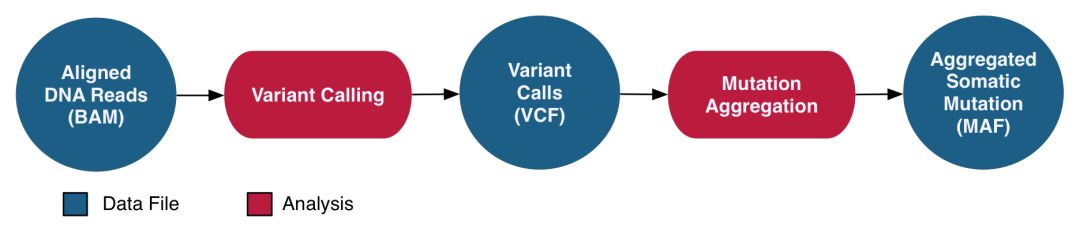
就是經典的sort->markduplicate->Realign->BQSR步驟,得到co-cleaned BAM文件。然后用配對的腫瘤和正常樣本進行somatic variant calling, 得到VCF文件。然后進行體細胞突變的注釋,得到突變注釋文件MAF, 示意如下
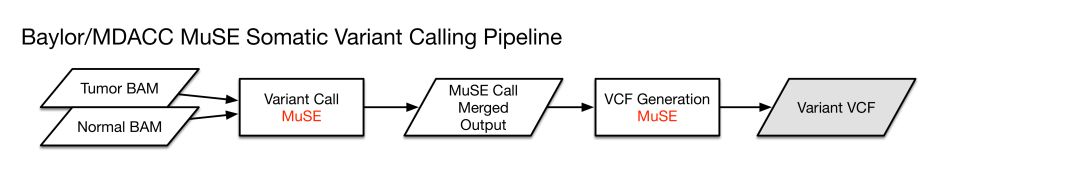
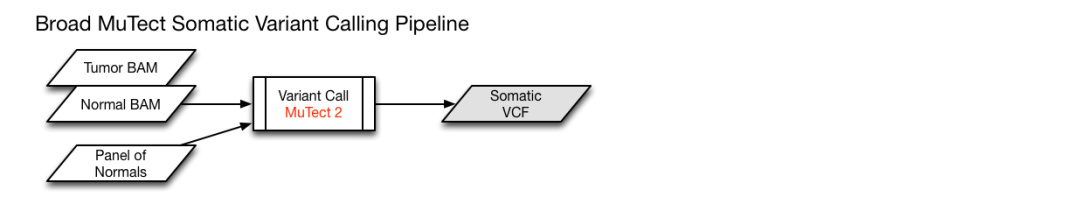
在進行體細胞突變位點分析時,使用了以下4款不同的軟件同時分析
MuSE
Mutect2
SomaticSniper
Varscan2
各自對應的pipeline示意如下
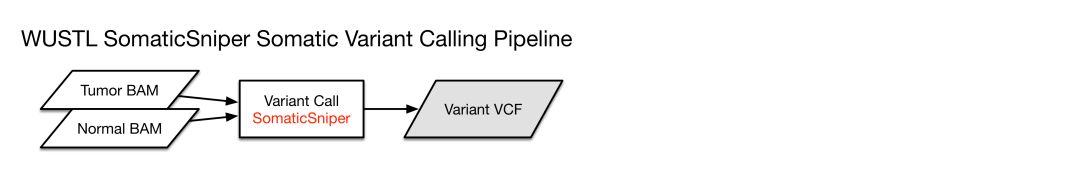
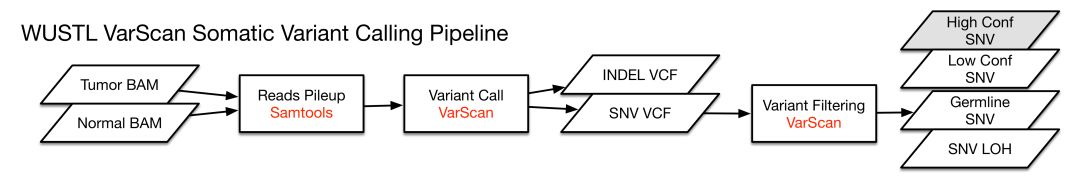
各自pipeline得到的VCF文件,使用VEP軟件對體細胞突變位點進行注釋,使用了以下數據庫進行注釋
GENCODE v.22
sift v.5.2.2
ESP v.20141103
polyphen v.2.2.2
dbSNP v.146
Ensembl genebuild v.2014-07
Ensembl regbuild v.13.0
HGMD public v.20154
ClinVar v.201601
注釋完成之后,會對突變位點進行過濾,去除低質量的突變位點和潛在的生殖細胞突變位點,剩余的位點作為最終的體細胞突變位點,保存在MAF文件中供下載。
當然對于沒有配對的正常樣本,也有tumor-only variant calling workflow來處理,具體請參考以下鏈接
https://docs.gdc.cancer.gov/Data/Bioinformatics_Pipelines/DNA_Seq_Variant_Calling_Pipeline
mRNA分析是通過STAR的2-pass模式比對hg38參考基因組,然后使用HTSeq進行定量,定量時基于Gencode V22版本的GTF文件,流程示意如下
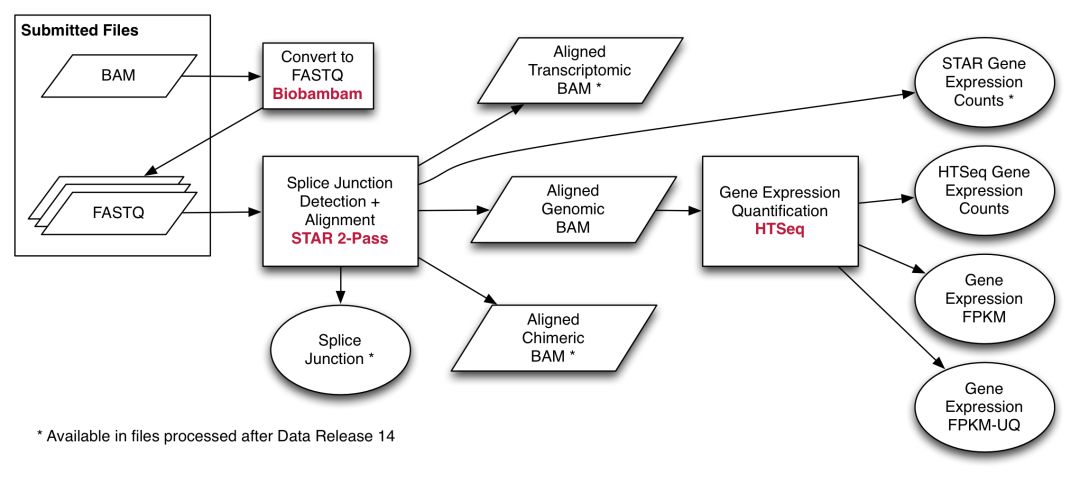
在定量時,提供了以下3種策略
Raw count
FPKM
FPKM-UQ
Raw count和FPKM是轉錄組分析中經典的定量策略,而FPKM-UQ則是在FPKM基礎上新提出的一種策略,計算公式如下
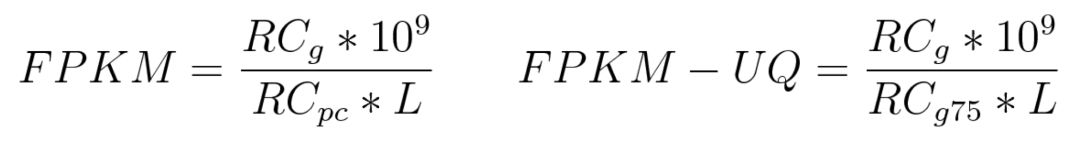
和FPKM不同的是,在FPKM-UQ中采用所有基因Mapping reads數目的上四分位數代替了所有基因Mapping Reads的總數。官方也提供了一個示例幫助我們理解具體的計算過程

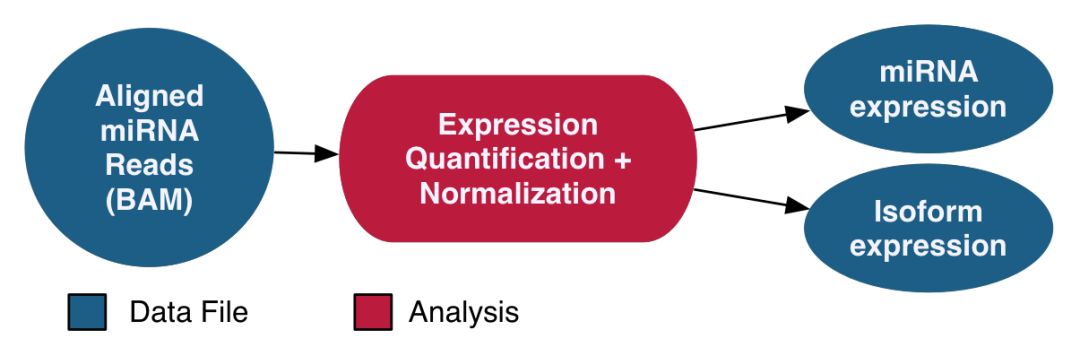
miRNA的分析采用了BCGSC開發的miRNA定量流程,這套流程只針對已知的miRNA進行定量,鏈接如下
https://github.com/bcgsc/mirna
流程示意如下
使用Affymetrix SNP 6.0芯片來分析CNV, 首先使用DNACopy這個R包來計算拷貝數,然后用GISTIC2根據CNV來評估基因的變化情況,是loss還是gain, 流程示意如下

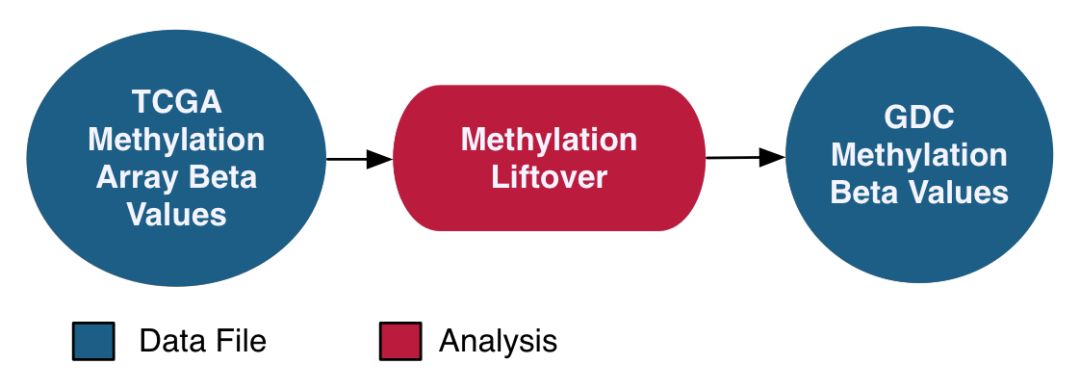
通過illumina Infinum Human Methylation 27和HumanMethylation450 兩個芯片平臺來分析DNA甲基化,采用了beta值的定量策略。同時考慮到這兩個探針是針對hg19來設計的,將探針序列與hg38進行比對,當MAPQ<10或者I型和II型探針比對到不同基因組區域時,過濾到這部分探針。剩余的CpG文件根據GENCODE V22版本的GTF來進行注釋,根據這樣的策略將hg19上的甲基化移植到hg38版本的基因組上,具體流程示意如下
了解TCGA數據分析的流程,可以更好的在GDC數據庫中篩選數據,也可以更好的和自己的數據進行比較。
看完上述內容,你們對如何理解CGA中的分析結果有進一步的了解嗎?如果還想了解更多知識或者相關內容,請關注億速云行業資訊頻道,感謝大家的支持。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。