您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
如何進行GSEA分析結果的詳細解讀,針對這個問題,這篇文章詳細介紹了相對應的分析和解答,希望可以幫助更多想解決這個問題的小伙伴找到更簡單易行的方法。
在解讀傳統的富集分析結果時,經常會有這樣的疑問,一個富集到的通路下,既有上調差異基因,也有下調差異基因,那么這條通路總體的表現形式究竟是怎樣呢,是被抑制還是激活?或者更直觀點說,這條通路下的基因表達水平在實驗處理后是上升了呢,還是下降了呢?
在這里我說下自己的觀點,在傳統的富集分析時,我們只需要一個差異基因的列表,根本不關心這個差異基因究竟是上調還是下調。這是因為,傳統的富集分析根本不需要考慮基因表達量的變化趨勢,其算法的核心只關注這些差異基因的分布是否和隨機抽樣得到的分布一致,即使后期在可視化時,我們在通路圖上用不同顏色標記了上下調的基因,但是由于沒有采用有效的統計學手段去分析這條通路下所有差異基因的總體變化趨勢,這使得傳統的富集分析結果無法回答上述的問題。
當然也有人靈光一閃,想出一個解決方案,在進行傳統的富集分析時,每次只提取上調或者下調的差異基因來進行分析,由于事先根據表達量變化趨勢對差異基因進行了篩選,從而回避了上面的問題。在我個人看來,這樣的做法有失偏頗,因為費舍爾精確檢驗就是想要證明我這個差異基因列表不是隨機抽樣得到的,而我們事先對差異基因列表的過濾已經對結果的隨機性造成了干擾,最后得出的結論其準確性也大大降低。
想象一下,上調基因和下調基因分開富集,然后富集到了同一條通路,這怎么解釋?所以在我看來,傳統的富集分析只能定位到功能,這些差異基因與哪些功能相關,而不能回答一開始的這個問題。想要回答一開始的這個問題,我們需要GSEA富集方法的結果。
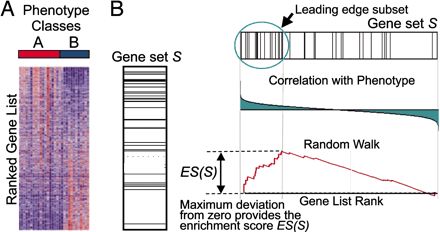
還是這張原理圖,GSEA的輸入是一個基因表達量矩陣,其中的樣本分成了A和B兩組,首先對所有基因進行排序,在之前的文章中也有提到排序的標準,這里簡單理解就是foldchange, 用來表示基因在兩組間表達量的變化趨勢。排序之后的基因列表其頂部可以看做是上調的差異基因,其底部是下調的差異基因。
GSEA分析的是一個基因集下的所有基因是否在這個排序列表的頂部或者底部富集,如果在頂部富集,我們可以說,從總體上看,該基因集是上調趨勢,反之,如果在底部富集,則是下調趨勢。
理解這個觀點之后,在來看GSEA富集分析的結果。由于結果很多,所以給出了一個匯總的html頁面。對于富集結果,根據上調還是下調分成了兩個部分,對應兩個分組,示例如下

在每個組別下富集到的基因集,從總體上看,其表達量在該組中高表達。點擊enrichment results in html,可以在網頁查看富集的結果,示例如下
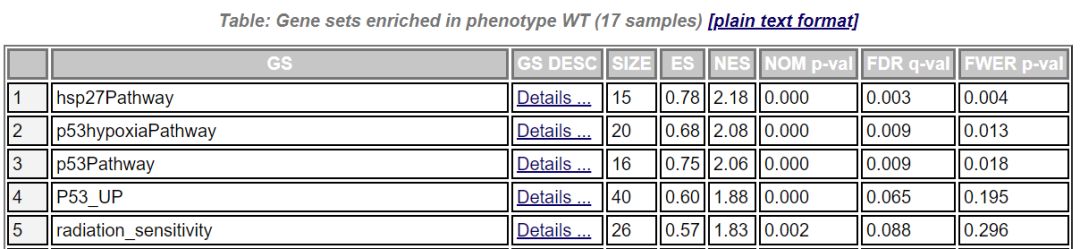
GS為基因集的名字,SIZE代表該基因集下的基因總數,ES代表Enrichment score,NES代表歸一化后的Enrichment score,NOM p-val代表pvalue,表征富集結果的可信度,FDR q-val`代表qvalue, 是多重假設檢驗矯正后的p值,注意GSEA采用pvalue < 5%, qvalue < 25% 對結果進行過濾。
點擊GS DESC可以跳轉到每個基因集詳細結果頁面,示例如下
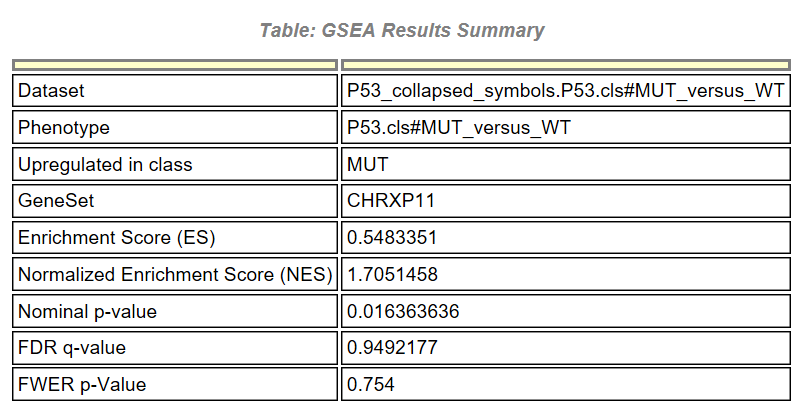
首先是一個匯總的結果,Upregulated in class說明該基因集在MUT這組中高表達,其他信息和之前介紹的一樣,除此之外,還有一個詳細的表格,示例如下
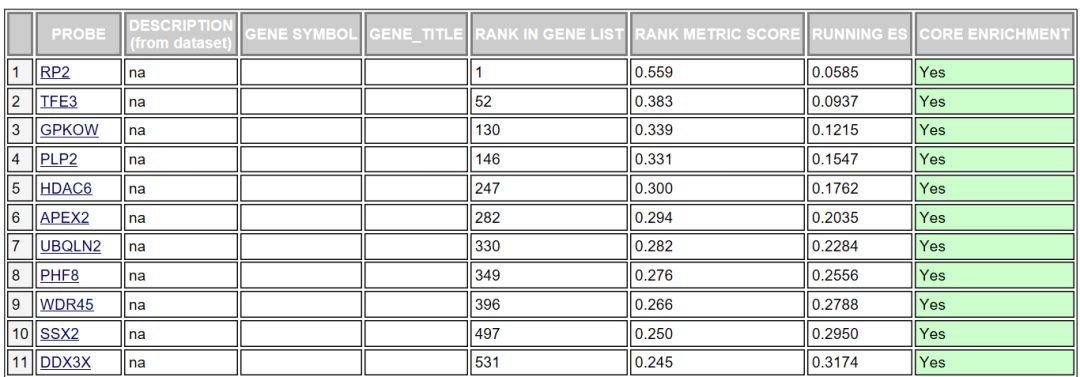
對于該基因集下的每個基因給出了詳細的統計信息,RANK IN GENE LIST代表該基因在排序號的列表中的位置, RANK METRIC SCORE代表該基因排序量的值,比如foldchange值,RUNNIG ES代表累計的Enrichment score, CORE ENRICHMENT代表是否屬于核心基因,即對該基因集的Enerchment score做出了主要貢獻的基因。這個表格中的數據對應下面這張圖
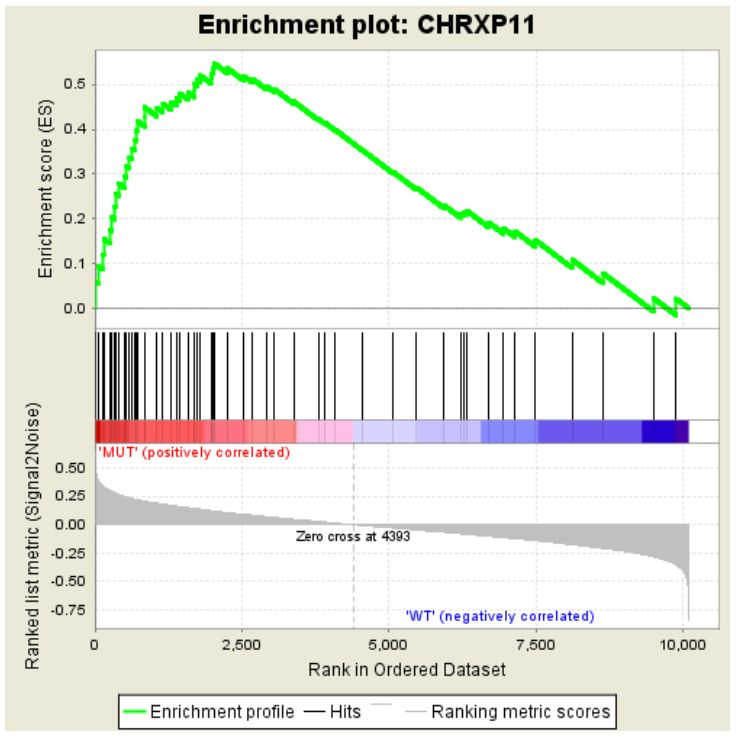
分成3個部分,第一部分為基因Enrichment Score的折線圖,橫軸為該基因下的每個基因,縱軸為對應的Running ES, 在折線圖中有個峰值,該峰值就是這個基因集的Enrichemnt score,峰值之前的基因就是該基因集下的核心基因。
第二部分為hit,用線條標記位于該基因集下的基因,第三部分為所有基因的rank值分布圖, 默認采用Signal2Noise算法,對應了縱軸的標題。
從該圖中可以看出,這個基因集是在MUT這一組高表達的,下面是一個在另一組組中高表達的示例
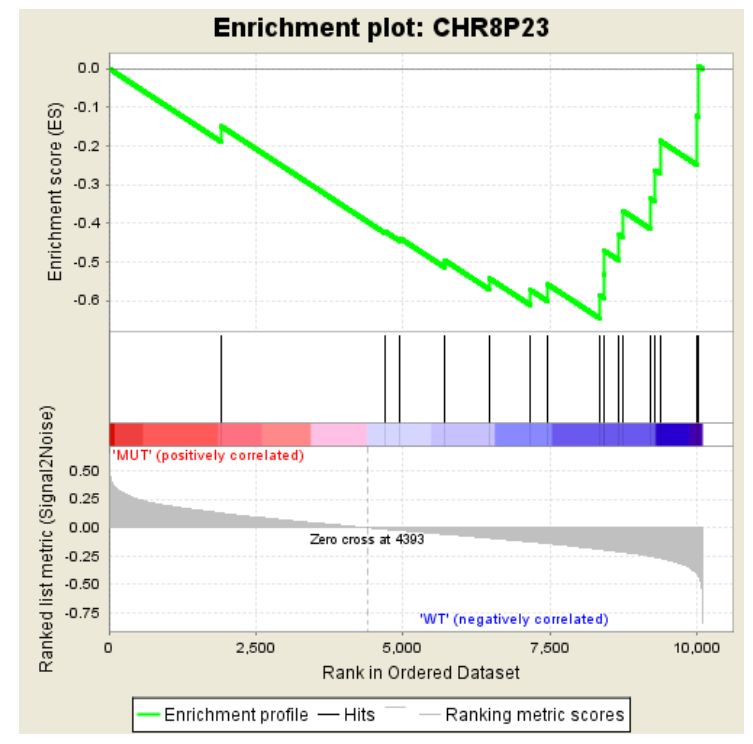
可以看到,其Enrichment score值全部為負數,對應的在其峰值右側的基因為該基因集下的核心基因。除此之外,還有一張熱圖,示例如下
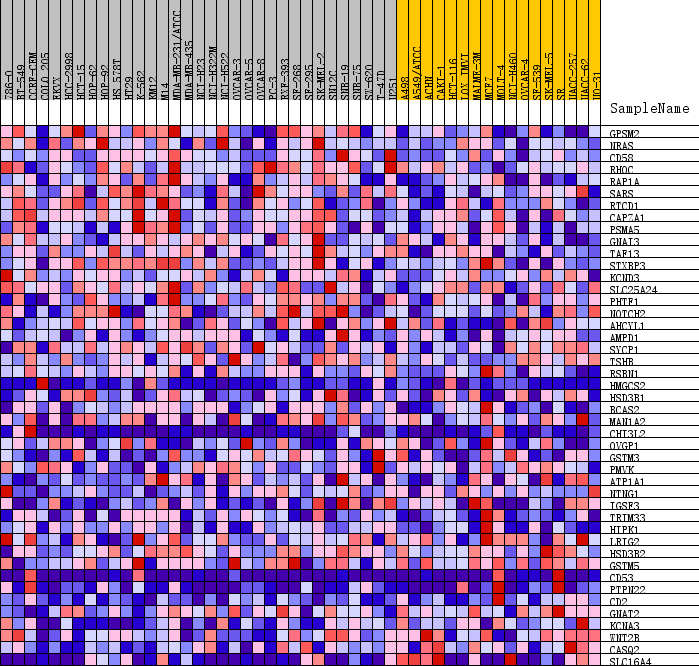
這張熱圖展示的是位于該基因集下的基因在所有樣本中表達量的分布,其中每一列代表一個樣本。每一行代表一個基因,基因表達量從低到高,顏色從藍色過渡到紅色。
在總的html頁面中,還給出了如下信息
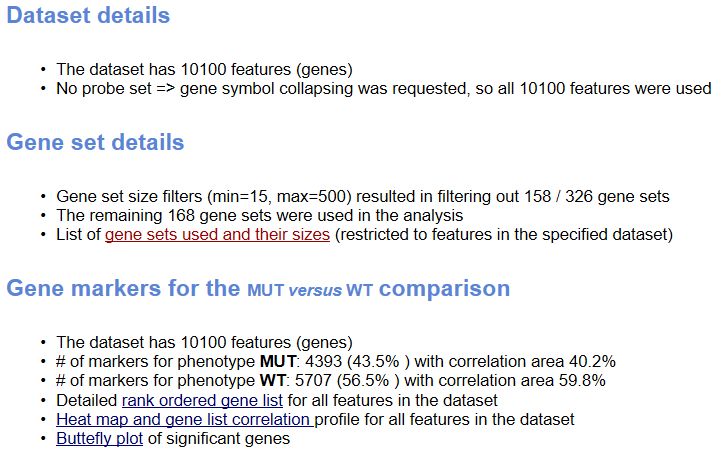
Dataset details給出了基因總數,Gene Set details給出了基因集的信息,注意軟件默認根據基因集包含的基因個數是先對基因集進行過濾,最小15個,最大500個基因,過濾掉了158個基因集,剩余的168個基因集用于分析。
Gene markers給出了排序之后的基因列表和對應的統計量rank ordered gene list,根據排序的統計量,將基因分成了兩部分,對應在每一組中高表達。
heatmap and gene list包含了所有基因表達量的熱圖和排序值的分布圖,示意如下
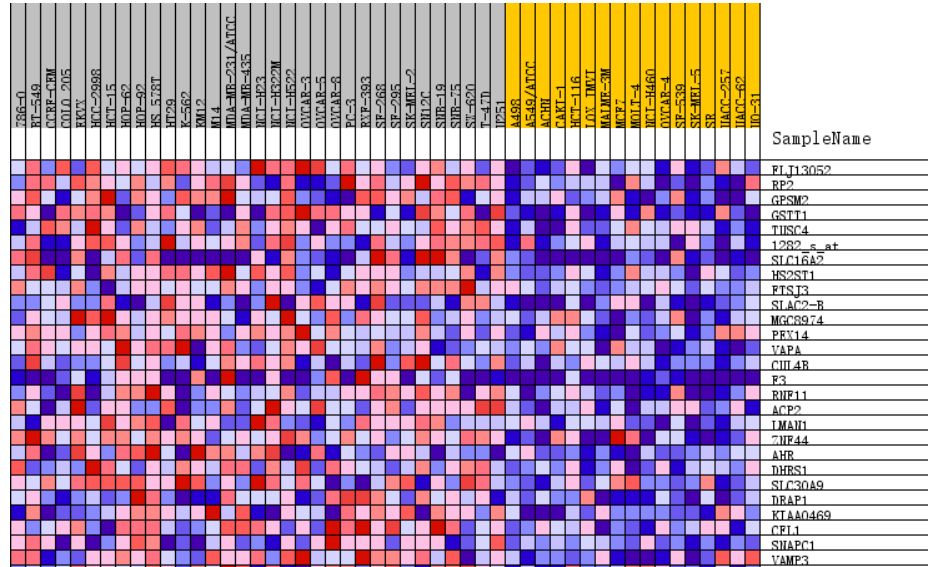
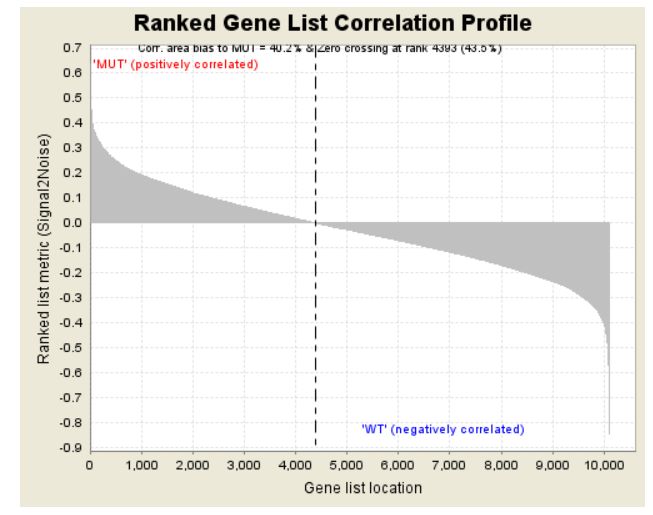
熱圖由于基因太多,截取了部分,排序值的分布圖其實就是每個基因集的Enrichment plot中的第三部分。
關于如何進行GSEA分析結果的詳細解讀問題的解答就分享到這里了,希望以上內容可以對大家有一定的幫助,如果你還有很多疑惑沒有解開,可以關注億速云行業資訊頻道了解更多相關知識。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。