您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
本篇內容介紹了“kallisto怎么使用”的有關知識,在實際案例的操作過程中,不少人都會遇到這樣的困境,接下來就讓小編帶領大家學習一下如何處理這些情況吧!希望大家仔細閱讀,能夠學有所成!
kallisto 是2016年發布的一款無須比對的轉錄本定量工具,采用了名為pseudo-alignment的算法。傳統的定量算法是根據reads的比對位置來確認其屬于哪個轉錄本或者基因,而pseudo-alignment 算法不關系reads具體的比對位置,而是通過reads的kmer特征來判斷其屬于哪一條轉錄本,示意圖如下
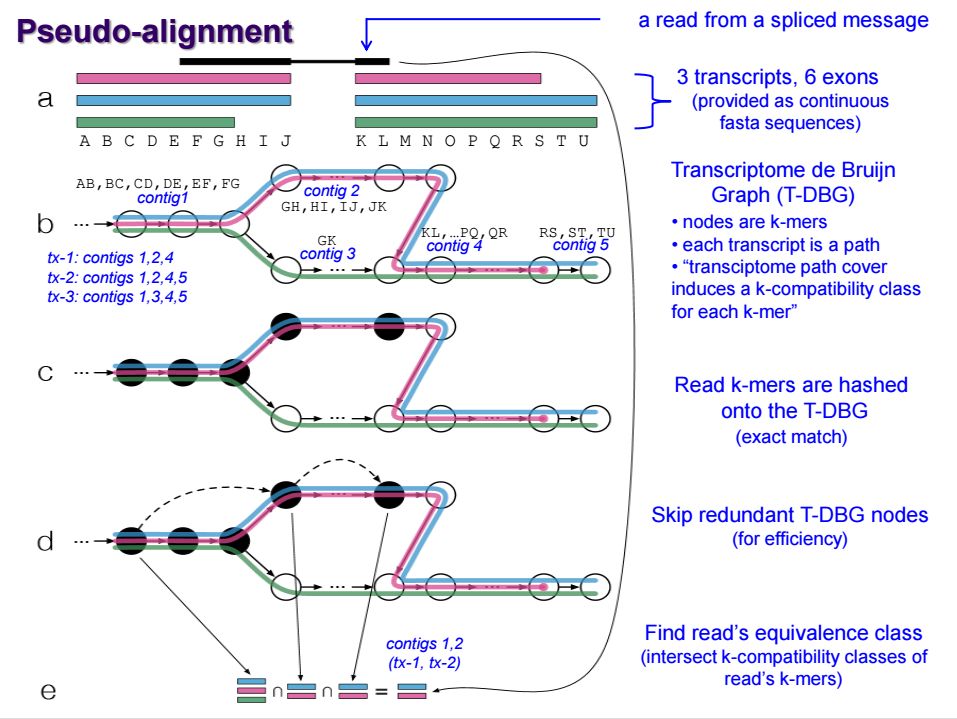
首先將每個轉錄本序列劃分為kmer, 利用所有轉錄本的kmer序列構建de Bgujin Graph, 簡稱T-DBG,在這個圖中,每個節點是一個kmer, 每條路徑代表一個轉錄本, 由于轉錄本序列的冗余,實際上每個kmer對應多條路徑,也就是對應多個轉錄本; 然后將測序的reads也劃分為kmer, 并將其映射到T-DBG中。
最終定量時,將該reads的所有kmer對應的轉錄本取交集,就能夠分析出reads可能屬于哪些轉錄本序列。
官網有編譯好的可執行文件,下載解壓即可。代碼如下
wget https://github.com/pachterlab/kallisto/releases/download/v0.44.0/kallisto_linux-v0.44.0.tar.gz tar xzvf kallisto_linux-v0.44.0.tar.gz
解壓之后,在文件夾下可以看到名為kallisto的可執行文件。從算法也可以看到,軟件的運行需要兩步,第一步對轉錄本的序列劃分kmer, 構建T-DBG, 也稱之為建索引;第二步對reads 定量。
kallisto支持讀取gzip壓縮的轉錄本序列,用法如下
kallisto index -k 31 -i hg19.idx hg19.refMrna.fa
只需要提供轉錄本的fasta格式的序列即可。-k參數指定kmer的長度,-i參數指定輸出的索引的名字,注意kallisto建立的索引為一個文件。
kallisto 支持單端和雙端數據的定量,雙端數據用法如下
kallisto quant \ -i hg19.idx \ -o out_dir \ -t 20 \ R1.fastq.gz R2.fastq.gz
-i參數指定轉錄本的索引文件,-o參數指定輸出結果的目錄,-t參數指定線程數,kallisto支持gzip壓縮的序列文件。
單端數據用法如下
kallisto quant \ -i hg19.idx \ -o output \ --single \ -l 180 \ -s 20 \ -t 20 \ reads.fastq.gz
對于單端數據而言,必須指定fragment長度的均值和方差,分別對應-l和-s參數。
在輸出目錄,會生成以下3個文件
├── abundance.h6 ├── abundance.tsv └── run_info.json
run_info.json 文件為JSON格式,保存了運行的命令和參數。
前綴為abundance 的文件,保存了轉錄本的定量信息。其中h6為HDF5格式的文件,當轉錄本數量較多時,相比純文本,這種格式的文件大小會小很多;tsv為純文本的文件,內容如下
| target_id | length | eff_length | est_counts | tpm |
|---|---|---|---|---|
| NR_103451 | 865 | 664.449 | 9 | 0.493026 |
| NM_001243523 | 577 | 376.636 | 31 | 2.99591 |
| NR_038931 | 2432 | 2231.4 | 36.9964 | 0.603491 |
對于HDF5的文件,可以采用如下命令轉換為tsv格式的文件
kallisto h6dump -o out_dir abundance.h6
-o參數指定輸出結果的目錄,最終生成的文件名稱為abundance.tsv。
“kallisto怎么使用”的內容就介紹到這里了,感謝大家的閱讀。如果想了解更多行業相關的知識可以關注億速云網站,小編將為大家輸出更多高質量的實用文章!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。