您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章給大家分享的是有關QUAST有什么用的內容。小編覺得挺實用的,因此分享給大家做個參考,一起跟隨小編過來看看吧。
對于不同kmer或者不同軟件的基因組組裝結果,我們通常會通過N50等指標來進行評估。
對于一個組裝出來的序列,不論是contig還是scaffold, 首先將各個序列根據長度從大到小排序,然后從第一個序列開始,將長度進行累加,直到累加的長度超過了總長度的50%,此時,最后一個累加的contig的長度就是N50的長度。示意圖如下
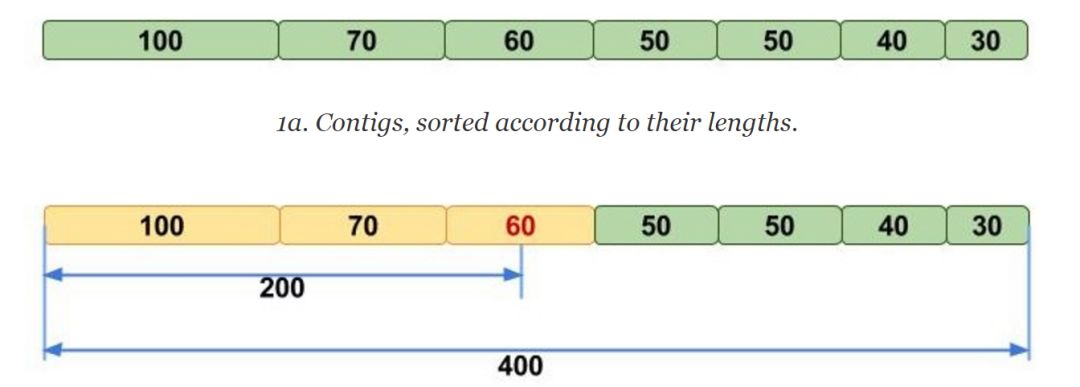
上圖中N50的長度就是60,和N50的概念類似,還有N75, N90等說法,這些指標可以統稱為Nx。Nx越大,說明組裝出來的片段長度越長一定程度上,證實了組裝結果越好。
除了Nx指標外,還有Lx指標,比如L50,指的是累加的contig的個數,示意圖如下
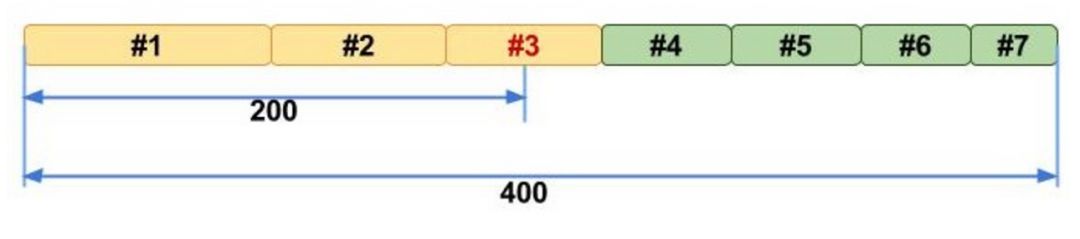
上圖中L50的值是3。在實際分析中,我們可以通過現有軟件來計算N50, L50等指標,quast就是最常用的軟件之一。該軟件有在線服務,鏈接如下
http://quast.bioinf.spbau.ru/
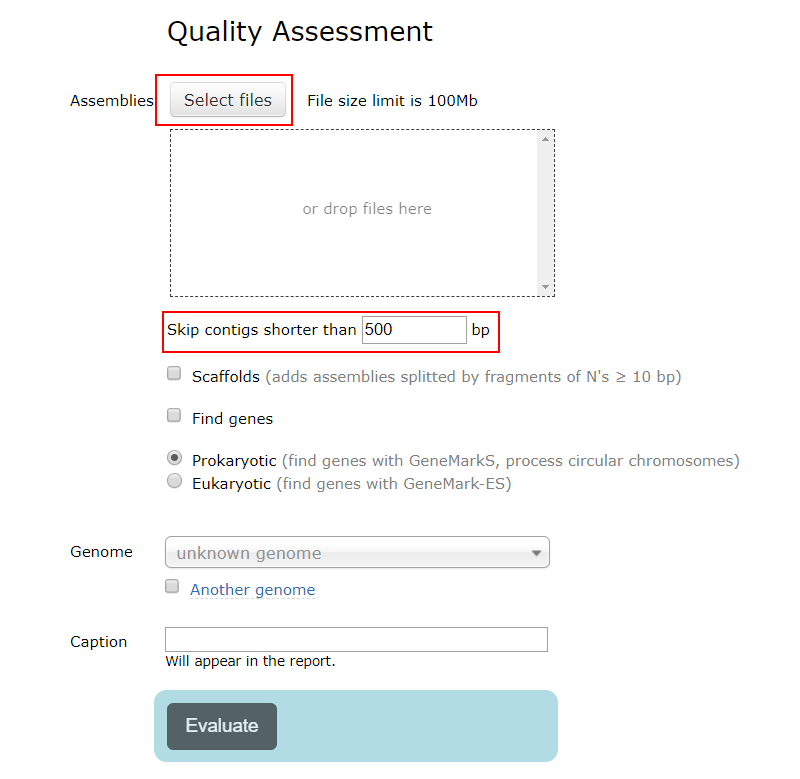
只需要上傳fasta格式的contig或者scaffold序列,然后提交即可。在線服務雖然方便,但是也是有限制的,上傳的fasta文件大小不能夠超過100Mb,對于實際的基因組項目而言,當是不能滿足要求。此時,可以下載軟件到本地服務器,然后運行。
安裝過程如下
wget https://sourceforge.net/projects/quast/files/quast-4.6.3.tar.gz tar xzvf quast-4.6.3.tar.gz cd quast-4.6.3/
quast基于python開發,以來matplotlib庫進行繪圖,保證python和matplotlib安裝好,然后直接下載源代碼,解壓縮就可以使用了。
用法如下
python quast.py -t 10 -o test1_out contigs.fasta
-t參數指定線程數,-o參數指定輸出結果的目錄。運行完成后,輸出目錄會生成如下文件
├── basic_stats ├── icarus.html ├── icarus_viewers ├── quast.log ├── report.html ├── report.tex ├── report.tsv ├── report.txt ├── transposed_report.tex ├── transposed_report.tsv └── transposed_report.txt
直接看report.html文件就就可以了。
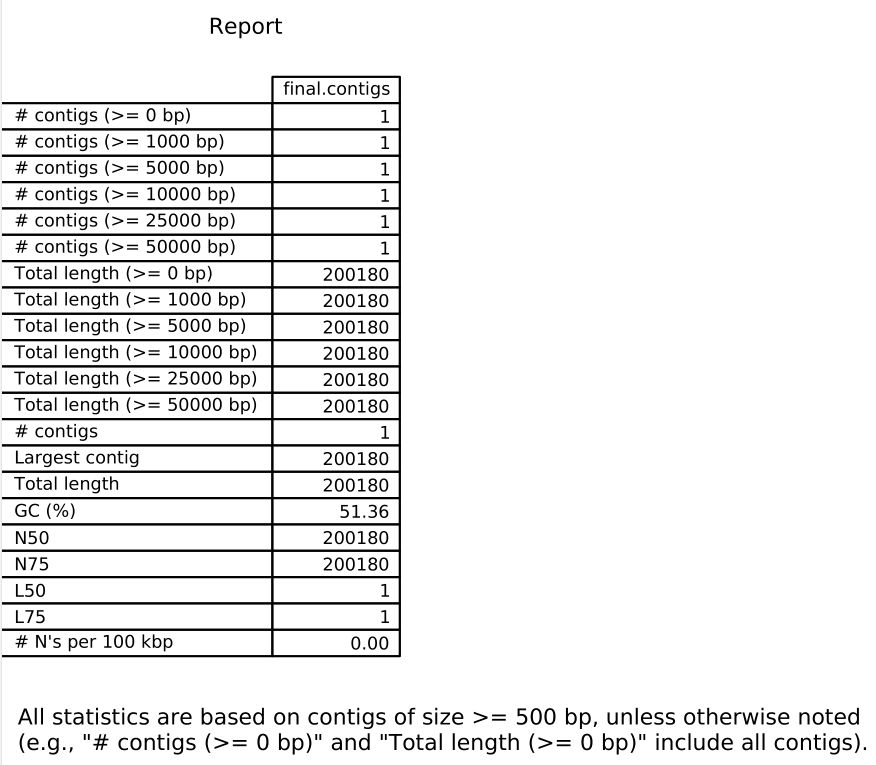
quast 會統計不同長度的contig的個數,以及N50,L50等指標,示例結果如下
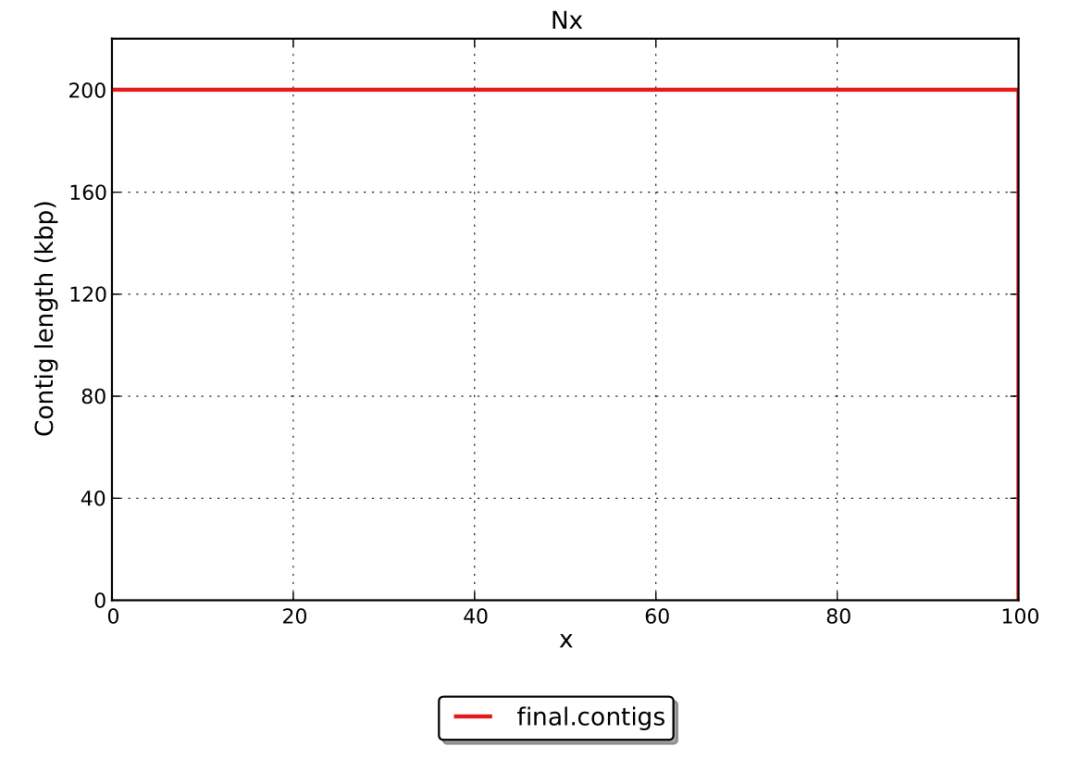
橫坐標為Nx,縱坐標為Nx的值,示意圖如下
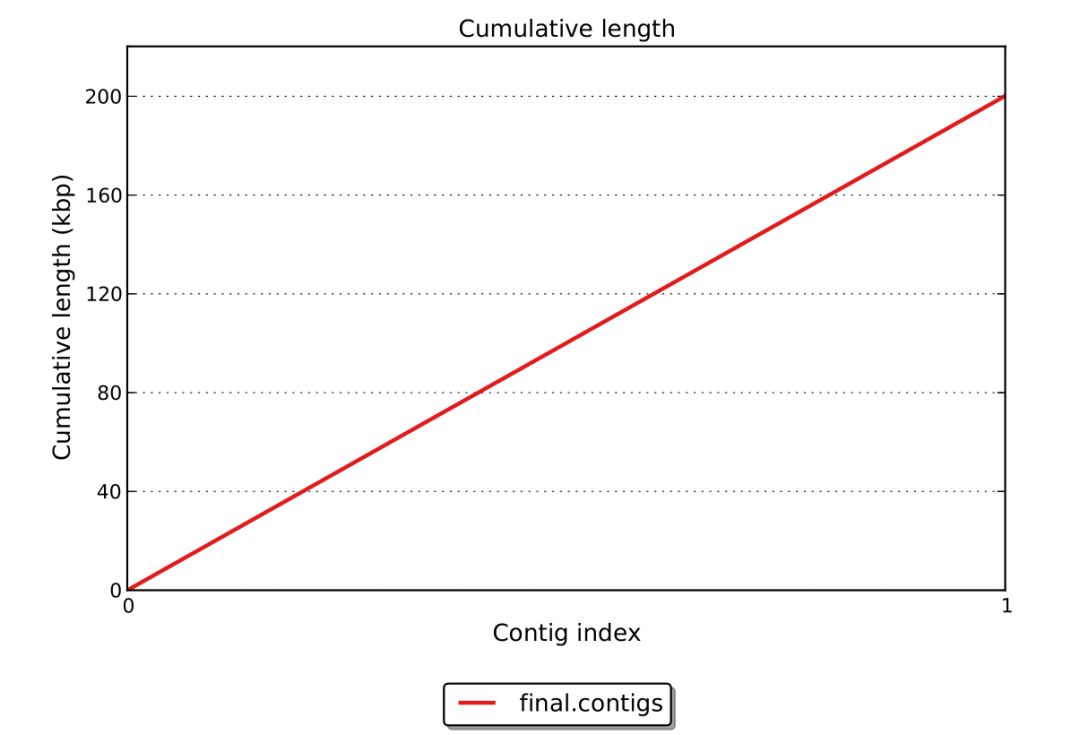
橫坐標為contig個數,縱坐標為累加的長度,示意圖如下
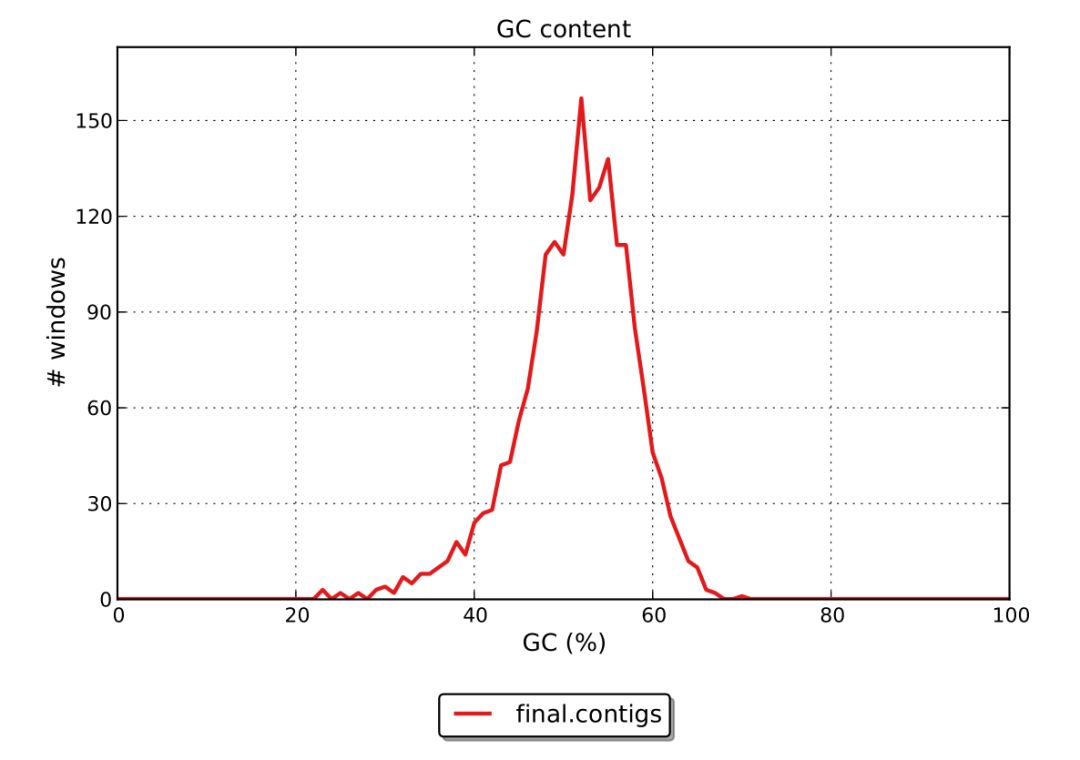
窗口的GC含量分布圖,quast將每個contig劃分為長度100bp的窗口,統計每個窗口的GC含量, 橫坐標為GC含量,縱坐標為窗口個數, 示意圖如下
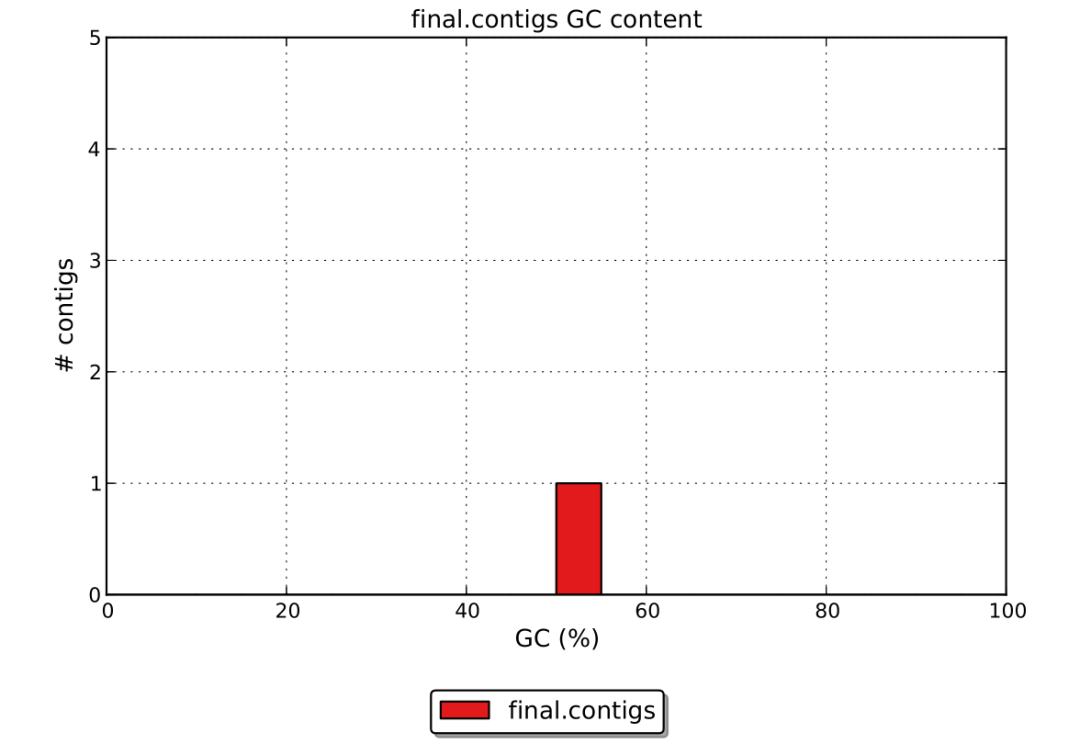
contig GC含量分布圖,對于每個contig,統計GC含量,橫坐標為GC含量,縱坐標為contig個數,示意圖如下
感謝各位的閱讀!關于“QUAST有什么用”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,讓大家可以學到更多知識,如果覺得文章不錯,可以把它分享出去讓更多的人看到吧!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。