您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
這篇文章給大家分享的是有關SSRIT是什么工具的內容。小編覺得挺實用的,因此分享給大家做個參考,一起跟隨小編過來看看吧。
微衛星microsatellite, 又叫做簡單重復序列(simple sequence repeats, SSR)或者短串聯重復序列(short tandem repeats, STR), 指的是以2到10bp的短序列為單位,重復出現多次所構成的DNA序列。
重復的最小單位稱之為motif, 示例如下
agagagagagag
上述片段就是一段SSR序列,motif為ag, 重復出現了6次。
微衛星DNA種類多,分布廣,在基因組中平均50bp就有一個重復序列;在不同種族,不同人群中重復單位和重復次數都大不相同,構成了SSR遺傳多態性。
SSRIT是一款識別簡單重復系列的軟件,官網如下:
http://archive.gramene.org/db/markers/ssrtool
該軟件的官網提供了在線服務,用法如下

參數a用于選擇motif的最小長度,可選范圍為2到10bp; 參數b用于選擇最小重復次數,建議最小重復次數為5以上。
在文本框中,輸入fasta格式的序列,然后點擊右下角的FIND SSRs提交即可。
輸出結果如下
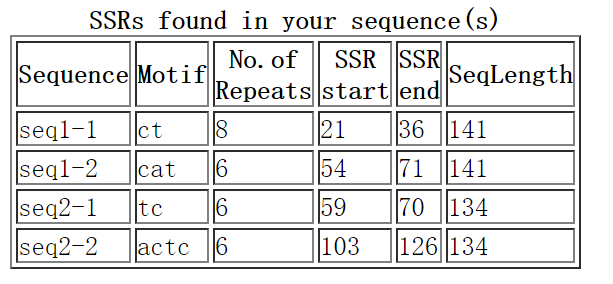
第一列為SSR區域的ID,由序列標識符和數字編號構成,第二列為Motif的堿基序列,第三列為重復次數,第四列和第五列對應SSR區域的起始和終止位置,第六列為輸入序列的總長度。
也可以下載腳本本地運行,安裝過程如下
wget ftp://ftp.gramene.org/pub/gramene/archives/software/scripts/ssr.pl
該軟件采用perl語言開發,直接下載對應的perl腳本就可以了,這個perl腳本寫的是比較簡陋的,并沒有提供幫助文檔之類的信息。從源代碼可以看出,用法如下
perl ssr.pl input.fasta > ssr.txt
只需要提供fasta格式的輸入文件就可以了,一次可以提供多個fasta文件,示例文件如下
>seq1 agagattaggatcgatcgcgctctctctctctctctcgatcgagatcgat ggccatcatcatcatcatcattgagatatagcgcgatatcgagagatctc agaatagatatcgcgctatagagagatcgagagagagtaga >seq2 agagataggaatatgagatagcgggggggggggggcgctatacgcgctcg gagagagatctctctctctcttatagagatcgatcgactagctagatata agactcactcactcactcactcactcagcgcgat
輸出結果通過重定向保存在ssr.txt文件中,該文件的內容如下
seq1 1 3 cat 6 54 71 141 seq2 1 4 actc 6 103 126 134
輸出內容和在線服務基本一致,第2列和第三列不需要看。需要注意的是,同樣的輸入文件,在線服務識別到了4個SSR區域,而本地版只識別到了2個,這個是因為參數設定不同。
本地版的motif長度和重復序列次數只能通過修改源代碼實現,對應代碼如下
my @specs = ([2,9], #dinucl. with >= 9 repeats [3,6], #trinucl. with >= 6 repeats [4,5]); #tetranucl. with >= 5 repeats
默認情況下,對于2bp的motif, 要求最小重復次數為9次,ct這個motif只出現了8次,所以過濾掉了,如果想要和官網保持一致,可以修改源代碼,然后再次運行就可以了。
感謝各位的閱讀!關于“SSRIT是什么工具”這篇文章就分享到這里了,希望以上內容可以對大家有一定的幫助,讓大家可以學到更多知識,如果覺得文章不錯,可以把它分享出去讓更多的人看到吧!
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。