您好,登錄后才能下訂單哦!
您好,登錄后才能下訂單哦!
haploview怎樣進行連鎖不平衡分析,很多新手對此不是很清楚,為了幫助大家解決這個難題,下面小編將為大家詳細講解,有這方面需求的人可以來學習下,希望你能有所收獲。
haploview 是基于圖形界面的軟件,其界面設計良好,用法簡單,是進行連鎖不平衡分析的主流軟件之一。
需要兩個輸入文件, 后綴分別為ped和info。ped文件保存的是樣本的基因分型結果,這種格式在之前的文章中詳細介紹過了;info文件保存的是SNP位點的ID和位置信息,內容如下
IGR1118a_1 274044 IGR1119a_1 274541 IGR1143a_1 286593
第一列為SNP位點的ID, 第二列為SNP位點在基因組上的位置。
點擊Linkage Format菜單,然后選擇對應的輸入文件。Data File 指定輸入的ped文件,Locus Information File 指定輸出的info文件,如果ped和info文件同名,在指定Data File的同時,程序會自動識別info文件;Ignore pairwise comparisons of markers 指定計算LD的范圍,默認只對距離在500kb以內的SNP位點分析連鎖不平衡,可以根據自己的需要進行調整,比如調整到1000Kb;Exclude individuals 對樣本進行過濾,默認基因型缺失的比例大于50%的樣本被剔除。當所有參數設置好之后,點擊OK按鈕即可。

check markers可以對輸入的SNP位點進行過濾, 可以根據HW pvalue, Min genotype, mendel error, MAF等閾值過濾。設置好相應的閾值之后,點擊Rescore Markers按鈕,就可以根據閾值過濾了,最后一列的Rating如果勾選上了,表示該SNP位點符合要求。

點擊LD plot 按鈕,就可以看到如下所示的連鎖不平衡的熱圖;每個格子代表了兩個SNP位點之間的LD分析結果,顏色從白色到紅色,代表連鎖程度從低到高。右鍵點擊格子,可以看到LD分析的詳細結果。方框中的數值為D'值,為了美觀,這里乘以了100。通過最上方的Display按鈕,可以調整熱圖的外觀。
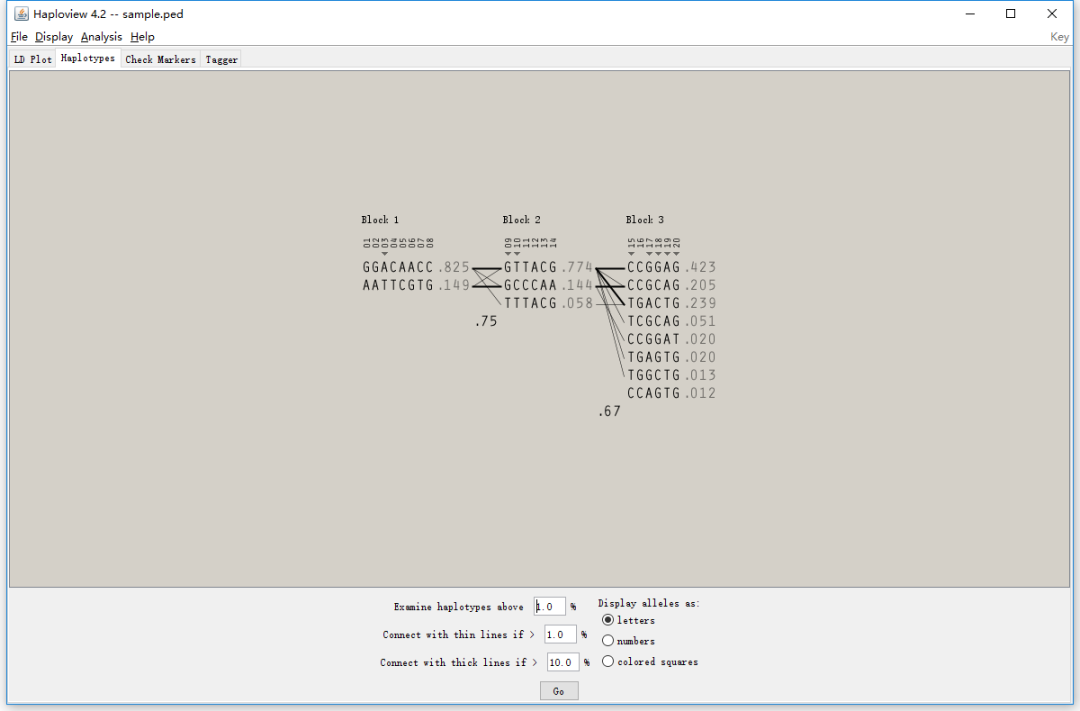
相互之間高度連鎖的SNP位點構成了haplotype block, 比如下圖中的1-8構成了block1, 長度為84kb。

基于LD分析的結果,可以去定義一個haplotype block。 通過最上方的Analysis按鈕,可以調整計算haplotype block的算法
Tagger按鈕用于挑選tagSNPs, Configuration有兩個用途,第一個是篩選SNP位點,通過勾選對應的單選框,可以指定想要進行分析的SNP位點;第二個用途是指定tagSNPs挑選的算法,默認是pairwise tagging only。
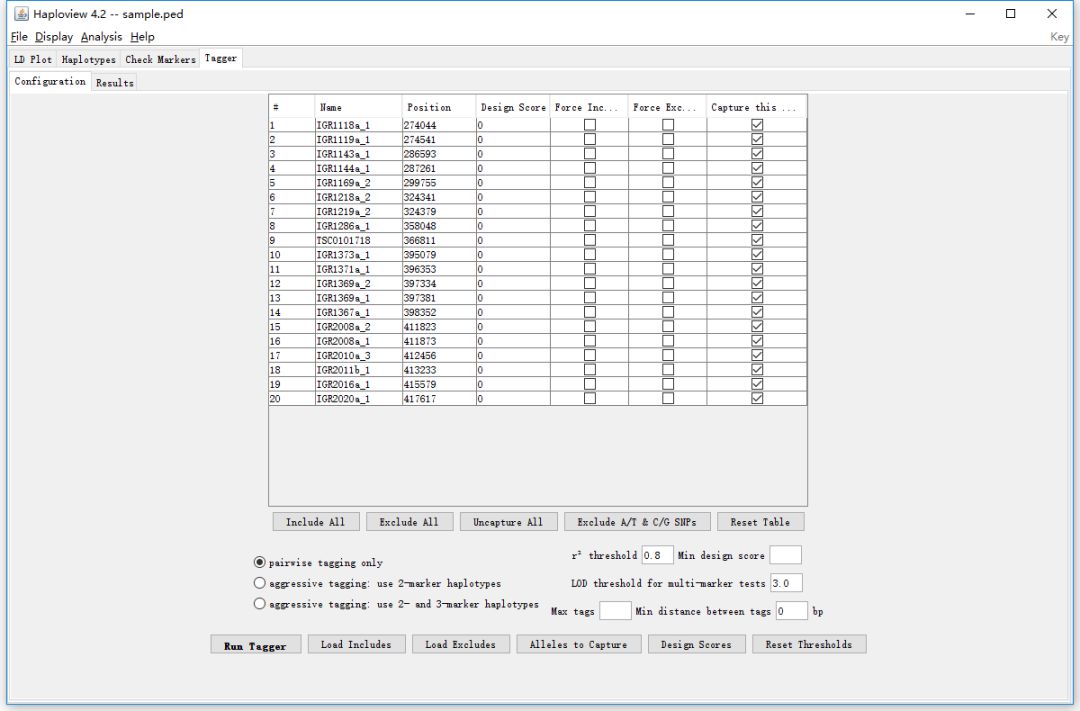
設置好之后,點擊Run Ragger按鈕,就可以了,結果界面如下
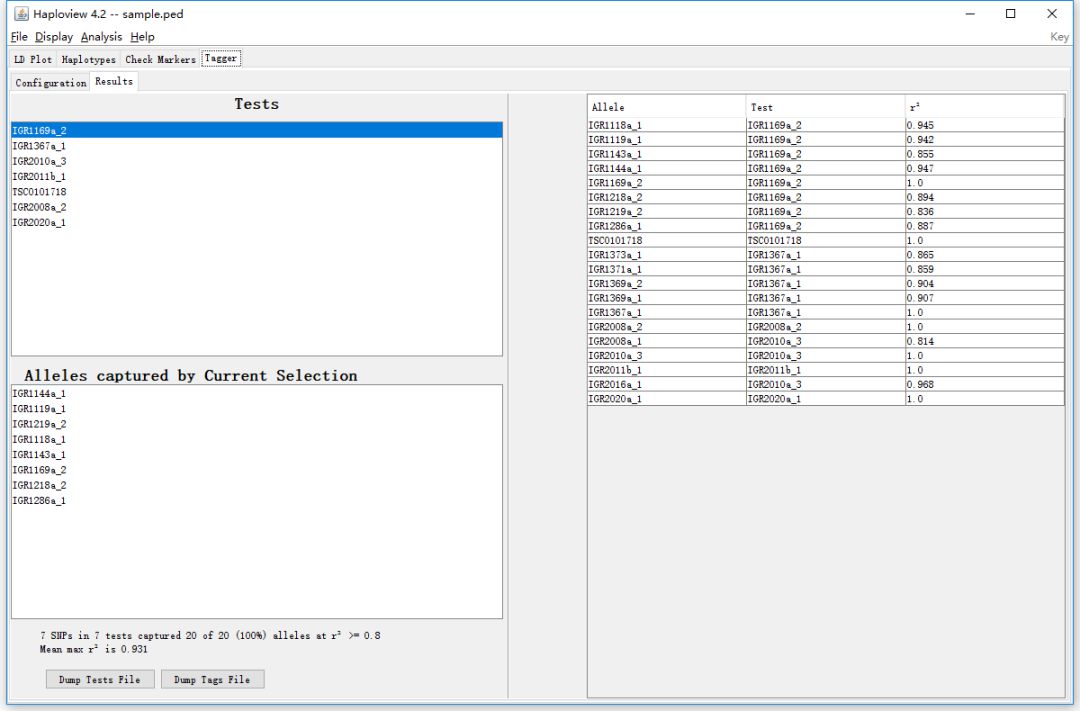
Test 框中顯示的就是挑選出的tagSNPs, 鼠標左鍵單擊每個SNP位點,在下方的Alleles captued by Current Selection 框中,會顯示該tagSNP代表的其他SNP位點。右側的表格展示了每個SNP位點對應的最佳的tagSNP。通過下方的Dump Tests File和Dump Tags File按鈕可以導出結果。
看完上述內容是否對您有幫助呢?如果還想對相關知識有進一步的了解或閱讀更多相關文章,請關注億速云行業資訊頻道,感謝您對億速云的支持。
免責聲明:本站發布的內容(圖片、視頻和文字)以原創、轉載和分享為主,文章觀點不代表本網站立場,如果涉及侵權請聯系站長郵箱:is@yisu.com進行舉報,并提供相關證據,一經查實,將立刻刪除涉嫌侵權內容。